Систематическая эволюция лигандов экспоненциальным обогащением

Системати́ческая эволю́ция лига́ндов экспоненциа́льным обогаще́нием (англ. SELEX от Systematic Evolution of Ligands by EXponential Enrichment) — метод комбинаторной химии, используемый в молекулярной биологии для получения олигонуклеотидов (коротких одноцепочечных молекул РНК или ДНК), которые специфически связываются с определённым лигандом (или лигандами). Такие олигонуклеотиды называют аптамерами[1][2][3]. Чаще всего этот метод называют SELEX, но встречаются также сокращения SAAB (от англ. selected and amplified binding site — отобранный и амплифицированный сайт связывания) и CASTing (от англ. cyclic amplification and selection of targets — циклическая амплификация и отбор мишеней)[4][5]. SELEX был разработан в 1990 году. В 2015 году журнал Journal of Molecular Evolution посвятил этому методу специальный выпуск в связи с 25-летием со времени его изобретения[6].
Процесс начинается с синтеза очень большой библиотеки олигонуклеотидов, состоящей из случайных последовательностей фиксированной длины с константными 5'- и 3'-участками, которые служат местами отжига праймеров (то есть комплементарного связывания праймеров с матрицей). Далее олигонуклеотиды, входящие в состав библиотеки, взаимодействуют с интересующим исследователя лигандом (чаще всего белком или небольшим органическим соединением). Те олигонуклеотиды, которые не смогли связаться с лигандом, удаляются при помощи аффинной хроматографии или магнитных шариков[7]. Олигонуклеотиды, связавшиеся с лигандом, элюируют, амплифицируют с помощью полимеразной цепной реакции (ПЦР) и подвергаются последующим циклам отбора, чтобы отобрать молекулы с наибольшим сродством к лиганду[2][3].
Метод SELEX уже был успешно использован для разработки ряда аптамеров для клинических и исследовательских целей[8]. С помощью SELEX также были получены аптамеры с нуклеотидами, включающими химически модифицированные сахара или азотистые основания. Модифицированные нуклеотиды могут давать аптамерам дополнительные возможности связывания, а также повышать их стабильность[9].
Метод[править | править код]
Создание библиотеки[править | править код]
Первый этап SELEX — это создание большой библиотеки олигонуклеотидов с фиксированной длиной, но полностью или частично случайными последовательностями. Однако области с вариабельной последовательностью должны быть фланкированы участками с жёстко фиксированной последовательностью, чтобы была возможна их амплификация с помощью ПЦР. Олигонуклеотиды изначально получают путём химического синтеза, а затем амплифицируют с помощью ПЦР так, чтобы каждая случайная последовательность присутствовала в библиотеке во множестве копий. В случае РНК-аптамеров проводят in vitro транскрипцию случайных последовательностей. Высокая копийность каждой случайной последовательности снижает вероятность того, что потенциальный сайт связывания будет утерян в силу стохастических причин. Чтобы олигонуклеотиды смогли приобрести нативную пространственную структуру, их переводят в одноцепочечный вид и лишь после этого инкубируют с лигандом-мишенью[2][3][10].
Инкубация с лигандом[править | править код]
Непосредственно перед добавлением олигонуклеотидов к лиганду библиотеку одноцепочечных олигонуклеотидов нагревают и медленно охлаждают, чтобы они приобрели термодинамически устойчивую вторичную и третичную структуру[3][7]. Далее библиотеку случайных последовательностей (рандомизированную библиотеку) инкубируют вместе с иммобилизированным лигандом-мишенью, чтобы дать возможность аптамерам связаться с ним. Протоколы инкубации библиотеки с лигандом могут различаться, в частности, могут использоваться различные походы для иммобилизации лигандов, отделения несвязавшихся олигонуклеотидов, различные время и температура инкубации, буферы, в которых происходит инкубация, а также соотношения концентраций олигонуклеотидов и лигандов. Лиганды иммобилизуют на колонках для аффинной хроматографии[3], нитроцеллюлозных фильтрах[2] или магнитных шариках[7]. Была также разработана разновидность SELEX, в которой в качестве лиганда выступает целая клетка. В этом случае инкубация библиотеки олигонуклеотидов происходит непосредственно в культуральных чашках, в которых растут клетки[11]. Состав буфера, в котором происходит инкубация, зависит от свойств лиганда и требований к аптамерам, успешно прошедшим отбор. Например, если лигандом являются отрицательно заряженные малые молекулы или белки, используются буферы с высокой концентрацией солей, способствующие связыванию аптамера с лигандом. Если же требуется получить аптамер, связывающийся с белком в условиях in vivo или с целой клеткой, который планируется использовать в диагностических или терапевтических целях, то для инкубации используют буферы, по концентрациям солей близкие к плазме крови, а сам процесс проводят при физиологических температурах, чтобы отобранные аптамеры наиболее вероятно связывались с лигандом именно in vivo. Буферы для инкубации также могут содержать неспецифичные вещества-компетиторы. Если есть высокая вероятность того, что олигонуклеотиды, не связывающиеся с лигандом, будут, тем не менее, оставаться в реакционной смеси за счёт неспецифичного связывания с матрицей, то неспецифичные компетиторы (малые молекулы или полимеры, по свойствам близкие к олигонуклеотидам библиотеки) будут препятствовать неспецифическому удержанию неподходящих аптамеров в реакционной смеси[11]. Свойства отобранных аптамеров могут также зависеть от соотношения концентраций лиганда и олигонуклеотидов. Если перед исследователем не стоит задачи получить аптамеры с очень сильным сродством к лиганду, то стоит использовать избыток лиганда, чтобы увеличить вероятность того, что хотя бы какие-то олигонуклеотиды смогут с ним связаться. Однако, если, напротив, необходим высокоспецифичный аптамер, сильно связывающий лиганд, то в избытке нужно брать олигонуклеотиды. Так будут созданы конкуренция между аптамерами и условия для отбора наиболее подходящих из них[2].
Элюция и амплификация[править | править код]
После того, как библиотека олигонуклеотидов была проинкубирована вместе с лигандом достаточное время, несвязавшиеся олигонуклеотиды вымывают, а связавшиеся при этом остаются связанными с иммобилизованным лигандом[3]. Когда несвязавшиеся последовательности удалены, необходимо элюировать связавшиеся аптамеры, разрушив его связь с иммобилизованным лигандом. Для элюции создают денатурирующие условия, при которых аптамеры теряют свою пространственную структуру, лишаются связей с лигандом и вымываются деионизированной водой[3]. Денатурирующие условия создаются за счёт растворов, содержащих мочевину или ЭДТА[11][12], а также под действием высокой температуры или физического воздействия. После элюции высвободившиеся олигонуклеотиды подвергаются обратной транскрипции, если они являются РНК или необходимо введение модифицированных нуклеотидов[2][3][11], а ДНК-нуклеотиды просто собираются для дальнейшей амплификации[13]. Полученные фрагменты ДНК далее амплифицируются с помощью ПЦР и переводятся в одноцепочечную ДНК (оцДНК), РНК или олигонуклеотид с модифицированными основаниями, которые будут использованы для следующего цикла отбора[2][3].
Получение оцДНК[править | править код]
Следующий этап SELEX — получение оцДНК, которая будет использована для дальнейшей амплификации с помощью ПЦР. Один из наиболее простых способов получения оцДНК — использование для ПЦР биотинилированных реверсных праймеров, благодаря чему синтезированные комплементарные цепи ДНК будут нести биотин. С помощью биотина их можно закрепить к смоле, а вторую цепь дуплекса элюировать раствором щёлочи. Другой метод для получения оцДНК — асимметричная ПЦР, при которой прямой праймер берётся в избытке, а обратный праймер — в очень небольшом количестве, благодаря чему синтезируется больше одноцепочечных фрагментов. Недостаток асимметричной ПЦР заключается в том, что после ПЦР одноцепочечный продукт необходимо очистить от двуцепочечной ДНК и других лишних фрагментов. Ненужные цепи можно разрушить ферментативно, предварительно пометив их так, чтобы её распознавала, к примеру, лямбда-экзонуклеаза. Ферменты разрушат ненужную цепь, а целевая оцДНК останется нетронутой[2][3].
Отрицательный отбор[править | править код]
Для повышения специфичности аптамеров, отобранных с помощью SELEX, непосредственно до инкубации или сразу после неё можно ввести дополнительный этап — отрицательный отбор. Этот этап нужен для удаления из смеси последовательностей, которые связываются не с лигандом, а с матрицей, на которой он иммобилизован[12][14][13]. Отрицательный отбор заключается в добавлении в смесь аналогов лиганда, неспецифичных белков и других молекул, которые собирают на себя все неспецифичные олигонуклеотиды[11][13][15].
Наблюдение за процессом селекции[править | править код]
Чтобы следить за ходом SELEX, можно сравнивать количество молекул лиганда, провзаимодействовавших с аптамерами, которое связано с числом элюированных олигонуклеотидов, с общим количеством олигонуклеотидов, которые теоретически элюируются на каждом этапе. Количество элюированных олигонуклеотидов оценивают, измеряя поглощение при длине волны 260 нм, или с использованием флуоресцентно-меченных олигонуклеотидов. Когда процесс SELEX подходит к концу, доля олигонуклеотидов, связывающихся с аптамером, доходит до 100 %, и число элюированных олигонуклеотидов становится равным (или несколько меньшим) размеру использовавшейся в инкубации библиотеки[3][16].
Химически модифицированные нуклеотиды[править | править код]
В настоящее время в SELEX используются химически модифицированные нуклеотиды. Присутствие химически модифицированных нуклеотидов в олигонуклеотидах может давать дополнительные преимущества потенциальным аптамерам, например, увеличивать их стабильность и устойчивость к нуклеазам, усиливать связывание со специфическими мишенями, изменять физические свойства (например, увеличивать гидрофобность) и увеличивать количество возможных пространственных структур для данного олигонуклеотида. В SELEX уже были использованы нуклеотиды с неприродными азотистыми основаниями, и в ряде случаев благодаря их использованию удалось получить ДНК-аптамеры с высоким сродством к мишени[8][9][17].
Применение[править | править код]
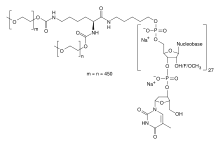
SELEX был разработан для получения путём направленной эволюции аптамеров с очень высоким сродством к определённым лигандам, в числе которых могут быть и малые молекулы наподобие АТФ[18] и аденозина[10][19], а также белки (например, прионы[20] и фактор роста эндотелия сосудов (VEGF)[21]). SELEX используют для получения аптамеров с высоким сродством и к более сложным структурам, таким как опухолевые клетки[22]. Разрабатываются аптамеры, специфично связывающие опухолевые маркеры[23], зелёный флуоресцентный белок и родственные ему флуорофоры[24]. Управление по санитарному надзору за качеством пищевых продуктов и медикаментов уже одобрило VEGF-связывающий аптамер, известный как пегаптаниб, для лечения макулодистрофии[21][25]. SELEX был использован для получения высокоспецифичной каталитической ДНК (ДНК-зимов). Известно несколько металл-специфичных ДНК-зимов[26], в числе которых ДНК-зимы, специфичные к меди[27], урану[28] и натрию[29]. Разрабатываются биосенсоры на основе аптамеров[30]; кроме того, планируется их использование для флуоресцентного мечения белков[31] и клеток[32], а также селективного ингибирования ферментов[33].
Примечания[править | править код]
- ↑ Oliphant A. R., Brandl C. J., Struhl K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. (англ.) // Molecular And Cellular Biology. — 1989. — July (vol. 9, no. 7). — P. 2944—2949. — doi:10.1128/mcb.9.7.2944. — PMID 2674675.
- ↑ 1 2 3 4 5 6 7 8 Tuerk C., Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. (англ.) // Science (New York, N.Y.). — 1990. — 3 August (vol. 249, no. 4968). — P. 505—510. — doi:10.1126/science.2200121. — PMID 2200121.
- ↑ 1 2 3 4 5 6 7 8 9 10 11 Ellington A. D., Szostak J. W. In vitro selection of RNA molecules that bind specific ligands. (англ.) // Nature. — 1990. — 30 August (vol. 346, no. 6287). — P. 818—822. — doi:10.1038/346818a0. — PMID 1697402.
- ↑ Blackwell T. K., Weintraub H. Differences and similarities in DNA-binding preferences of MyoD and E2A protein complexes revealed by binding site selection. (англ.) // Science (New York, N.Y.). — 1990. — 23 November (vol. 250, no. 4984). — P. 1104—1110. — doi:10.1126/science.2174572. — PMID 2174572.
- ↑ Wright W. E., Binder M., Funk W. Cyclic amplification and selection of targets (CASTing) for the myogenin consensus binding site. (англ.) // Molecular And Cellular Biology. — 1991. — August (vol. 11, no. 8). — P. 4104—4110. — doi:10.1128/mcb.11.8.4104. — PMID 1649388.
- ↑ Gold L. SELEX: How It Happened and Where It will Go. (англ.) // Journal Of Molecular Evolution. — 2015. — December (vol. 81, no. 5-6). — P. 140—143. — doi:10.1007/s00239-015-9705-9. — PMID 26480964.
- ↑ 1 2 3 Stoltenburg R., Schubert T., Strehlitz B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. (англ.) // PloS One. — 2015. — Vol. 10, no. 7. — P. e0134403—0134403. — doi:10.1371/journal.pone.0134403. — PMID 26221730.
- ↑ 1 2 Wu Y. X., Kwon Y. J. Aptamers: The "evolution" of SELEX. (англ.) // Methods (San Diego, Calif.). — 2016. — 15 August (vol. 106). — P. 21—28. — doi:10.1016/j.ymeth.2016.04.020. — PMID 27109056.
- ↑ 1 2 Keefe A. D., Cload S. T. SELEX with modified nucleotides. (англ.) // Current Opinion In Chemical Biology. — 2008. — August (vol. 12, no. 4). — P. 448—456. — doi:10.1016/j.cbpa.2008.06.028. — PMID 18644461.
- ↑ 1 2 Huizenga D. E., Szostak J. W. A DNA aptamer that binds adenosine and ATP. (англ.) // Biochemistry. — 1995. — 17 January (vol. 34, no. 2). — P. 656—665. — doi:10.1021/bi00002a033. — PMID 7819261.
- ↑ 1 2 3 4 5 Iwagawa T., Ohuchi S. P., Watanabe S., Nakamura Y. Selection of RNA aptamers against mouse embryonic stem cells. (англ.) // Biochimie. — 2012. — January (vol. 94, no. 1). — P. 250—257. — doi:10.1016/j.biochi.2011.10.017. — PMID 22085640.
- ↑ 1 2 Vater A., Jarosch F., Buchner K., Klussmann S. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach: tailored-SELEX. (англ.) // Nucleic Acids Research. — 2003. — 1 November (vol. 31, no. 21). — P. e130—130. — doi:10.1093/nar/gng130. — PMID 14576330.
- ↑ 1 2 3 Blank M., Weinschenk T., Priemer M., Schluesener H. Systematic evolution of a DNA aptamer binding to rat brain tumor microvessels. selective targeting of endothelial regulatory protein pigpen. (англ.) // The Journal Of Biological Chemistry. — 2001. — 11 May (vol. 276, no. 19). — P. 16464—16468. — doi:10.1074/jbc.M100347200. — PMID 11279054.
- ↑ Stoltenburg R., Reinemann C., Strehlitz B. SELEX--a (r)evolutionary method to generate high-affinity nucleic acid ligands. (англ.) // Biomolecular Engineering. — 2007. — October (vol. 24, no. 4). — P. 381—403. — doi:10.1016/j.bioeng.2007.06.001. — PMID 17627883.
- ↑ Haller A. A., Sarnow P. In vitro selection of a 7-methyl-guanosine binding RNA that inhibits translation of capped mRNA molecules. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1997. — 5 August (vol. 94, no. 16). — P. 8521—8526. — doi:10.1073/pnas.94.16.8521. — PMID 9238009.
- ↑ Sefah K., Shangguan D., Xiong X., O'Donoghue M. B., Tan W. Development of DNA aptamers using Cell-SELEX. (англ.) // Nature Protocols. — 2010. — June (vol. 5, no. 6). — P. 1169—1185. — doi:10.1038/nprot.2010.66. — PMID 20539292.
- ↑ Pinheiro V. B., Taylor A. I., Cozens C., Abramov M., Renders M., Zhang S., Chaput J. C., Wengel J., Peak-Chew S. Y., McLaughlin S. H., Herdewijn P., Holliger P. Synthetic genetic polymers capable of heredity and evolution. (англ.) // Science (New York, N.Y.). — 2012. — 20 April (vol. 336, no. 6079). — P. 341—344. — doi:10.1126/science.1217622. — PMID 22517858.
- ↑ Dieckmann T., Suzuki E., Nakamura G. K., Feigon J. Solution structure of an ATP-binding RNA aptamer reveals a novel fold. (англ.) // RNA (New York, N.Y.). — 1996. — July (vol. 2, no. 7). — P. 628—640. — PMID 8756406.
- ↑ Burke D. H., Gold L. RNA aptamers to the adenosine moiety of S-adenosyl methionine: structural inferences from variations on a theme and the reproducibility of SELEX. (англ.) // Nucleic Acids Research. — 1997. — 15 May (vol. 25, no. 10). — P. 2020—2024. — doi:10.1093/nar/25.10.2020. — PMID 9115371.
- ↑ Mercey R., Lantier I., Maurel M. C., Grosclaude J., Lantier F., Marc D. Fast, reversible interaction of prion protein with RNA aptamers containing specific sequence patterns. (англ.) // Archives Of Virology. — 2006. — November (vol. 151, no. 11). — P. 2197—2214. — doi:10.1007/s00705-006-0790-3. — PMID 16799875.
- ↑ 1 2 Ulrich H., Trujillo C. A., Nery A. A., Alves J. M., Majumder P., Resende R. R., Martins A. H. DNA and RNA aptamers: from tools for basic research towards therapeutic applications. (англ.) // Combinatorial Chemistry & High Throughput Screening. — 2006. — September (vol. 9, no. 8). — P. 619—632. — PMID 17017882.
- ↑ Daniels D. A., Chen H., Hicke B. J., Swiderek K. M., Gold L. A tenascin-C aptamer identified by tumor cell SELEX: systematic evolution of ligands by exponential enrichment. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2003. — 23 December (vol. 100, no. 26). — P. 15416—15421. — doi:10.1073/pnas.2136683100. — PMID 14676325.
- ↑ Ferreira C. S., Matthews C. S., Missailidis S. DNA aptamers that bind to MUC1 tumour marker: design and characterization of MUC1-binding single-stranded DNA aptamers. (англ.) // Tumour Biology : The Journal Of The International Society For Oncodevelopmental Biology And Medicine. — 2006. — Vol. 27, no. 6. — P. 289—301. — doi:10.1159/000096085. — PMID 17033199.
- ↑ Paige J. S., Wu K. Y., Jaffrey S. R. RNA mimics of green fluorescent protein. (англ.) // Science (New York, N.Y.). — 2011. — 29 July (vol. 333, no. 6042). — P. 642—646. — doi:10.1126/science.1207339. — PMID 21798953.
- ↑ Vavvas D., D'Amico D. J. Pegaptanib (Macugen): treating neovascular age-related macular degeneration and current role in clinical practice. (англ.) // Ophthalmology Clinics Of North America. — 2006. — September (vol. 19, no. 3). — P. 353—360. — doi:10.1016/j.ohc.2006.05.008. — PMID 16935210.
- ↑ Breaker R. R., Joyce G. F. A DNA enzyme that cleaves RNA. (англ.) // Chemistry & Biology. — 1994. — December (vol. 1, no. 4). — P. 223—229. — PMID 9383394.
- ↑ Carmi N., Shultz L. A., Breaker R. R. In vitro selection of self-cleaving DNAs. (англ.) // Chemistry & Biology. — 1996. — December (vol. 3, no. 12). — P. 1039—1046. — PMID 9000012.
- ↑ Liu J., Brown A. K., Meng X., Cropek D. M., Istok J. D., Watson D. B., Lu Y. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2007. — 13 February (vol. 104, no. 7). — P. 2056—2061. — doi:10.1073/pnas.0607875104. — PMID 17284609.
- ↑ Torabi S. F., Wu P., McGhee C. E., Chen L., Hwang K., Zheng N., Cheng J., Lu Y. In vitro selection of a sodium-specific DNAzyme and its application in intracellular sensing. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2015. — 12 May (vol. 112, no. 19). — P. 5903—5908. — doi:10.1073/pnas.1420361112. — PMID 25918425.
- ↑ Lubin A. A., Hunt B. V., White R. J., Plaxco K. W. Effects of probe length, probe geometry, and redox-tag placement on the performance of the electrochemical E-DNA sensor. (англ.) // Analytical Chemistry. — 2009. — 15 March (vol. 81, no. 6). — P. 2150—2158. — doi:10.1021/ac802317k. — PMID 19215066.
- ↑ Umrao Saurabh, Jain Vasundhara, Chakraborty Banani, Roy Rahul. Protein-induced fluorescence enhancement as aptamer sensing mechanism for thrombin detection (англ.) // Sensors and Actuators B: Chemical. — 2018. — August (vol. 267). — P. 294—301. — ISSN 0925-4005. — doi:10.1016/j.snb.2018.04.039.
- ↑ Terazono H., Anzai Y., Soloviev M., Yasuda K. Labelling of live cells using fluorescent aptamers: binding reversal with DNA nucleases. (англ.) // Journal Of Nanobiotechnology. — 2010. — 13 April (vol. 8). — P. 8—8. — doi:10.1186/1477-3155-8-8. — PMID 20388214.
- ↑ Mondragón E., Maher LJ 3rd. RNA aptamer inhibitors of a restriction endonuclease. (англ.) // Nucleic Acids Research. — 2015. — 3 September (vol. 43, no. 15). — P. 7544—7555. — doi:10.1093/nar/gkv702. — PMID 26184872.
Эта статья входит в число добротных статей русскоязычного раздела Википедии. |