Нетранслируемые области

Нетрансли́руемые о́бласти (НТО, англ. untranslated regions, UTR) — особые участки мРНК, не выступающие в качестве матрицы для синтеза белка и прилегающие с обеих сторон к транслируемой области (то есть той, на матрице которой синтезируется белок). Таких области две: 5'-нетранслируемая область, или 5'-НТО (англ. 5'-untranslated region, 5' UTR) и 3'-нетранслируемая область, или 3'-НТО (англ. 3'-untranslated region, 3' UTR), располагающиеся на 5'- и 3'-конце мРНК соответственно[1]. Такое же название имеют участки ДНК, соответствующие 5'-НТО и 3'-НТО транскрипта[2].
Нетранслируемые области вовлечены в регуляцию локализации, трансляции и деградации того транскрипта, в составе которого они находятся. Для них характерно наличие шпилек, внутренних инициаторных кодонов и открытых рамок считывания, сайтов связывания рибосомы, различных цис-регуляторных элементов, связывающихся с РНК-связывающими белками[3]. Так, в них локализованы такие элементы, как IRES, uORF, ARE[3], последовательность Шайна — Дальгарно, рибопереключатель и другие[4].
Структурные особенности
[править | править код]
Анализ геномов различных организмов показал наличие ряда консервативных свойств, присущих нетранслируемым областям. Общая длина 5'-НТО приблизительно одинакова среди всех таксономических групп эукариот и составляет от 100 до 200 нуклеотидов (впрочем, у дрожжей Schizosaccharomyces pombe длина 5'-НТО в транскрипте ste11 составляет 2273 нуклеотида[5]). В то же время длина 3'-НТО гораздо более вариабельна и может составлять от 200 нуклеотидов у растений и некоторых животных до 800 нуклеотидов у человека и других позвоночных. Поразительным является тот факт, что длина как 5'-, так и 3'-НТО значительно изменяется в пределах одного вида: она может принимать значение от 12 до нескольких тысяч нуклеотидов[6]. Действительно, в in vitro системе, моделирующей генетический аппарат млекопитающих, показано, что даже 5'-НТО длиной в 1 нуклеотид может обеспечивать нормальную инициацию трансляции[7].
Участок геномной ДНК, соответствующий нетранслируемым областям мРНК, может содержать интроны, причём чаще в области 5', чем в 3'. Около 30 % генов Metazoa имеют участки, соответствующие 5'-НТО, состоящие только из экзонов, в то же время в 3'-НТО, хотя она и длиннее, интронов гораздо меньше. Общая доля длины интронов от всей длины в 3'-НТО составляет 1—11 %. Образование альтернативных нетранслируемых участков имеет место при использовании различных сайтов начала транскрипции, сайтов полиаденилирования и сплайсинга. В зависимости от ткани, стадии развития, наличия болезненного состояния количество альтернативных нетранслируемых областей может изменяться, и они могут значительно влиять на экспрессию тех или иных генов[8].
Состав оснований также различается в 3'- и 5'-НТО. Так, например, в 5'-НТО выше содержание G+C, чем в 3'-НТО. Особенно это различие заметно в мРНК теплокровных позвоночных, у которых содержание G+C в 5'-НТО составляет 60 %, а в 3'-НТО — 45 %[6]. Существует также определённая связь между содержанием G+C в 5'-НТО и 3'-НТО и третьими позициями в кодонах соответствующей транслируемой области. Также обнаружена важная обратная связь между содержанием G+C в 5'-НТО и 3'-НТО и их длинами[9]. В частности, известно, что гены, локализованные в GC-богатых участках хромосом (тяжёлые изохоры), имеют более короткие 5'-НТО и 3'-НТО, чем гены, расположенные в изохорах, более бедных GC. Похожая связь была показана для кодирующих последовательностей и интронов[10].
Наконец, в нетранслируемых областях мРНК эукариот было установлено наличие повторяющихся последовательностей различных типов, например, SINEs (в том числе Alu-повторов) и LINEs, минисателлитов и микросателлитов. У человека повторы мРНК составляют 12 % 5'-НТО и 36 % 3'-НТО, в других таксонах, в том числе у других млекопитающих, было показано меньшее содержание повторов[3].
Функции
[править | править код]Нетранслируемые области выполняют ключевые функции в посттранскрипционной регуляции экспрессии генов, включая модуляцию транспорта мРНК из ядра, регуляцию внутриклеточной локализации мРНК[11], её стабильности[12] и эффективность трансляции[13]. Нетранслируемые области могут играть роль и в других процессах, например, в котрансляционном включении нестандартной аминокислоты селеноцистеина по кодону UGA (обычно это стоп-кодон) мРНК, кодирующих селенопротеины (в этом процессе участвует консервативная шпилька, расположенная в 3'-НТО — SECIS-элемент)[14]. Важность нетранслируемых областей в регуляции экспрессии генов подтверждается ещё и тем, что мутации, затрагивающие эти области, могут приводить к серьёзным патологиям[15] (подробнее о заболеваниях, обусловленных мутациями в НТО, см. ниже).
Регуляция, осуществляемая нетранслируемыми участками мРНК, может быть опосредована несколькими путями. Нуклеотидные мотивы, расположенные в 3'-НТО и 5'-НТО, могут взаимодействовать со специфическими РНК-связывающими белками. В отличие от регуляторных элементов, локализованных в ДНК, у которых ведущую роль играет первичная структура ДНК (то есть последовательность нуклеотидов), биологическая активность регуляторных мотивов, расположенных на РНК, определяется и их первичной, и вторичной структурой. Также ключевые роли в регуляции были показаны для взаимодействий между участками нетранслируемых областей и специфическими комплементарными некодирующими РНК, в частности, микроРНК[16]. Наконец, известны примеры повторяющихся элементов, важных для регуляции экспрессии генов на уровне РНК, например, CUG-связывающие белки могут взаимодействовать с CUG-повторами в 5'-НТО специфической мРНК (например, кодирующей транскрипционный фактор C/EBPβ) и тем самым влиять на эффективность трансляции[17].
Контроль эффективности трансляции
[править | править код]Эффективность трансляции мРНК может быть различной, поэтому возможна регуляция количества получающегося белка. Это является важным механизмом регуляции экспрессии гена. Действительно, лишь для секретируемых белков существует чёткая связь между количеством мРНК и белка (чем больше мРНК, тем больше белка). У белков, предназначенных для внутриклеточного использования, эта взаимосвязь в значительной мере искажается различными темпами трансляции различных мРНК[18].
Структурные особенности 5'-НТО имеют важное значение для контроля трансляции. Показано, что мРНК, кодирующие белки, участвующие в процессах развития, например, факторы роста, транскрипционные факторы или продукты протоонкогенов (белки, для которых необходим чёткий контроль экспрессии), имеют 5'-НТО длиннее среднего[19], содержащие внутри инициаторные кодоны (англ. upstream initiation codons) и открытые рамки считывания, а также стабильные элементы вторичной структуры, препятствующие процессу трансляции (например, квадруплексы). Другие специфические мотивы и элементы вторичной структуры 5'-НТО могут модулировать эффективность трансляции[3].
В норме после перемещения мРНК из ядра в цитоплазму в области кэпа, расположенном на 5'-конце мРНК, собирается белковый комплекс eIF4F. В этот комплекс входят 3 субъединицы: eIF4Е (кэп-связывающий белок); eIF4А, обладающий хеликазной активностью; eIF4G, взаимодействующий с различными другими белками, в том числе с полиаденилат-связывающим белком. АТФ-зависимая хеликазная активность eIF4А, стимулируемая РНК-связывающим белком eIF4B, обеспечивает расплетание любых элементов вторичной структуры мРНК, в результате чего образуется «площадка для посадки» малой (40S) субъединицы рибосомы[20]. Если трансляцию сдерживает количество рибосом или концентрация факторов трансляции, 3'-поли(А)-хвост может взаимодействовать с 5'-кэпом, усиливая инициацию трансляции путём внедрения полиаденилат-связывающего белка, который может взаимодействовать с комплексом eIF4F[21].
Считается, что в эукариотических мРНК трансляция начинается с первого кодона AUG (стартовый кодон), который встретится рибосоме на её пути от 5'- к 3'-концу. Последовательности, окружающие стартовые кодоны, неслучайны и составляют консенсусную последовательность Козак. У млекопитающих эта последовательность имеет следующий вид: GCCRCCaugG, и наиболее консервативными нуклеотидами являются R (пурин, обычно А) на позиции −3 относительно AUG и G в позиции +4 относительно AUG. Строгое расположение А в позиции −3 и G в позиции +4 также характерно для других животных, растений и грибов. Последовательности, составляющие окружение AUG (частично они заходят в нетранслируемую область), могут модулировать эффективность трансляции, обеспечивая подходящее окружение для инициации[3].
Следует отметить, что от 15 % до 50 % 5'-НТО содержат внутри стартовый кодон AUG. Поэтому правило, согласно которому рибосома начинает трансляцию с первого стартового кодона, который встретится ей при перемещении от 5'- к 3'-концу мРНК, выполняется далеко не всегда. Это означает, что иногда рибосома может пропускать встречающиеся ей триплеты AUG и начинать трансляцию с более удалённого стартового кодона, возможно из-за того, что эти триплеты имеют «слабое» нуклеотидное окружение. Таким образом, с одной мРНК может быть синтезировано несколько различных белков[22]. Более того, установлено, что наличие внутренних кодонов AUG в 5'-НТО коррелирует с увеличенной длиной этой области и более «слабым» окружением обычно используемого стартового кодона, а в транскриптах с оптимальным окружением стартового AUG 5'-нетранслируемая область короткая и не содержит AUG[23]. В связи с этим AUG в 5'-НТО могут уменьшать уровень трансляции своей мРНК.
Если в 5'-НТО после внутреннего AUG, но перед основным стартовым кодоном располагается внутренний стоп-кодон, то образуется короткая открытая рамка считывания (англ. upstream open reading frame, uORF). После трансляции uORF и диссоциации от мРНК большой (60S) субъединицы рибосомы, судьба малой субъединицы может быть различной, и это может оказать влияние на эффективность трансляции и стабильность мРНК. Малая субъединица может остаться на мРНК, возобновить считывание и начать трансляцию с нижележащего кодона AUG, а может покинуть мРНК и тем самым понизить уровень трансляции основной открытой рамки считывания. У эукариот способность рибосомы к возобновлению трансляции ограничивается, во-первых, стоп-кодонами[24] и, во-вторых, длиной uORF: если длина uORF превышает 30 кодонов[25], рибосома не сможет возобновить трансляцию. Таким способом блокируется трансляция мРНК, содержащих uORF, кодирующих транскрипционные факторы дрожжей GCN4 и YAP1[26].
Важное значение в регуляции трансляции имеет вторичная структура 5'-НТО. Экспериментальные данные показывают, что умеренно стабильные элементы вторичной структуры (значение изменения свободной энергии ΔG выше −30 ккал/моль), непосредственно содержащие стартовый кодон AUG, не останавливают малую субъединицу рибосомы. Существенное влияние на эффективность трансляции оказывают очень стабильные структуры (ΔG ниже −50 ккал/моль), снижая её. Преодолению влияния таких структур способствует повышение концентрации eIF4A[27].
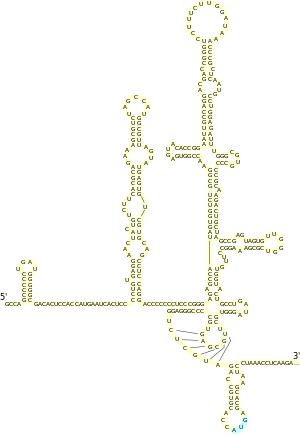
Существует альтернативный механизм инициации трансляции, не связанный с 5'-кэпом. Впервые он был описан у пикорнавирусов[28]. В этом случае внутри 5'-НТО существует особая последовательность, служащая для связывания рибосомы — IRES. В дальнейшем IRES были обнаружены во многих клеточных мРНК, кодирующих регуляторные белки, например, продукты протоонкогена c-Myc, гомеодоменные белки, факторы роста (например, фактор роста фибробластов FGF2[англ.]), а также рецепторы к ним[3]. Сравнительный анализ известных клеточных IRES позволил выделить общий структурный мотив, характерный для содержащих их мРНК. В частности, для мРНК белка, связывающегося с тяжёлой цепью иммуноглобулина (BiP), и мРНК белка FGF-2 была описана Y-образная шпилька, расположенная непосредственно перед стартовым кодоном AUG[29]. Установлено, что короткие структурные мотивы[англ.], комплементарные малым рибосомным РНК, могут также выступать как IRES[30].
Последовательности, являющиеся мишенями транс-функционирующих РНК-связывающих белков, могут также принимать участие в регуляции трансляции. Например, железозависимый элемент IRE (англ. iron-responsive element), локализованный в 5'-НТО мРНК, кодирующих белки, вовлечённые в метаболизм железа (ферритин, 5-аминолевулинатсинтаза[англ.] и аконитаза), может блокировать трансляцию. При этом происходит железозависимое связывание белков метаболизма железа, подавляющее нормальное сканирование мРНК, осуществляемое малой субъединицей рибосомы при инициации трансляции. Наконец, в большей части мРНК позвоночных, кодирующих рибосомные белки и факторы элонгации трансляции, содержится 5'-терминальный пиримидиновый тракт (англ. terminal oligopyrimidine tract (TOP)), непосредственно прилегающий к кэпу. Этот тракт необходим для координированной репрессии трансляции в ходе задержки роста, дифференциации, развития, а также активируется при употреблении некоторых препаратов[31].
Регуляция стабильности мРНК
[править | править код]Судьба мРНК является другой ключевой точкой в посттранскрипционной регуляции экспрессии генов, так как в отсутствие разрушения определённых мРНК их число будет расти, а значит, увеличится количество кодируемого ими белка, что может повлиять на экспрессию тех или иных генов. Было предложено несколько возможных механизмов разрушения мРНК: её деградация может запускаться укорочением или отрывом 3'-поли(А)-хвоста или 5'-кэпа[32]. Судьба мРНК в основном регулируется цис-регуляторными элементами, расположенными в 3'-НТО, например, AU-богатыми элементами (AREs), которые запускают распад мРНК под влиянием различных внутри- и внеклеточных сигналов. По имеющимся экспериментальным данным AREs разделили на 3 класса: для членов I и II класса характерно наличие множества копий пентануклеотида AUUUA, чего нет у представителей III класса[33]. AREs I класса контролируют цитоплазматическое деаденилирование путём деградации всего поли(А)-хвоста с одинаковой скоростью у всех транскриптов с образованием сначала промежуточных соединений с поли(А)-хвостом в 30—60 нуклеотидов, которые потом полностью деградируют. Такие элементы обнаруживаются в основном в мРНК, кодирующих ядерные факторы транскрипции, например, c-Fos и c-Myc (продукты генов «быстрого ответа»), а также мРНК, кодирующих некоторые цитокины, например, интерлейкины 4 и 6. Структурной характеристикой AREs I класса является наличие одной или нескольких копий пентануклеотида AUUUA после U-богатого участка. AREs II класса управляют асинхронным цитоплазматическим деаденилированием (то есть поли(А)-хвост различных транскриптов разрушается с различной скоростью), в результате которого образуется мРНК без поли(А)-хвоста. В число мРНК, содержащих такие элементы, входят мРНК цитокинов GM-CSF, интерлейкин 2, фактор некроза опухоли α (TNF-α), интерферон-α. Структурной особенностью AREs второго класса является наличие тандемных повторов пентануклеотида AUUUA, а AU-богатый участок располагается до этих повторов. У AREs III класса отсутствует пентануклеотид AUUUA и есть только U-богатый участок. Такой элемент есть, например, у мРНК, кодирующей c-Jun. Кинетика деградации мРНК в этом случае схожа с таковой у AREs I[3].
Деградация мРНК может также осуществляться за счёт эндонуклеазной активности, причём такой механизм зависим и от деаденилирования, и декэпирования. Этот механизм был обнаружен у мРНК, кодирующих трансферриновый рецептор[англ.]. Деградация этих мРНК включает эндонуклеазное разрезание 3'-НТО, причём промежуточным этапом является распознавание IRE, а регуляция определяется уровнем внутриклеточного железа[34].

Инициаторные кодоны в 5'-НТО и uORF также могут играть роль в особом механизме распада бессмысленных (англ. nonsense) («бракованных») мРНК — NMD (англ. nonsense-mediated mRNA decay). Сигналом, запускающим этот путь, является бессмысленный кодон, вслед за которым идёт соединение между двумя экзонами, образовавшееся при сплайсинге — комплекс сращивания экзонов, или EJC (англ. Exon junction complex)[35] (наличие такого соединения отличает преждевременный терминаторный кодон от основного, так как стоп-кодон и 3'-НТО располагаются после последнего экзона). Соединения экзонов распознаются благодаря маркерным белкам, присоединяющимся к непроцессированному транскрипту ещё в ядре и остающимся связанными с ним после процессинга и перехода мРНК в цитоплазму[36]. В случае мРНК дикого типа (то есть «небракованных») аппарат трансляции убирает маркерный белок для предотвращения деградации транскрипта. Если же рибосома встречает преждевременный стоп-кодон или если в транскрипте присутствуют uORF, она распадается, и меченная маркерными белками дефектная мРНК вовлекается в NMD[37]. У дрожжей Saccharomyces cerevisiae (у них имеется второй сигнал, запускающий NMD — нижестоящие экзонные элементы (англ. downstream exonic element (DSE)) мРНК, содержащие функционально активные uORF, например, кодирующие GCN4 и YAP1, не деградируют по пути NMD. Дело в том, что между uORF и кодирующей последовательностью располагаются мРНК-специфические стабилизирующие последовательности, предотвращающие активацию NMD за счёт взаимодействия с РНК-связывающей убиквитин-лигазой Pub1[38].
uORF могут регулировать стабильность мРНК и независимо от NMD. В 5'-НТО мРНК гена YAP2 S. cerevisiae содержится 2 uORF, которые подавляют сканирование транскрипта рибосомой и способствуют распаду мРНК[26]. Дестабилизирующий эффект зависит от окружения терминаторного кодона, которое регулирует эффективность трансляции и стабильность мРНК.
Несколько исследований дают основание предположить, что многие гетерогенные ядерные рибонуклеопротеины[англ.] (англ. heterogeneous nuclear ribonucleoproteins (hnRNPs)) функционируют не только в ядре, но также контролируют судьбу мРНК в цитоплазме[39], регулируют трансляцию, стабильность мРНК и её локализацию в цитоплазме[37]. Примером может служить белок-предшественник амилоидов (англ. amyloid precursor protein (APP)). Повышение содержания APP является важным фактором развития болезни Альцгеймера. Стабильность мРНК APP зависит от высококонсервативного 29-нуклеотидного элемента, расположенного в 3'-НТО и взаимодействующего с различными РНК-связывающими цитоплазматическими белками[40].
Контроль внутриклеточной локализации мРНК
[править | править код]Контроль экспрессии генов на посттранскрипционном уровне, осуществляемый нетранслируемыми областями, особенно важен в период развития. Асимметричное расположение в клетке некоторых мРНК приводит к асимметричному расположению кодируемых ими белков. Это — наиболее удобный механизм локализации белков, так как одна мРНК может служить матрицей для нескольких кругов трансляции. Во многих случаях мРНК располагаются в рибонуклеопротеиновых комплексах вместе с белками трансляционного аппарата, тем самым гарантируя необходимую эффективность трансляции[3].
Существуют 3 основных механизма асимметричного расположения мРНК:
- активный направленный транспорт, в котором задействован функциональный цитоскелет и моторные белки, взаимодействующие с мРНК;
- локальная стабилизация транскриптов;
- диффузия мРНК, определяемая её локальным окружением[3].
мРНК основного белка миелина (англ. Myelin basic protein (MBP)) доставляется в отростки олигодендроцитов, образующих миелиновую обкладку аксонов ЦНС, через активный направленный транспорт. У мыши для транспорта и локализации мРНК служат особые сигнальные последовательности, располагающиеся в 3'-НТО: РНК-транспортный сигнал (длиной 21 нуклеотид), а также дополнительный элемент — регион РНК-локализации[41].
Многие примеры локальной стабилизации транскриптов обнаружены в ранних стадиях развития дрозофилы. Так, транскрипты, кодирующие РНК-связывающий белок Nanos и белок теплового шока Hsp83 деградируются везде, кроме цитоплазмы на заднем полюсе эмбриона. Различные цис-регуляторные элементы, располагающиеся в 3'-НТО соответствующих мРНК, отвечают как за деградацию этих мРНК по всему зародыщу, так и за их стабилизацию на заднем конце зародыша[42].
Явление диффузии мРНК, определяемой окружением, хорошо демонстрирует мРНК белка Bicoid у дрозофилы. Элементы, осуществляющие ключевой этап в этом процессе — заякоривание транскрипта, не полностью описаны, но один из участвующих в этом белок — Staufen[англ.] — является дцРНК-связывающим белком, необходимым для остановки Bicoid на переднем конце зародыша[43].
Во всех приведённых выше примерах локализация регулировалась цис-регуляторными элементами, расположенными в 3'-НТО, но известны и такие элементы, расположенные в 5'-НТО и даже кодирующей области. Такие элементы известны как мРНК-архивирующие коды (англ. mRNA zip codes), они взаимодействуют с соответствующими связывающими белками (англ. zip-code-binding proteins), например, уже упоминавшимся Staufen. У архивирующих кодов отсутствует какое-либо сходство в первичной и вторичной структуре. Они могут иметь сложную (комплексную) вторичную и третичную структуру, в которой первичная структура (последовательность нуклеотидов) не так важна, как пространственная структура[44], а могут, напротив, быть короткими нуклеотидными последовательностями[45], иногда входящими в повторяющиеся элементы (например, в случае транскрипта Vg1[англ.] у лягушки Xenopus[46]).
Ремоделирование НТО
[править | править код]Альтернативный сплайсинг является важнейшим путём образования из одного исходного транскрипта различных мРНК, кодирующих одинаковые или различные белки. При этом, помимо длинных белоккодирующих мРНК, могут образовываться и некодирующие РНК. Разрезанная мРНК может подвергнуться процессу рекэпирования с образованием мРНК с укороченной 5'-нетранслируемой областью или же кодирующая лишь фрагмент исходного белка. Кроме того, известно, что образовавшиеся в ходе альтернативного сплайсинга 3'-НТО-фрагменты могут начать функционировать как транс-регуляторные некодирующие РНК независимо от основной РНК[47].
Взаимодействие 5'-НТО и 3'-НТО
[править | править код]
Известно, что мРНК способна замыкаться в петлю (циркуляризация) за счёт взаимодействия специфических белков, связывающихся с поли(А)-хвостом, способствующих связыванию фактора eIF4F с кэпом. В результате мРНК приобретает замкнутую форму, стимулируется инициация трансляции и повышается эффективность трансляции. Однако в некоторых случаях 5'-НТО и 3'-НТО одной и той же мРНК могут связываться друг с другом комплементарно. Так, у мРНК человеческого гена, кодирующего транскрипционный фактор p53, имеются участки в 5'-НТО и 3'-НТО, комплементарные друг другу. Связываясь друг с другом и с трансляционным фактором RPL26[англ.], они тем самым способствуют повышению эффективности трансляции, что является одной из причин быстрого накопления белка р53 в ответ на повреждения ДНК[48].
Анализ мРНК различных генов человека показал, что в 5'-НТО присутствует мотив, специфически взаимодействующий с 3'-концами микроРНК, при этом во многих таких мРНК на 5'-конце имеется сайт, комплементарный 3'-НТО. Дальнейшие исследования показали, что связывание 5'-НТО с микроРНК облегчает связь 5'-конца мРНК с 3'-концом, и мРНК, активность которых значительно определяется микроРНК, имеют предсказуемые сайты связывания на обеих НТО. Такие мРНК получили название miBridge. Далее было установлено, что утрата этих сайтов связывания уменьшала репрессию трансляции транскрипта, управляемую микроРНК. Так было выяснено, что сайты связывания НТО друг с другом необходимы для подавления трансляции мРНК. Это свидетельствует о том, что комплементарное взаимодействие 5'-НТО и 3'-НТО необходимо для точной регуляции экспрессии генов[49].
НТО прокариот и вирусов
[править | править код]Бактерии
[править | править код]
В мРНК бактерий также имеются 5'- и 3'-нетранслируемые области[51][52]. Длина 5'-НТО бактерий значительно меньше таковой у эукариот и составляет обычно 3—10 нуклеотидов. Например, длина 5'-НТО транскрипта лактозного оперона Escherichia coli составляет лишь 7 нуклеотидов[53]. В 5'-НТО бактерий локализована последовательность Шайна — Дальгарно (AGGAGG)[54], служащая для связывания рибосомы и отделённая спейсером от стартового кодона AUG. Хотя 5'-НТО бактерий и эукариот различны, было показано, что добавление нуклеотидов СС в спейсер мРНК гена Ner бактериофага Mu, хорошо экспрессирующегося в клетках Escherichia coli и Streptomyces, привело к успешной экспрессии этого гена в клетках ретикулоцитов кролика[55].
Элементы вторичной структуры, локализованные в 5'-НТО, как правило, оказывают подавляющее действие на трансляцию[56]. В частности, именно в 5'-НТО обычно располагаются аттенюаторы[англ.] — элементы оперонов, вызывающие преждевременную терминацию трансляции[57] (наиболее известным примером аттенюации является экспрессия триптофанового оперона).
Кроме того, в 5'-НТО бактерий располагается большая часть рибопереключателей[58] — регуляторных элементов мРНК, способных связываться с малыми молекулами, что приводит к изменению эффективности образования белка, кодируемого этой мРНК[59].
В отличие от эукариот, длинные 3'-НТО редки у бактерий и слабо изучены. Тем не менее, известно, что у некоторых бактерий, в частности, Salmonella enterica, имеются мРНК с длинными 3'-НТО, похожими на эукариотические (у S. enterica это мРНК hilD). Предполагается, что 3'-НТО hilD выполняют различные функции, в частности, влияют на обращение своих мРНК, поскольку делеция этих областей вызывала увеличение количества соответствующих мРНК[60].
Археи
[править | править код]Нетранслируемые области имеются и в мРНК многих архей. В частности, в 5'- и 3'-НТО мРНК метаногенной археи Methanocaldococcus jannaschii[англ.] (как и у других представителей порядков Methanopyrales и Methanococcales) локализован элемент SECIS, ответственный за вставку аминокислоты селеноцистеина в полипептидную цепь[61].
Установлено, что мРНК большинства галоархей, а также Pyrobaculum[англ.] и Sulfolobus[англ.] лишены выраженной 5'-НТО, но у мРНК архей-метаногенов имеются длинные 5'-НТО. В связи с этим предполагается, что механизм инициации трансляции метаногенных архей может быть отличен от такового у остальных представителей этого домена[56]. Тем не менее, в мРНК галоархей имеются 3'-НТО и их 3'-концы не подвергаются посттранскрипционной модификации. Удивительным является тот факт, что в тех транскриптах галоархей, у которых есть 5'-НТО, отсутствует последовательность Шайна — Дальгарно. Длина же 3'-НТО галоархей составляла от 20 до 80 нуклеотидов; никаких консервативных структурных мотивов и последовательностей, кроме пента-U-нуклеотида в области терминации трансляции, выявлено не было[62].
Для мРНК архей описаны рибопререключатели, в том числе TPP-рибопереключатели[англ.] (связываются с тиаминпирофосфатом (TPP)), которые располагаются в 5'-НТО (подобные рибопереключатели имеются также у бактерий и эукариот)[63].
Вирусы
[править | править код]
У многих вирусов инициация трансляции происходит по кэп-независимому механизму и осуществляется через уже упоминавшиеся элементы IRES, локализованные в 5'-НТО[64]. Например, так происходит у ВИЧ, вирусов гепатита A и С[65]. Такой механизм инициации трансляции удобен тем, что в его случае нет необходимости в сборке пре-инициаторного белкового комплекса, и вирус может быстро размножаться[53].
У вирусов существует и другой кэп-независимый механизм инициации трансляции, не связанный с IRES. Такой механизм имеется у многих вирусов растений. В этом случае имеется особый кэп-независимый трансляционный элемент (англ. cap-independent translation element (CITE)), расположенный в 3'-НТО. Нередко CITE связывает факторы трансляции, например, комплекс eIF4F, и затем комплементарно взаимодействует с 5'-концом, доставляя факторы инициации трансляции к месту её начала[66].
У вирусов, геном которых представлен одноцепочечной молекулой РНК положительной полярности[англ.], 3'-НТО не только оказывает влияние на трансляцию, но также задействована в репликации: именно с неё начинается репликация вирусного генома[67].
Вирус кори (род Morbillivirus семейства Paramyxoviridae) имеет геном, представленный одноцепочечной молекулой РНК отрицательной полярности. Для его генов М и F был установлен интересный механизм. мРНК этих генов имеют длинные НТО, на их долю приходится ~6,4 % всей мРНК. Хотя эти гены непосредственно не участвуют в репликации, 3'-НТО мРНК гена М увеличивает скорость накопления белка М и тем самым запускает репликацию генома. В то же время 5'-НТО мРНК гена F снижает образование белка F и тем самым подавляет репликацию[68].
Клиническое значение
[править | править код]Так как нетранслируемые области играют важнейшие роли в регуляции экспрессии генов, различные изменения, затрагивающие эти области, нередко ведут к появлению болезненных состояний, таких как наследственная тромбоцитемия[англ.], рак молочной железы, синдром ломкой Х-хромосомы, биполярное аффективное расстройство, болезнь Альцгеймера и других[69]. В представленных ниже схемах показаны связи между мутациями, затрагивающими тот или иной функциональный элемент 3'-НТО и 5'-НТО, и различными заболеваниями.
См. также
[править | править код]Примечания
[править | править код]- ↑ Коничев, Севастьянова, 2012, с. 108.
- ↑ Barrett et. al., 2013, p. 9.
- ↑ 1 2 3 4 5 6 7 8 9 Mignone F., Gissi C., Liuni S., Pesole G. Untranslated regions of mRNAs. (англ.) // Genome biology. — 2002. — Vol. 3, no. 3. — P. 0004. — PMID 11897027.
- ↑ Humbio: 3'- или 5'-нетранслируемые области (3'- or 5'-non-translated regions, 3'-NTR or 5'-NTR). Дата обращения: 2 мая 2014. Архивировано 3 мая 2014 года.
- ↑ Rhind N., Chen Z., Yassour M., Thompson D. A., Haas B. J., Habib N., Wapinski I., Roy S., Lin M. F., Heiman D. I., Young S. K., Furuya K., Guo Y., Pidoux A., Chen H. M., Robbertse B., Goldberg J. M., Aoki K., Bayne E. H., Berlin A. M., Desjardins C. A., Dobbs E., Dukaj L., Fan L., FitzGerald M. G., French C., Gujja S., Hansen K., Keifenheim D., Levin J. Z., Mosher R. A., Müller C. A., Pfiffner J., Priest M., Russ C., Smialowska A., Swoboda P., Sykes S. M., Vaughn M., Vengrova S., Yoder R., Zeng Q., Allshire R., Baulcombe D., Birren B. W., Brown W., Ekwall K., Kellis M., Leatherwood J., Levin H., Margalit H., Martienssen R., Nieduszynski C. A., Spatafora J. W., Friedman N., Dalgaard J. Z., Baumann P., Niki H., Regev A., Nusbaum C. Comparative functional genomics of the fission yeasts. (англ.) // Science (New York, N.Y.). — 2011. — Vol. 332, no. 6032. — P. 930—936. — doi:10.1126/science.1203357. — PMID 21511999.
- ↑ 1 2 Pesole G., Mignone F., Gissi C., Grillo G., Licciulli F., Liuni S. Structural and functional features of eukaryotic mRNA untranslated regions. (англ.) // Gene. — 2001. — Vol. 276, no. 1-2. — P. 73—81. — PMID 11591473.
- ↑ Hughes M. J., Andrews D. W. A single nucleotide is a sufficient 5' untranslated region for translation in an eukaryotic in vitro system. (англ.) // FEBS letters. — 1997. — Vol. 414, no. 1. — P. 19—22. — PMID 9305724.
- ↑ Grabowski P. J., Black D. L. Alternative RNA splicing in the nervous system. (англ.) // Progress in neurobiology. — 2001. — Vol. 65, no. 3. — P. 289—308. — PMID 11473790.
- ↑ Pesole G., Bernardi G., Saccone C. Isochore specificity of AUG initiator context of human genes. (англ.) // FEBS letters. — 1999. — Vol. 464, no. 1-2. — P. 60—62. — PMID 10611483.
- ↑ Duret L., Mouchiroud D., Gautier C. Statistical analysis of vertebrate sequences reveals that long genes are scarce in GC-rich isochores. (англ.) // Journal of molecular evolution. — 1995. — Vol. 40, no. 3. — P. 308—317. — PMID 7723057.
- ↑ Jansen R. P. mRNA localization: message on the move. (англ.) // Nature reviews. Molecular cell biology. — 2001. — Vol. 2, no. 4. — P. 247—256. — doi:10.1038/35067016. — PMID 11283722.
- ↑ Bashirullah A., Cooperstock R. L., Lipshitz H. D. Spatial and temporal control of RNA stability. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2001. — Vol. 98, no. 13. — P. 7025—7028. — doi:10.1073/pnas.111145698. — PMID 11416182.
- ↑ van der Velden A. W., Thomas A. A. The role of the 5' untranslated region of an mRNA in translation regulation during development. (англ.) // The international journal of biochemistry & cell biology. — 1999. — Vol. 31, no. 1. — P. 87—106. — PMID 10216946.
- ↑ Walczak R., Westhof E., Carbon P., Krol A. A novel RNA structural motif in the selenocysteine insertion element of eukaryotic selenoprotein mRNAs. (англ.) // RNA (New York, N.Y.). — 1996. — Vol. 2, no. 4. — P. 367—379. — PMID 8634917.
- ↑ Conne B., Stutz A., Vassalli J. D. The 3' untranslated region of messenger RNA: A molecular 'hotspot' for pathology? (англ.) // Nature medicine. — 2000. — Vol. 6, no. 6. — P. 637—641. — doi:10.1038/76211. — PMID 10835679.
- ↑ Sweeney R., Fan Q., Yao M. C. Antisense ribosomes: rRNA as a vehicle for antisense RNAs. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1996. — Vol. 93, no. 16. — P. 8518—8523. — PMID 8710902.
- ↑ Timchenko L. T. Myotonic dystrophy: the role of RNA CUG triplet repeats. (англ.) // American journal of human genetics. — 1999. — Vol. 64, no. 2. — P. 360—364. — doi:10.1086/302268. — PMID 9973273.
- ↑ Anderson L., Seilhamer J. A comparison of selected mRNA and protein abundances in human liver. (англ.) // Electrophoresis. — 1997. — Vol. 18, no. 3-4. — P. 533—537. — doi:10.1002/elps.1150180333. — PMID 9150937.
- ↑ Kozak M. An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs. (англ.) // Nucleic acids research. — 1987. — Vol. 15, no. 20. — P. 8125—8148. — PMID 3313277.
- ↑ Maitra U., Stringer E. A., Chaudhuri A. Initiation factors in protein biosynthesis. (англ.) // Annual review of biochemistry. — 1982. — Vol. 51. — P. 869—900. — doi:10.1146/annurev.bi.51.070182.004253. — PMID 7051967.
- ↑ Michel Y. M., Poncet D., Piron M., Kean K. M., Borman A. M. Cap-Poly(A) synergy in mammalian cell-free extracts. Investigation of the requirements for poly(A)-mediated stimulation of translation initiation. (англ.) // The Journal of biological chemistry. — 2000. — Vol. 275, no. 41. — P. 32268—32276. — doi:10.1074/jbc.M004304200. — PMID 10922367.
- ↑ Xiong W., Hsieh C. C., Kurtz A. J., Rabek J. P., Papaconstantinou J. Regulation of CCAAT/enhancer-binding protein-beta isoform synthesis by alternative translational initiation at multiple AUG start sites. (англ.) // Nucleic acids research. — 2001. — Vol. 29, no. 14. — P. 3087—3098. — PMID 11452034.
- ↑ Rogozin I. B., Kochetov A. V., Kondrashov F. A., Koonin E. V., Milanesi L. Presence of ATG triplets in 5' untranslated regions of eukaryotic cDNAs correlates with a 'weak' context of the start codon. (англ.) // Bioinformatics. — 2001. — Vol. 17, no. 10. — P. 890—900. — PMID 11673233.
- ↑ Cassan M., Rousset J. P. UAG readthrough in mammalian cells: effect of upstream and downstream stop codon contexts reveal different signals. (англ.) // BMC molecular biology. — 2001. — Vol. 2. — P. 3. — PMID 11242562.
- ↑ Luukkonen B. G., Tan W., Schwartz S. Efficiency of reinitiation of translation on human immunodeficiency virus type 1 mRNAs is determined by the length of the upstream open reading frame and by intercistronic distance. (англ.) // Journal of virology. — 1995. — Vol. 69, no. 7. — P. 4086—4094. — PMID 7769666.
- ↑ 1 2 Vilela C., Ramirez C. V., Linz B., Rodrigues-Pousada C., McCarthy J. E. Post-termination ribosome interactions with the 5'UTR modulate yeast mRNA stability. (англ.) // The EMBO journal. — 1999. — Vol. 18, no. 11. — P. 3139—3152. — doi:10.1093/emboj/18.11.3139. — PMID 10357825.
- ↑ Svitkin Y. V., Pause A., Haghighat A., Pyronnet S., Witherell G., Belsham G. J., Sonenberg N. The requirement for eukaryotic initiation factor 4A (elF4A) in translation is in direct proportion to the degree of mRNA 5' secondary structure. (англ.) // RNA (New York, N.Y.). — 2001. — Vol. 7, no. 3. — P. 382—394. — PMID 11333019.
- ↑ Pelletier J., Kaplan G., Racaniello V. R., Sonenberg N. Cap-independent translation of poliovirus mRNA is conferred by sequence elements within the 5' noncoding region. (англ.) // Molecular and cellular biology. — 1988. — Vol. 8, no. 3. — P. 1103—1112. — PMID 2835660.
- ↑ Le S. Y., Maizel J. V. Jr. A common RNA structural motif involved in the internal initiation of translation of cellular mRNAs. (англ.) // Nucleic acids research. — 1997. — Vol. 25, no. 2. — P. 362—369. — PMID 9016566.
- ↑ Chappell S. A., Edelman G. M., Mauro V. P. A 9-nt segment of a cellular mRNA can function as an internal ribosome entry site (IRES) and when present in linked multiple copies greatly enhances IRES activity. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 2000. — Vol. 97, no. 4. — P. 1536—1541. — PMID 10677496.
- ↑ Shama S., Meyuhas O. The translational cis-regulatory element of mammalian ribosomal protein mRNAs is recognized by the plant translational apparatus. (англ.) // European journal of biochemistry / FEBS. — 1996. — Vol. 236, no. 2. — P. 383—388. — PMID 8612606.
- ↑ Brown C. E., Sachs A. B. Poly(A) tail length control in Saccharomyces cerevisiae occurs by message-specific deadenylation. (англ.) // Molecular and cellular biology. — 1998. — Vol. 18, no. 11. — P. 6548—6559. — PMID 9774670.
- ↑ Peng S. S., Chen C. Y., Shyu A. B. Functional characterization of a non-AUUUA AU-rich element from the c-jun proto-oncogene mRNA: evidence for a novel class of AU-rich elements. (англ.) // Molecular and cellular biology. — 1996. — Vol. 16, no. 4. — P. 1490—1499. — PMID 8657122.
- ↑ Hentze M. W., Kühn L. C. Molecular control of vertebrate iron metabolism: mRNA-based regulatory circuits operated by iron, nitric oxide, and oxidative stress. (англ.) // Proceedings of the National Academy of Sciences of the United States of America. — 1996. — Vol. 93, no. 16. — P. 8175—8182. — PMID 8710843.
- ↑ Hentze M. W., Kulozik A. E. A perfect message: RNA surveillance and nonsense-mediated decay. (англ.) // Cell. — 1999. — Vol. 96, no. 3. — P. 307—310. — PMID 10025395.
- ↑ Kataoka N., Yong J., Kim V. N., Velazquez F., Perkinson R. A., Wang F., Dreyfuss G. Pre-mRNA splicing imprints mRNA in the nucleus with a novel RNA-binding protein that persists in the cytoplasm. (англ.) // Molecular cell. — 2000. — Vol. 6, no. 3. — P. 673—682. — PMID 11030346.
- ↑ 1 2 Shyu A. B., Wilkinson M. F. The double lives of shuttling mRNA binding proteins. (англ.) // Cell. — 2000. — Vol. 102, no. 2. — P. 135—138. — PMID 10943833.
- ↑ Ruiz-Echevarría M. J., Peltz S. W. The RNA binding protein Pub1 modulates the stability of transcripts containing upstream open reading frames. (англ.) // Cell. — 2000. — Vol. 101, no. 7. — P. 741—751. — PMID 10892745.
- ↑ Xu N., Chen C. Y., Shyu A. B. Versatile role for hnRNP D isoforms in the differential regulation of cytoplasmic mRNA turnover. (англ.) // Molecular and cellular biology. — 2001. — Vol. 21, no. 20. — P. 6960—6971. — doi:10.1128/MCB.21.20.6960-6971.2001. — PMID 11564879.
- ↑ Zaidi S. H., Malter J. S. Amyloid precursor protein mRNA stability is controlled by a 29-base element in the 3'-untranslated region. (англ.) // The Journal of biological chemistry. — 1994. — Vol. 269, no. 39. — P. 24007—24013. — PMID 7929051.
- ↑ Ainger K., Avossa D., Diana A. S., Barry C., Barbarese E., Carson J. H. Transport and localization elements in myelin basic protein mRNA // J Cell Biol.. — 1997. — Т. 138, № 5. — С. 1077—1087. Архивировано 20 августа 2014 года.
- ↑ Bashirullah A., Cooperstock R. L., Lipshitz H. D. Spatial and temporal control of RNA stability // Proc Natl Acad Sci USA.. — 2001. — Т. 98, № 13. — С. 7025—7028. — doi:10.1083/jcb.138.5.1077.
- ↑ St Johnston D., Beuchle D., Nüsslein-Volhard C. Staufen, a gene required to localize maternal RNAs in the Drosophila egg. (англ.) // Cell. — 1991. — Vol. 66, no. 1. — P. 51—63. — PMID 1712672.
- ↑ Macdonald P. M., Kerr K., Smith J. L., Leask A. RNA regulatory element BLE1 directs the early steps of bicoid mRNA localization. (англ.) // Development (Cambridge, England). — 1993. — Vol. 118, no. 4. — P. 1233—1243. — PMID 8269850.
- ↑ Chan A. P., Kloc M., Etkin L. D. fatvg encodes a new localized RNA that uses a 25-nucleotide element (FVLE1) to localize to the vegetal cortex of Xenopus oocytes. (англ.) // Development (Cambridge, England). — 1999. — Vol. 126, no. 22. — P. 4943—4953. — PMID 10529413.
- ↑ Mowry K. L., Melton D. A. Vegetal messenger RNA localization directed by a 340-nt RNA sequence element in Xenopus oocytes. (англ.) // Science (New York, N.Y.). — 1992. — Vol. 255, no. 5047. — P. 991—994. — PMID 1546297.
- ↑ Flavio Mignone, Graziano Pesole. mRNA Untranslated Regions (UTRs) // eLS. — С. 1—5. — doi:10.1002/9780470015902.a0005009.pub2. Архивировано 5 сентября 2014 года.
- ↑ Barrett et. al., 2013, с. 32.
- ↑ Barrett et. al., 2013, с. 32—33.
- ↑ Edwards T. E., Ferré-D'Amaré A. R. Crystal structures of the thi-box riboswitch bound to thiamine pyrophosphate analogs reveal adaptive RNA-small molecule recognition. (англ.) // Structure (London, England : 1993). — 2006. — Vol. 14, no. 9. — P. 1459—1468. — doi:10.1016/j.str.2006.07.008. — PMID 16962976.
- ↑ Льюин Б. Гены. — БИНОМ, 2012. — С. 144. — 896 с. — ISBN 978-5-94774-793-5.
- ↑ Н. В. Равин, С. В. Шестаков. Геном прокариот // Вавиловский журнал генетики и селекции. — 2013. — Т. 17, № 4/2. — С. 972—984. Архивировано 31 мая 2014 года.
- ↑ 1 2 Brown, T.A. Genomes 3 (неопр.). — New York, New York: Garland Science Publishing, 2007. — С. 397. — ISBN 0 8153 4138 5.
- ↑ John W. Pelley. Elsevier’s Integrated Review Biochemistry. — 2nd Edition. — 2012. — ISBN 978-0-32307-446-9.
- ↑ Al-Qahtani A., Mensa-Wilmot K. A 5' untranslated region which directs accurate and robust translation by prokaryotic and mammalian ribosomes. (англ.) // Nucleic acids research. — 1996. — Vol. 24, no. 6. — P. 1173—1174. — PMID 8604355.
- ↑ 1 2 Jian Zhang. Gene expression in Archaea: Studies of transcriptional promoters, messenger RNA processing, and five prime untranslated regions in Methanocaldococcus jannashchii. — 2009. Архивировано 31 мая 2014 года.
- ↑ Naville M., Gautheret D. Transcription attenuation in bacteria: theme and variations. (англ.) // Briefings in functional genomics & proteomics. — 2009. — Vol. 8, no. 6. — P. 482—492. — doi:10.1093/bfgp/elp025. — PMID 19651704.
- ↑ Riboswitches: A Common RNA Regulatory Element. Дата обращения: 31 мая 2014. Архивировано 31 мая 2014 года.
- ↑ Nudler E., Mironov A. S. The riboswitch control of bacterial metabolism. (англ.) // Trends in biochemical sciences. — 2004. — Vol. 29, no. 1. — P. 11—17. — doi:10.1016/j.tibs.2003.11.004. — PMID 14729327.
- ↑ López-Garrido J., Puerta-Fernández E., Casadesús J. A eukaryotic-like 3' untranslated region in Salmonella enterica hilD mRNA. (англ.) // Nucleic acids research. — 2014. — Vol. 42, no. 9. — P. 5894—5906. — doi:10.1093/nar/gku222. — PMID 24682814.
- ↑ Wilting R., Schorling S., Persson B. C., Böck A. Selenoprotein synthesis in archaea: identification of an mRNA element of Methanococcus jannaschii probably directing selenocysteine insertion. (англ.) // Journal of molecular biology. — 1997. — Vol. 266, no. 4. — P. 637—641. — doi:10.1006/jmbi.1996.0812. — PMID 9102456.
- ↑ Brenneis M., Hering O., Lange C., Soppa J. Experimental characterization of Cis-acting elements important for translation and transcription in halophilic archaea. (англ.) // PLoS genetics. — 2007. — Vol. 3, no. 12. — P. e229. — doi:10.1371/journal.pgen.0030229. — PMID 18159946.
- ↑ Kosuke Fujishima, Akio Kanai. Diversity, Function, and Processing of Archaeal Non-Coding RNAs // Sakura Y. Kato Archaea: Structure, Habitats and Ecological Significance. — Nova Science Publishers, Inc., 2011. — С. 69—94. — ISBN 978-1-61761-932-8. Архивировано 31 мая 2014 года.
- ↑ Thompson S. R. Tricks an IRES uses to enslave ribosomes. (англ.) // Trends in microbiology. — 2012. — Vol. 20, no. 11. — P. 558—566. — doi:10.1016/j.tim.2012.08.002. — PMID 22944245.
- ↑ Kieft J. S. Viral IRES RNA structures and ribosome interactions. (англ.) // Trends in biochemical sciences. — 2008. — Vol. 33, no. 6. — P. 274—283. — doi:10.1016/j.tibs.2008.04.007. — PMID 18468443.
- ↑ Fan Q., Treder K., Miller W. A. Untranslated regions of diverse plant viral RNAs vary greatly in translation enhancement efficiency. (англ.) // BMC biotechnology. — 2012. — Vol. 12. — P. 22. — doi:10.1186/1472-6750-12-22. — PMID 22559081.
- ↑ Dreher T. W. FUNCTIONS OF THE 3'-UNTRANSLATED REGIONS OF POSITIVE STRAND RNA VIRAL GENOMES. (англ.) // Annual review of phytopathology. — 1999. — Vol. 37. — P. 151—174. — doi:10.1146/annurev.phyto.37.1.151. — PMID 11701820.
- ↑ Takeda M., Ohno S., Seki F., Nakatsu Y., Tahara M., Yanagi Y. Long untranslated regions of the measles virus M and F genes control virus replication and cytopathogenicity. (англ.) // Journal of virology. — 2005. — Vol. 79, no. 22. — P. 14346—14354. — doi:10.1128/JVI.79.22.14346-14354.2005. — PMID 16254369.
- ↑ Chatterjee S., Pal J. K. Role of 5'- and 3'-untranslated regions of mRNAs in human diseases. (англ.) // Biology of the cell / under the auspices of the European Cell Biology Organization. — 2009. — Vol. 101, no. 5. — P. 251—262. — doi:10.1042/BC20080104. — PMID 19275763.
Литература
[править | править код]- Спирин А. С. Молекулярная биология. Рибосомы и биосинтез белка. — М.: Издательский центр «Академия», 2011. — 496 с. — ISBN 978-5-7695-6668-4.
- Коничев А. С., Севастьянова Г. А. Молекулярная биология. — Издательский центр «Академия», 2012. — 400 с. — ISBN 978-5-7695-9147-1.
- Lucy W. Barrett, Sue Fletcher, Steve D. Wilton. Untranslated Gene Regions and Other Non-coding Elements. — SpringerBriefs in Biochemistry and Molecular Biology, 2013. — 57 p. — ISBN 978-3-0348-0679-4.
Ссылки
[править | править код]- UTRdb — база данных, содержащая информацию о последовательностях UTR и их функциональной нагрузке различных мРНК. Дата обращения: 30 мая 2014. Архивировано 9 августа 2020 года. (англ.)
- Кондратенко, Юлия. МикроРНК уменьшают шум экспрессии генов. // Сайт Biomolecula.ru (2 июня 2015). Дата обращения: 5 апреля 2018. Архивировано 18 марта 2018 года.
Эта статья входит в число избранных статей русскоязычного раздела Википедии. |

