Каталитическая триада

Каталитическая триада — это набор из трех скоординированных аминокислот, которые можно найти в активном центре некоторых ферментов.[1][2] Каталитические триады чаще всего встречаются в ферментах гидролаз и трансфераз (например, протеазы, амидазы, эстеразы, ацилазы, липазы и β-лактамазы). Триада кислота-основание-нуклеофил является распространенным мотивом для образования нуклеофильного остатка для ковалентного катализа. Остатки образуют сеть реле заряда для поляризации и активации нуклеофила, который атакует субстрат, образуя ковалентный промежуточный продукт, который затем гидролизуется с высвобождением продукта и регенерацией свободного фермента. Нуклеофилом чаще всего является аминокислота серин или цистеин, но иногда треонин или даже селеноцистеин. Трехмерная структура фермента объединяет триадные остатки в точной ориентации, даже если они могут находиться далеко друг от друга в последовательности (первичная структура).[3]
Помимо дивергентной эволюции функции (и даже нуклеофила триады), каталитические триады демонстрируют одни из лучших примеров конвергентной эволюции. Химические ограничения катализа привели к тому, что одно и то же строение каталитических центров развилось независимо по крайней мере в 23 отдельных суперсемействах.[2] Вследствие этого, их механизм действия является одним из наиболее изученных в биохимии.[4][5]
История[править | править код]
Ферменты трипсин и химотрипсин были впервые очищены в 1930-х годах.[6] Для них серин был идентифицирован как каталитический нуклеофил (путем модификации диизопропилфторфосфата) в 1950-х годах.[7] Структура химотрипсина была изучена с помощью рентгеновской кристаллографии в 1960-х годах, что показало ориентацию каталитической триады в активном центре.[8] Другие протеазы, которые были секвенированы и выровнены, чтобы выявить семейство родственных протеаз,[9][10][11] теперь называется семейством S1. Одновременно в структурах эволюционно не связанных протеаз папаина и субтилизина были обнаружены аналогичные триады. В конце 1960-х годов был предложен механизм «реле заряда», который подразумевает активацию нуклеофила другими членами триады.[12] Поскольку в 1970-х и 80-х годах с помощью рентгеновской кристаллографии было изучено все больше протеазных структур, были обнаружены гомологичные (например, протеаза TEV) и аналогичные (например, папаин) триады.[13][14][15] Система классификации MEROPS в 1990-х и 2000-х годах начала классифицировать протеазы по структурно связанным суперсемействам ферментов и, таким образом, действует как база данных конвергентной эволюции триад в более чем 20 суперсемействах.[16][17] Понимание того, как химические ограничения эволюции привели к конвергенции стольких семейств ферментов с одной и той же геометрией триад, появилось в 2010-х годах.[2]
С момента первоначального открытия каталитических триад, их точный каталитический механизм подвергался все более и более детальным исследованиям. В 1990-х и 2000-х годах особое внимание уделялось вопросу о том, способствуют ли водородные связи с низким барьером катализу,[18][19][20] или достаточно обычной водородной связи, чтобы объяснить механизм.[21][22] Огромный объём работ по ковалентному катализу с реле заряда, используемому каталитическими триадами, привел к тому, что этот механизм лучше всего охарактеризован во всей биохимии.[4][5]
Функция[править | править код]
Ферменты, содержащие каталитическую триаду, используют её для одного из двух типов реакций: либо для расщепления субстрата (гидролазы), либо для переноса одной части субстрата на второй субстрат (трансферазы). Триады представляют собой взаимозависимый набор остатков в активном центре фермента и действуют совместно с другими остатками (например, сайт связывания и оксианионное отверстие) для достижения нуклеофильного катализа. Эти остатки триады действуют вместе, чтобы сделать нуклеофильный элемент высокореактивным, образуя ковалентный промежуточный продукт с субстратом, который затем растворяется для завершения катализа.
Механизм[править | править код]
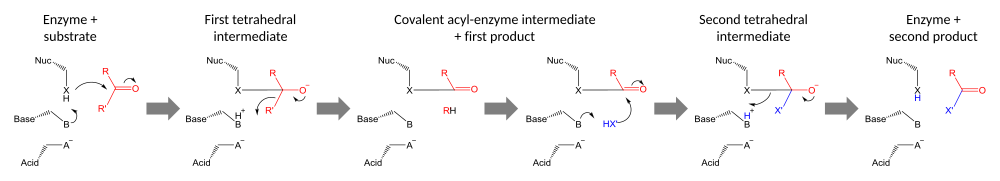
Каталитические триады выполняют ковалентный катализ, используя остаток в качестве нуклеофила. Реакционная способность нуклеофильного остатка увеличивается за счет функциональных групп других членов триады. Нуклеофил поляризован и ориентирован основанием, которое само связывается и стабилизируется кислотой.
Катализ проводится в два этапа. Во-первых, активированный нуклеофил атакует карбонильный углерод и заставляет карбонильный кислород принять электронную пару, что приводит к тетраэдрическому промежуточному соединению. Наращивание отрицательного заряда на этом промежуточном продукте обычно стабилизируется оксианионной дырой в активном центре. Промежуточный продукт затем коллапсирует обратно до карбонила, выбрасывая первую половину субстрата, но оставляя вторую половину все ещё ковалентно связанной с ферментом в качестве промежуточного ацил-фермента. Хотя общий кислотный катализ разрушения первого и второго тетраэдрических интермедиатов может происходить по пути, показанному на диаграмме, доказательства, подтверждающие этот механизм с химотрипсином[23], были оспорены.[24]
Вторая стадия катализа — это разделение промежуточного ацил-фермента путем атаки на второй субстрат. Если этот субстрат — вода, то результатом будет гидролиз; если это органическая молекула, то результатом является перенос этой молекулы на первый субстрат. Атака на второй субстрат формирует новый тетраэдрический промежуточный продукт, который распадается путем выброса нуклеофила фермента, высвобождения второго продукта и регенерации свободного фермента.
Идентичность членов триады[править | править код]

Нуклеофил[править | править код]
Боковая цепь нуклеофильного остатка выполняет ковалентный катализ на субстрате. Неподеленная пара электронов, присутствующая на кислороде или сере, атакует электроположительный карбонильный углерод.[3] 20 природных биологических аминокислот не содержат достаточно нуклеофильных функциональных групп для многих сложных каталитических реакций . Включение нуклеофила в триаду увеличивает его реакционную способность для эффективного катализа. Наиболее часто используемые нуклеофилы — это гидроксил (ОН) серина и тиол / тиолат-ион (SH / S-) цистеина.[2] Альтернативно, треониновые протеазы используют вторичный гидроксил треонина, однако из-за стерической помехи дополнительной метильной группы боковой цепи такие протеазы используют свой N -концевой амид в качестве основания, а не отдельную аминокислоту.[1][25]
Использование кислорода или серы в качестве нуклеофильного атома вызывает незначительные различия в катализе. По сравнению с кислородом дополнительная d-орбиталь серы делает её больше (на 0,4 Å)[26] и мягче, позволяет ей образовывать более длинные связи (dC-X и dX-H больше в 1,3 раза) и дает более низкое значение pKa (на 5 единиц).[27] Следовательно, серин в большей степени, чем цистеин, зависит от оптимальной ориентации членов кислотно-основной триады для снижения его pKa для достижения согласованного депротонирования с катализом.[2] Низкий pKa цистеина работает для него невыгодно при разделении первого тетраэдрического промежуточного соединения, поскольку непродуктивное обращение исходной нуклеофильной атаки является более благоприятным продуктом распада. Таким образом, основание триады предпочтительно ориентировано на протонирование амида уходящей группы, чтобы гарантировать, что он выбрасывается, оставляя серу фермента ковалентно связанной с N-концом субстрата. Наконец, разделение ацил-фермента (для высвобождения С-конца субстрата) требует повторного протонирования серина, тогда как цистеин может уйти в виде S-. С точки зрения стерического эффекта сера цистеина также образует более длинные связи и имеет более объемный радиус Ван-дер-Ваальса и при мутации в серин может быть захвачен в непродуктивных ориентациях в активном центре.
Очень редко атом селена аминокислоты селеноцистеина используется в качестве нуклеофила.[28] Депротонированное состояние Se- сильно предпочтительнее в каталитической триаде.
Основание[править | править код]
Поскольку никакие природные аминокислоты не являются сильно нуклеофильными, основание в каталитической триаде поляризует и депротонирует нуклеофил для увеличения его реакционной способности.[3] Кроме того, он протонирует первый продукт, чтобы способствовать его уходу из группы.
Основанием чаще всего является гистидин, поскольку его pKa позволяет осуществлять эффективный щелочной катализ, водородную связь с кислотным остатком и депротонирование нуклеофильного остатка.[1] β-лактамазы, такие как ТЕМ-1, используют остаток лизина в качестве основания. Поскольку pKa лизина очень велико (pKa = 11), глутамат и несколько других остатков действуют как кислота, стабилизируя его депротонированное состояние во время каталитического цикла.[29][30] Треониновые протеазы используют свой N-концевой амид в качестве основания, поскольку стерическое вытеснение метила каталитического треонина препятствует тому, чтобы другие остатки находились достаточно близко друг к другу.[31][32]
Кислота[править | править код]
Кислотный член триады образует водородную связь с основным остатком. Это выравнивает основный остаток, ограничивая вращение его боковой цепи, и поляризует его, стабилизируя его положительный заряд.[3] Две аминокислоты имеют кислые боковые цепи при физиологическом pH (аспартат или глутамат) и поэтому наиболее часто используются для этого члена триады. Цитомегаловирусная протеаза использует пару гистидинов, один как обычно, а другой как кислоту.[1] Второй гистидин не является такой эффективной кислотой, как более распространенный аспартат или глутамат, что приводит к более низкой каталитической эффективности. В некоторых ферментах кислотный член триады менее необходим, а некоторые действуют только как диада. Например, папаин [b] использует аспарагин в качестве своего третьего члена триады, который ориентирует гистидиновое основание, но не действует как кислота. Точно так же протеаза вируса гепатита A [c] содержит упорядоченную воду в том месте, где должен находиться кислотный остаток.
Примеры триад[править | править код]

Сер-Гис-Асп[править | править код]
Мотив серин-гистидин-аспартат является одним из наиболее подробно описанных каталитических мотивов в биохимии.[3] Примером этой триады является химотрипсин,[d] модельная сериновая протеаза из суперсемейства PA, которая использует свою триаду для гидролиза белковых скелетов. Аспартат связан водородными связями с гистидином, увеличивая pKa его имидазольного азота с 7 до примерно 12. Это позволяет гистидину действовать как мощная общая основа и активировать сериновый нуклеофил. Он также имеет оксианионное отверстие, состоящее из нескольких амидов основной цепи, которое стабилизирует накопление заряда на промежуточных соединениях. Основание гистидина помогает первой уходящей группе, отдавая протон, а также активирует гидролитический водный субстрат, отводя протон, поскольку оставшийся ОН- атакует промежуточный ацил-фермент.
Та же самая триада также конвергентно эволюционировала в α / β гидролазах, таких как некоторые липазы и эстеразы, однако ориентация членов триады обратная.[33][34] Кроме того, было обнаружено, что ацетилгидролаза головного мозга (которая имеет ту же форму, что и небольшой G-белок) также имеет эту триаду. Эквивалентная триада Сер-Гис-Глу используется в ацетилхолинэстеразе.
Цис-Гис-Асп[править | править код]
Вторая наиболее изученная триада — это мотив цистеин-гистидин-аспартат.[2] Этот набор триад используют несколько семейств цистеиновых протеаз[e] и папаин[f]. Триада действует аналогично триадам сериновых протеаз с некоторыми заметными отличиями. Из-за низкого pKa цистеина важность Асп для катализа варьируется, и некоторые цистеиновые протеазы эффективно являются Цис-Гис-диадами (например, протеазой вируса гепатита A), тогда как в других цистеин депротонируется ещё до начала катализа (например, папаин).[35] Эта триада также используется некоторыми амидазами, такими как N-гликаназа, для гидролиза непептидных связей CN.[36]
Сер-Гис-Гис[править | править код]
Триада цитомегаловирусной протеазы [g] использует гистидин в качестве членов как кислотной, так и основной триады. Удаление кислотного гистидина приводит только к 10-кратной потере активности (по сравнению с более чем 10 000-кратной, когда аспартат удаляется из химотрипсина). Эта триада была интерпретирована как возможный способ создания менее активного фермента для контроля скорости расщепления.[25]
Сер-Глу-Асп[править | править код]
Необычная триада обнаружена в протеазах селдолизина.[h] Низкое значение pKa глутаматкарбоксилатной группы означает, что она действует как основание в триаде только при очень низком pH. Предполагается, что эта триада является адаптацией к специфической среде, такой как кислые горячие источники (например, кумамолизин) или клеточные лизосомы (например, трипептидилпептидаза).[25]
Цис-Гис-Сер[править | править код]
Эндотелиальная протеаза вазохибин [i] использует цистеин в качестве нуклеофила, но серин для координации гистидинового основания.[37][38] Несмотря на то, что серин является слабой кислотой, он по-прежнему эффективен в ориентации гистидина в каталитической триаде. Некоторые гомологи альтернативно содержат треонин вместо серина в кислотном месте.
Тре-Nконц, Сер-Nконц и Цис-Nконц[править | править код]
Треониновые протеазы, такие как субъединица протеасомы [j] и орнитинацилтрансферазы [k] используют вторичный гидроксил треонина аналогично использованию первичного гидроксила серина.[31][32] Однако из-за стерического вмешательства дополнительной метильной группы треонина основным членом триады являетвой амид, который поляризует упорядоченную воду, которая, в свою очередь, депротонирует каталитический гидроксил для увеличения его реакционной способности.[1][25] Точно так же существуют эквивалентные конфигурации «только серин» и «только цистеин», такие как пенициллинацилаза G [l] и пенициллинацилаза V [m] которые эволюционно связаны с протеасомными протеазами. Опять же, они используют их N-концевой амид в качестве основания.
Сер-цисСер-Лиз[править | править код]
Эта необычная триада встречается только в одном суперсемействе амидаз. В этом случае лизин поляризует средний серин.[39] Затем средний серин образует две сильные водородные связи с нуклеофильным серином, чтобы активировать его (одну с гидроксилом боковой цепи, а другую с амидом основной цепи). Средний серин удерживается в необычной цис- ориентации для облегчения точных контактов с двумя другими остатками триады. Триада необычна ещё и тем, что лизин и цис-серин действуют как основание при активации каталитического серина, но один и тот же лизин также выполняет роль кислотного члена, а также устанавливает ключевые структурные контакты.[40]
Sec-Гис-Глу[править | править код]
Редкая, но встречающаяся в природе аминокислота селеноцистеин (Sec) также может быть найдена в качестве нуклеофила в некоторых каталитических триадах.[28] Селеноцистеин похож на цистеин, но содержит атом селена вместо серы. Примером может служить активный центр тиоредоксинредуктазы, который использует селен для восстановления дисульфида в тиоредоксине.
Спроектированные триады[править | править код]
В дополнение к естественным типам каталитических триад, белковая инженерия использовалась для создания вариантов ферментов с ненативными аминокислотами или полностью синтетическими аминокислотами.[41] Каталитические триады также были вставлены в некаталитические белки или имитаторы белков.
Кислородный нуклеофил субтилизина (сериновой протеазы) заменен на серу,[42][43] селен[44] или теллур.[45] Цистеин и селеноцистеин были вставлены с помощью мутагенеза, тогда как неприродная аминокислота, теллуроцистеин, была вставлена с использованием ауксотрофных клеток, питаемых синтетическим теллуроцистеином. Все эти элементы находятся в 16-м столбце таблицы Менделеева (халькогены), поэтому обладают схожими свойствами.[46][47] В каждом случае изменение нуклеофила уменьшало активность протеазы фермента, но увеличивало другую активность. Нуклеофил серы улучшал активность ферментов трансферазы (иногда называемой субтилигазой). Нуклеофилы селена и теллура превратили фермент в оксидоредуктазу Когда нуклеофил протеазы TEV превращался из цистеина в серин, его протеазная активность сильно снижалась, но могла быть восстановлена путем направленной эволюции.[48]
Некаталитические белки использовались в качестве каркасов, в них были вставлены каталитические триады, которые затем были улучшены путем направленной эволюции. Триада Сер-Гис-Асп была вставлена в антитело[49] а также в ряд других белков.[50] Точно так же имитаторы каталитических триад были созданы в небольших органических молекулах, таких как диарилдиселенид,[51][52] и отображены на более крупных полимерах, таких как смолы Меррифилда,[53] и самособирающихся коротких пептидных наноструктурах.[54]
Дивергентная эволюция[править | править код]
Сложность сети активных центров заставляет остатки, участвующие в катализе (и остатки, контактирующие с ними), быть высоко эволюционно консервативными.[55] Однако есть примеры дивергентной эволюции в каталитических триадах, как в катализируемой реакции, так и в остатках, используемых в катализе. Триада остается ядром активного центра, но эволюционно приспособлена для выполнения различных функций.[56][57] Некоторые белки, называемые псевдоферментами, выполняют некаталитические функции (например, регуляцию путем ингибирующего связывания) и имеют накопленные мутации, которые инактивируют их каталитическую триаду.[58]
Изменения реакции[править | править код]
Каталитические триады осуществляют ковалентный катализ через промежуточный ацил-фермент. Если этот промежуточный продукт растворяется водой, происходит гидролиз субстрата. Однако, если промежуточное звено растворяется путем атаки на второй субстрат, то фермент действует как трансфераза. Например, атака ацильной группой приводит к реакции ацилтрансферазы. Несколько семейств ферментов трансфераз произошли от гидролаз в результате адаптации, исключающей воду и способствующей атаке второго субстрата.[59] У разных членов суперсемейства α / β-гидролаз триада Сер-Гис-Асп настраивается окружающими остатками на выполнение по меньшей мере 17 различных реакций.[34][60] Некоторые из этих реакций также достигаются с помощью механизмов, которые изменили образование или разделение промежуточного ацил-фермента, или которые не протекают через промежуточное соединение ацил-фермент.
Кроме того, альтернативный механизм трансферазы был разработан амидофосфорибозилтрансферазой, которая имеет два активных центра. [n] В первом активном центре цистеиновая триада гидролизует глутаминовый субстрат с высвобождением свободного аммиака. Затем аммиак диффундирует через внутренний туннель в ферменте ко второму активному центру, где он переносится на второй субстрат.[61][62]
Нуклеофильные изменения[править | править код]

Дивергентная эволюция остатков активного центра происходит медленно из-за сильных химических ограничений. Тем не менее, некоторые суперсемейства протеаз эволюционировали от одного нуклеофила к другому. Это может произойти, если суперсемейство (с одной и той же структурой белков) содержит семейства, использующие разные нуклеофилы.[48] Такие нуклеофильные замены происходили несколько раз в течение эволюционной истории, однако механизмы, с помощью которых это происходит, до сих пор неясны.[17]
В суперсемействах протеаз, которые содержат смесь нуклеофилов (например, клан PA), семейства обозначаются их каталитическими нуклеофилами (C = цистеиновые протеазы, S = сериновые протеазы).
| Надсемейство | Семьи | Примеры |
|---|---|---|
| Клан ПА | C3, C4, C24, C30, C37, C62, C74, C99 | Протеаза TEV (вирус травления табака) |
| S1, S3, S6, S7, S29, S30, S31, S32, S39, S46, S55, S64, S65, S75 | Химотрипсин (млекопитающие, например Bos taurus) | |
| Клан PB | C44, C45, C59, C69, C89, C95 | Предшественник амидофосфорибозилтрансферазы (Homo sapiens) |
| S45, S63 | Предшественник ацилазы пенициллина G (Escherichia coli) | |
| Т1, Т2, Т3, Т6 | Протеасома архей, бета-компонент (Thermoplasma acidophilum) | |
| Клан ПК | C26, C56 | Гамма-глутамилгидролаза (Rattus norvegicus) |
| S51 | Дипептидаза Е (кишечная палочка) | |
| Клан PD | C46 | Протеин ежа (Drosophila melanogaster) |
| N9, N10, N11 | Каталитическая субъединица А протонной АТФазы V-типа, содержащая интеин (Saccharomyces cerevisiae) | |
| Клан PE | P1 | Аминопептидаза DmpA (Ochrobactrum anthropi) |
| T5 | Предшественник орнитинацетилтрансферазы (Saccharomyces cerevisiae) |
Псевдоферменты[править | править код]
Следующим подклассом вариантов каталитических триад являются псевдоферменты, которые имеют триадные мутации, которые делают их каталитически неактивными, но способными функционировать как связывающие или структурные белки.[64][65] Например, связывающий гепарин белок азуроцидин является членом клана PA, но с глицином вместо нуклеофила и серином вместо гистидина.[66] Точно так же RHBDF1 является гомологом ромбовидных протеаз семейства S54 с аланином вместо нуклеофильного серина.[67][68] В некоторых случаях псевдоферменты могут все ещё иметь неповрежденную каталитическую триаду, но мутации в остальной части белка снимают каталитическую активность. Клан CA содержит каталитически неактивных членов с мутантными триадами (кальпамодулин имеет лизин вместо цистеинового нуклеофила) и с интактными триадами, но инактивирует мутации в другом месте (тестин крысы сохраняет триаду Цис-Гис-Асн).[69]
| Надсемейство | Семьи, содержащие псевдоферменты | Примеры |
|---|---|---|
| Клан CA | C1, C2, C19 | Кальпамодулин |
| CD клан | C14 | CFLAR |
| Клан SC | S9, S33 | Нейролигин |
| Клан SK | S14 | ClpR |
| Клан SR | S60 | Домен серотрансферрина 2 |
| Клан ST | S54 | RHBDF1 |
| Клан ПА | S1 | Азуроцидин 1 |
| Клан PB | Т1 | PSMB3 |
Конвергентная эволюция[править | править код]
 Эволюционная конвергенция сериновой и цистеиновой протеаз в направлении одной и той же каталитической организации триад кислотно-основного нуклеофила в разных суперсемействах протеаз. Показаны триады субтилизина,[o] пролилолигопептидаза,[p] протеазы TEV, и папаина. (PDB 1ST2) |
 Эволюционная конвергенция треониновой протеазыs к одной и той же "N"-концевой организации активного сайта. Показаны каталитический треонин протеасомы [q] и орнитинацетилтрансферазы.[r] (PDB 1VRA) |
Энзимология протеаз дает одни из самых ярких известных примеров конвергентной эволюции. Такое же геометрическое расположение триадных остатков встречается более чем в 20 отдельных суперсемействах ферментов. Каждое из этих суперсемейств является результатом конвергентной эволюции одного и того же расположения триад в пределах разных структурных складок. Это связано с тем, что существуют ограниченные продуктивные способы организации трех остатков триады, ферментного скелета и субстрата. Эти примеры отражают внутренние химические и физические ограничения ферментов, что приводит эволюции к неоднократному и независимому поиску равноценных решений.[1][2]
Цистеиновые и сериновые гидролазы[править | править код]
К той же геометрии триады сходятся сериновые протеазы, такие как суперсемейства химотрипсина и субтилизина. Подобная конвергентная эволюция произошла с цистеиновыми протеазами, такими как суперсемейства вирусной C3 протеазы и папаина Эти триады сходятся к почти одинаковому расположению из-за механистического сходства в механизмах протеолиза цистеина и серина.[2]
| Суперсемейство | Семейство | Примеры |
|---|---|---|
| CA | C1, C2, C6, C10, C12, C16, C19, C28, C31, C32, C33, C39, C47, C51, C54, C58, C64, C65, C66, C67, C70, C71, C76, C78, C83, C85, C86, C87, C93, C96, C98, C101 | Папаин (Карика папайя) и кальпаин (Homo sapiens) |
| CD | C11, C13, C14, C25, C50, C80, C84 | Каспаза-1 (Rattus norvegicus) и сепараза (Saccharomyces cerevisiae) |
| CE | C5, C48, C55, C57, C63, C79 | Аденаин (аденовирус человека 2 типа) |
| CF | C15 | Пироглутамилпептидаза I (Bacillus amyloliquefaciens) |
| CL | C60, C82 | Сортаза А (золотистый стафилококк) |
| СМ | C18 | Пептидаза 2 вируса гепатита C (вирус гепатита C) |
| CN | C9 | Пептидаза nsP2 вируса Синдбис (вирус Синдбис) |
| CO | C40 | Дипептидилпептидаза VI (Lysinibacillus sphaericus Архивная копия от 17 сентября 2020 на Wayback Machine) |
| CP | C97 | Пептидаза DeSI-1 (Mus musculus) |
| PA | C3, C4, C24, C30, C37, C62, C74, C99 | Протеаза TEV (вирус травления табака) |
| PB | C44, C45, C59, C69, C89, C95 | Предшественник амидофосфорибозилтрансферазы (Homo sapiens) |
| ПК | C26, C56 | Гамма-глутамилгидролаза (Rattus norvegicus) |
| PD | C46 | Протеин ежа (Drosophila melanogaster) |
| PE | P1 | Аминопептидаза DmpA (Ochrobactrum anthropi) |
| неназначенный | C7, C8, C21, C23, C27, C36, C42, C53, C75 |
| Суперсемейство | Семейство | Примеры |
|---|---|---|
| SB | S8, S53 | Субтилизин (Bacillus licheniformis) |
| SC | S9, S10, S15, S28, S33, S37 | Пролилолигопептидаза (Sus scrofa) |
| SE | S11, S12, S13 | D-Ala-D-Ala пептидаза C (Escherichia coli) |
| SF | S24, S26 | Сигнальная пептидаза I (Escherichia coli) |
| SH | S21, S73, S77, S78, S80 | Ассемблер цитомегаловируса (человеческий вирус герпеса 5) |
| SJ | S16, S50, S69 | Lon-A пептидаза (Escherichia coli) |
| SK | S14, S41, S49 | Протеаза Clp (кишечная палочка) |
| ТАК | S74 | Саморасщепляющийся белок CIMCD отростка шейки фага GA-1 (Bacillus phage GA-1) |
| SP | S59 | Нуклеопорин 145 (Homo sapiens) |
| SR | S60 | Лактоферрин (Homo sapiens) |
| SS | S66 | Муреинтетрапептидаза LD-карбоксипептидаза (Pseudomonas aeruginosa) |
| ST | S54 | Ромбовидный −1 (Drosophila melanogaster) |
| PA | S1, S3, S6, S7, S29, S30, S31, S32, S39, S46, S55, S64, S65, S75 | Химотрипсин А (Bos taurus) |
| PB | S45, S63 | Предшественник ацилазы пенициллина G (Escherichia coli) |
| ПК | S51 | Дипептидаза Е (кишечная палочка) |
| PE | P1 | Аминопептидаза DmpA (Ochrobactrum anthropi) |
| неназначенный | S48, S62, S68, S71, S72, S79, S81 |
Треониновые протеазы[править | править код]
Треониновые протеазы используют аминокислоту треонин в качестве каталитического нуклеофила. В отличие от цистеина и серина, треонин является вторичным гидроксилом (то есть имеет метильную группу). Эта метильная группа сильно ограничивает возможные ориентации триады и субстрата, поскольку метил сталкивается либо с основной цепью фермента, либо с гистидиновым основанием.[2] Когда нуклеофил сериновой протеазы был мутирован в треонин, метил занимал несколько положений, большинство из которых препятствовало связыванию субстрата.[70] Следовательно, каталитический остаток треониновой протеазы находится на её N- конце.
Известно, что два эволюционно независимых суперсемейства ферментов с разными белковыми складками используют N -концевой остаток в качестве нуклеофила: суперсемейство PB (протеасомы, использующие складку Ntn)[31] и суперсемейство PE (ацетилтрансферазы, использующие складку DOM)[32] структура активного сайта в совершенно разных белковых складках указывает на то, что активный центр эволюционировал конвергентно в этих суперсемействах.[2][25]
| Суперсемейство | Семейство | Примеры |
|---|---|---|
| PB clan | T1, T2, T3, T6 | Архейская протеасома, бета-компонент (Thermoplasma acidophilum) |
| PE clan | T5 | Орнитинацетилтрансфераза (Saccharomyces cerevisiae) |
См. также[править | править код]
Примечания[править | править код]
Заметки[править | править код]
- ↑ TEV
- ↑ Papain
- ↑ Hepatitis A virus protease
- ↑ Chymotrypsin
- ↑ TEV protease
- ↑ Papain
- ↑ Cytomegalovirus protease
- ↑ Seldolisin protease
- ↑ Vasohibin protease
- ↑ Proteasome
- ↑ Ornithine acyltransferases
- ↑ Penicillin acylase G
- ↑ Penicillin acylase V
- ↑ amidophosphoribosyltransferase
- ↑ Субтилизин
- ↑ пролилолигопептидаза
- ↑ Протеасома
- ↑ OAT
Цитаты[править | править код]
- ↑ 1 2 3 4 5 6 "Catalytic triads and their relatives". Trends Biochem. Sci. 23 (9): 347—52. 1998. doi:10.1016/S0968-0004(98)01254-7. PMID 9787641.
- ↑ 1 2 3 4 5 6 7 8 9 10 "Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad". Proc. Natl. Acad. Sci. U.S.A. 110 (8): E653—61. 2013. Bibcode:2013PNAS..110E.653B. doi:10.1073/pnas.1221050110. PMID 23382230.
- ↑ 1 2 3 4 5 Biochemistry. — ISBN 9780716749554.
- ↑ 1 2 Perutz, Max. Protein structure. New approaches to disease and therapy. — New York : W.H. Freeman and Co, 1992. — ISBN 9780716770213.
- ↑ 1 2 "Proteolytic enzymes past and present: the second golden era. Recollections, special section in honor of Max Perutz". Protein Sci. 3 (10): 1734—9. 1994. doi:10.1002/pro.5560031013. PMID 7849591.
- ↑ "Endothelin-induced vasoconstriction and release of atrial natriuretic peptides in the rat". Acta Physiol. Scand. 138 (4): 549—56. 1990. doi:10.1111/j.1748-1716.1990.tb08883.x. PMID 2141214.
- ↑ "Amino Acid Sequence in the Region of Diisopropyl Phosphoryl Binding in Dip-Trypsin". J. Am. Chem. Soc. 80 (5): 1260—1. 1958. doi:10.1021/ja01538a059.
- ↑ "Three-dimensional structure of tosyl-α-chymotrypsin". Nature. 214 (5089): 652—656. 1967. Bibcode:1967Natur.214..652M. doi:10.1038/214652a0. PMID 6049071.
- ↑ "Trypsinogen and Chymotrypsinogen as Homologous Proteins". Proc. Natl. Acad. Sci. U.S.A. 52 (4): 884—9. 1964. Bibcode:1964PNAS...52..884W. doi:10.1073/pnas.52.4.884. PMID 14224394.
- ↑ "The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships". J. Mol. Biol. 92 (2): 225—59. 1975. doi:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ↑ "Conservation and variability in the structures of serine proteinases of the chymotrypsin family". J. Mol. Biol. 258 (3): 501—37. 1996. doi:10.1006/jmbi.1996.0264. PMID 8642605.
- ↑ "Role of a buried acid group in the mechanism of action of chymotrypsin". Nature. 221 (5178): 337—40. 1969. Bibcode:1969Natur.221..337B. doi:10.1038/221337a0. PMID 5764436.
- ↑ "Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families". FEBS Lett. 194 (2): 253—7. 1986. doi:10.1016/0014-5793(86)80095-3. PMID 3000829.
- ↑ "Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications". Proc. Natl. Acad. Sci. U.S.A. 85 (21): 7872—6. 1988. Bibcode:1988PNAS...85.7872B. doi:10.1073/pnas.85.21.7872. PMID 3186696.
- ↑ "Structural basis for the substrate specificity of tobacco etch virus protease". J. Biol. Chem. 277 (52): 50564—72. 2002. doi:10.1074/jbc.M207224200. PMID 12377789.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ "Evolutionary families of peptidases". Biochem. J. 290 (1): 205—18. 1993. doi:10.1042/bj2900205. PMID 8439290.
- ↑ 1 2 "MEROPS: the peptidase database". Nucleic Acids Res. 38 (supl_1): D227—33. 2010. doi:10.1093/nar/gkp971. PMID 19892822.
- ↑ Frey PA, Whitt SA, Tobin JB (1994). "A low-barrier hydrogen bond in the catalytic triad of serine proteases". Science. 264 (5167): 1927—30. Bibcode:1994Sci...264.1927F. doi:10.1126/science.7661899. PMID 7661899.
- ↑ Ash EL, Sudmeier JL, De Fabo EC, et al. (1997). "A low-barrier hydrogen bond in the catalytic triad of serine proteases? Theory versus experiment". Science. 278 (5340): 1128—32. Bibcode:1997Sci...278.1128A. doi:10.1126/science.278.5340.1128. PMID 9353195.
- ↑ Agback P, Agback T (2018). "Direct evidence of a low barrier hydrogen bond in the catalytic triad of a Serine protease". Sci. Rep. 8 (1): 10078. Bibcode:2018NatSR...810078A. doi:10.1038/s41598-018-28441-7. PMC 6031666. PMID 29973622.
- ↑ "The low barrier hydrogen bond (LBHB) proposal revisited: the case of the Asp... His pair in serine proteases". Proteins. 55 (3): 711—23. 2004. doi:10.1002/prot.20096. PMID 15103633.
- ↑ "Energy considerations show that low-barrier hydrogen bonds do not offer a catalytic advantage over ordinary hydrogen bonds". Proc. Natl. Acad. Sci. U.S.A. 93 (24): 13665—70. 1996. Bibcode:1996PNAS...9313665W. doi:10.1073/pnas.93.24.13665. PMID 8942991.
- ↑ Fersht, A.R. (1971). "Mechanism of the Chymotrypsin-Catalyzed Hydrolysis of Amides. pH Dependence of kc and Km.' Kinetic Detection of an Intermediate". J. Am. Chem. Soc. 93: 7079—87.
- ↑ Zeeberg, B (1973). "Concerning a reported change in rate-determining step in chymotrypsin catalysis". J. Am. Chem. Soc. 95: 2734—5.
- ↑ 1 2 3 4 5 "Unconventional serine proteases: variations on the catalytic Ser/His/Asp triad configuration". Protein Sci. 17 (12): 2023—37. 2008. doi:10.1110/ps.035436.108. PMID 18824507.
- ↑ "Crystal structures of two engineered thiol trypsins". Biochemistry. 28 (24): 9264—70. 1989. doi:10.1021/bi00450a005. PMID 2611228.
- ↑ "The basic difference in catalyses by serine and cysteine proteinases resides in charge stabilization in the transition state". J. Theor. Biol. 121 (3): 323—6. 1986. doi:10.1016/s0022-5193(86)80111-4. PMID 3540454.
- ↑ 1 2 "The functional role of selenocysteine (Sec) in the catalysis mechanism of large thioredoxin reductases: proposition of a swapping catalytic triad including a Sec-His-Glu state". ChemBioChem. 6 (2): 386—94. 2005. doi:10.1002/cbic.200400276. PMID 15651042.
- ↑ "The catalytic mechanism of beta-lactamases: NMR titration of an active-site lysine residue of the TEM-1 enzyme". Proc. Natl. Acad. Sci. U.S.A. 93 (5): 1747—52. 1996. Bibcode:1996PNAS...93.1747D. doi:10.1073/pnas.93.5.1747. PMID 8700829.
- ↑ "Beta-lactamase TEM1 of E. coli. Crystal structure determination at 2.5 A resolution". FEBS Lett. 299 (2): 135—42. 1992. doi:10.1016/0014-5793(92)80232-6. PMID 1544485.
- ↑ 1 2 3 "A protein catalytic framework with an N-terminal nucleophile is capable of self-activation". Nature. 378 (6555): 416—9. 1995. Bibcode:1995Natur.378..416B. doi:10.1038/378416a0. PMID 7477383.
- ↑ 1 2 3 "DOM-fold: a structure with crossing loops found in DmpA, ornithine acetyltransferase, and molybdenum cofactor-binding domain". Protein Sci. 14 (7): 1902—10. 2005. doi:10.1110/ps.051364905. PMID 15937278.
- ↑ "Molecular basis of the general base catalysis of an α/β-hydrolase catalytic triad". J. Biol. Chem. 289 (22): 15867—79. 2014. doi:10.1074/jbc.m113.535641. PMID 24737327.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ 1 2 "How the Same Core Catalytic Machinery Catalyzes 17 Different Reactions: the Serine-Histidine-Aspartate Catalytic Triad of α/β-Hydrolase Fold Enzymes". ACS Catal. 5 (10): 6153—6176. 2015. doi:10.1021/acscatal.5b01539. PMID 28580193.
- ↑ "A theoretical study of the active sites of papain and S195C rat trypsin: implications for the low reactivity of mutant serine proteinases". Protein Sci. 5 (7): 1355—65. 1996. doi:10.1002/pro.5560050714. PMID 8819168.
- ↑ "The PUB domain functions as a p97 binding module in human peptide N-glycanase". J. Biol. Chem. 281 (35): 25502—8. 2006. doi:10.1074/jbc.M601173200. PMID 16807242.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ "Vasohibins: new transglutaminase-like cysteine proteases possessing a non-canonical Cys-His-Ser catalytic triad". Bioinformatics. 32 (10): 1441—5. 2016. doi:10.1093/bioinformatics/btv761. PMID 26794318.
- ↑ "The vasohibin family: a negative regulatory system of angiogenesis genetically programmed in endothelial cells". Arter. Thromb. Vasc. Biol. 27 (1): 37—41. 2007. doi:10.1161/01.atv.0000252062.48280.61. PMID 17095714.
- ↑ "Characterization of a novel Ser-cisSer-Lys catalytic triad in comparison with the classical Ser-His-Asp triad". J. Biol. Chem. 278 (27): 24937—43. 2003. doi:10.1074/jbc.M302156200. PMID 12711609.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ "The mechanism of the Ser-(cis)Ser-Lys catalytic triad of peptide amidases". Phys. Chem. Chem. Phys. 19 (19): 12343—12354. 2017. Bibcode:2017PCCP...1912343C. doi:10.1039/C7CP00277G. PMID 28453015. Архивировано 24 декабря 2017. Дата обращения: 9 июля 2021.
- ↑ "Minimalist active-site redesign: teaching old enzymes new tricks". Angew. Chem. 46 (18): 3212—36. 2007. doi:10.1002/anie.200604205. PMID 17450624.
- ↑ "Engineering subtilisin and its substrates for efficient ligation of peptide bonds in aqueous solution". Biochemistry. 30 (17): 4151—9. 1991. doi:10.1021/bi00231a007. PMID 2021606.
- ↑ "A designed peptide ligase for total synthesis of ribonuclease A with unnatural catalytic residues". Science. 266 (5183): 243—7. 1994. Bibcode:1994Sci...266..243J. doi:10.1126/science.7939659. PMID 7939659.
- ↑ "Crystal structure of selenosubtilisin at 2.0-A resolution". Biochemistry. 32 (24): 6157—64. 1993. doi:10.1021/bi00075a007. PMID 8512925.
- ↑ "Semisynthetic tellurosubtilisin with glutathione peroxidase activity". J. Am. Chem. Soc. 127 (33): 11588—9. 2005. doi:10.1021/ja052451v. PMID 16104720.
- ↑ Handbook of Chalcogen Chemistry. — Vol. Vol. 1: new perspectives in sulfur, selenium and tellurium. — ISBN 9781849736237.
- ↑ Electrochemistry of Metal Chalcogenides. — P. 57–75. — ISBN 9783642039669. — doi:10.1007/978-3-642-03967-6_2.
- ↑ 1 2 "Handicap-Recover Evolution Leads to a Chemically Versatile, Nucleophile-Permissive Protease". ChemBioChem. 16 (13): 1866—9. 2015. doi:10.1002/cbic.201500295. PMID 26097079.
- ↑ "Design of a serine protease-like catalytic triad on an antibody light chain displayed on the yeast cell surface". Appl. Microbiol. Biotechnol. 77 (3): 597—603. 2007. doi:10.1007/s00253-007-1197-0. PMID 17899065.
- ↑ "Design of activated serine-containing catalytic triads with atomic-level accuracy". Nat. Chem. Biol. 10 (5): 386—91. 2014. doi:10.1038/nchembio.1498. PMID 24705591.
- ↑ "Introduction of a catalytic triad increases the glutathione peroxidase-like activity of diaryl diselenides". Org. Biomol. Chem. 13 (34): 9072—82. 2015. doi:10.1039/C5OB01294E. PMID 26220806.
- ↑ "Insights into the catalytic mechanism of synthetic glutathione peroxidase mimetics". Org. Biomol. Chem. 13 (41): 10262—72. 2015. doi:10.1039/c5ob01665g. PMID 26372527.
- ↑ "Simple Design of an Enzyme-Inspired Supported Catalyst Based on a Catalytic Triad". Chem. 2 (5): 732—745. 2017. doi:10.1016/j.chempr.2017.04.004.
- ↑ "Catalytic supramolecular self-assembled peptide nanostructures for ester hydrolysis". J. Mater. Chem. B. 4 (26): 4605—4611. 2016. doi:10.1039/c6tb00795c. PMID 32263403.
- ↑ "Protein sectors: evolutionary units of three-dimensional structure". Cell. 138 (4): 774—86. 2009. doi:10.1016/j.cell.2009.07.038. PMID 19703402.
- ↑ "How far divergent evolution goes in proteins". Current Opinion in Structural Biology. 8 (3): 380—387. 1998. doi:10.1016/S0959-440X(98)80073-0. PMID 9666335.
- ↑ "Divergent evolution of enzymatic function: mechanistically diverse superfamilies and functionally distinct suprafamilies". Annu. Rev. Biochem. 70 (1): 209—46. 2001. doi:10.1146/annurev.biochem.70.1.209. PMID 11395407.
- ↑ "Bio-Zombie: the rise of pseudoenzymes in biology". Biochem. Soc. Trans. 45 (2): 537—544. 2017. doi:10.1042/bst20160400. PMID 28408493.
- ↑ "Sinapoyltransferases in the light of molecular evolution". Phytochemistry. 70 (15—16): 1652—62. 2009. doi:10.1016/j.phytochem.2009.07.023. PMID 19695650.
- ↑ "Alpha/beta-hydrolases: A unique structural motif coordinates catalytic acid residue in 40 protein fold families". Proteins. 85 (10): 1845—1855. 2017. doi:10.1002/prot.25338. PMID 28643343.
- ↑ "Glutamine PRPP amidotransferase: snapshots of an enzyme in action". Current Opinion in Structural Biology. 8 (6): 686—94. 1998. doi:10.1016/s0959-440x(98)80087-0. PMID 9914248.
- ↑ "Structure of the allosteric regulatory enzyme of purine biosynthesis". Science. 264 (5164): 1427—33. 1994. Bibcode:1994Sci...264.1427S. doi:10.1126/science.8197456. PMID 8197456.
- ↑ Clans of Mixed (C, S, T) Catalytic Type. www.ebi.ac.uk. MEROPS. Дата обращения: 20 декабря 2018. Архивировано 25 июля 2021 года.
- ↑ 1 2 "Pseudoproteases: mechanisms and function". Biochem. J. 468 (1): 17—24. 2015. doi:10.1042/BJ20141506. PMID 25940733.
- ↑ "Sequence and structural differences between enzyme and nonenzyme homologs". Structure. 10 (10): 1435—51. 2002. doi:10.1016/s0969-2126(02)00861-4. PMID 12377129.
- ↑ "Structure of HBP, a multifunctional protein with a serine proteinase fold". Nat. Struct. Biol. 4 (4): 265—8. 1997. doi:10.1038/nsb0497-265. PMID 9095193.
- ↑ "Rhomboid family pseudoproteases use the ER quality control machinery to regulate intercellular signaling". Cell. 145 (1): 79—91. 2011. doi:10.1016/j.cell.2011.02.047. PMID 21439629.
- ↑ "Inactive rhomboid proteins: New mechanisms with implications in health and disease". Semin. Cell Dev. Biol. 60: 29—37. 2016. doi:10.1016/j.semcdb.2016.06.022. PMID 27378062.
- ↑ "Testins Are Structurally Related to the Mouse Cysteine Proteinase Precursor But Devoid of Any Protease/Anti-Protease Activity". Biochem. Biophys. Res. Commun. 191 (1): 224—231. 1993. doi:10.1006/bbrc.1993.1206. PMID 8447824.
- ↑ "Why Ser and not Thr brokers catalysis in the trypsin fold". Biochemistry. 54 (7): 1457—64. 2015. doi:10.1021/acs.biochem.5b00014. PMID 25664608.