Протеомика: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
Minina (обсуждение | вклад) Нет описания правки |
Minina (обсуждение | вклад) Нет описания правки |
||
| Строка 28: | Строка 28: | ||
{{main|Масс-спектрометрия}} |
{{main|Масс-спектрометрия}} |
||
[[Файл:MALDI TOF EN.png|thumb|300px|Схема масс-спектрометра, использующего ионизацию методом MALDI-TOF. Матрица, содержащая исследуемые молекулы, облучается лазером и ионизируется, ионизируя исследуемые пептиды, которые впоследствии и детектируются]] |
[[Файл:MALDI TOF EN.png|thumb|300px|Схема масс-спектрометра, использующего ионизацию методом MALDI-TOF. Матрица, содержащая исследуемые молекулы, облучается лазером и ионизируется, ионизируя исследуемые пептиды, которые впоследствии и детектируются]] |
||
Масс-спектрометрия включает ряд методов, которые направлены на получение молекулярной массы исследуемых соединений. Она нашла огромное применение и в биологии, в особенности, в протеомике. Сначала белки, находящиеся в образце, ионизируют, потом в условиях вакуума ионы сортируются и детектируются, давая на выходе спектр, который дальше анализируется специальными вычислительными методами. В конечном итоге для каждого иона определяется значение отношения массы к заряду. Если заряд иона равен единице, то отношение численно равно его молекулярной массе. Поначалу использование масс-спектрометрии в биологии было ограничено из-за того, что ионизация была очень жёсткой и приводила к разрушению молекул. В 1980-х годах был разработан метод ионизации молекул лазером при их сокристаллизации со светочувствительным органическим веществом (его называют матрицей). Матрица окружает молекулы исследуемого вещества и под действием лазера ионизирует соседние молекулы. В некоторых условиях ионизацию можно провести без разрушения исследуемых молекул. Этот метод получил название опосредованная матрицей лазерная десорбция-ионизация ({{lang-en|matrix-assisted laser desorption ionisation, MALDI}}). Новый метод ионизации совместили с обычным масс-спектрометрометрическим детектором (времяпролётным, {{lang-en|time-of-flight, TOF}}). В этом детекторе ионы движутся в вакуумной трубке и достигают чувствительной пластины (фотоэлектронного умножителя), которая и является детектором. Время, за которое ион преодолевает длину трубки, обратно пропорционально его массе. В 1990-е и в начале 2000-х метод MALDI-TOF очень активно использовался для исследований белков<ref>{{cite pmid|1789447}}</ref><ref>{{cite |
Масс-спектрометрия включает ряд методов, которые направлены на получение молекулярной массы исследуемых соединений. Она нашла огромное применение и в биологии, в особенности, в протеомике. Сначала белки, находящиеся в образце, ионизируют, потом в условиях вакуума ионы сортируются и детектируются, давая на выходе спектр, который дальше анализируется специальными вычислительными методами. В конечном итоге для каждого иона определяется значение отношения массы к заряду. Если заряд иона равен единице, то отношение численно равно его молекулярной массе. Поначалу использование масс-спектрометрии в биологии было ограничено из-за того, что ионизация была очень жёсткой и приводила к разрушению молекул. В 1980-х годах был разработан метод ионизации молекул лазером при их сокристаллизации со светочувствительным органическим веществом (его называют матрицей). Матрица окружает молекулы исследуемого вещества и под действием лазера ионизирует соседние молекулы. В некоторых условиях ионизацию можно провести без разрушения исследуемых молекул. Этот метод получил название опосредованная матрицей лазерная десорбция-ионизация ({{lang-en|matrix-assisted laser desorption ionisation, MALDI}}). Новый метод ионизации совместили с обычным масс-спектрометрометрическим детектором (времяпролётным, {{lang-en|time-of-flight, TOF}}). В этом детекторе ионы движутся в вакуумной трубке и достигают чувствительной пластины (фотоэлектронного умножителя), которая и является детектором. Время, за которое ион преодолевает длину трубки, обратно пропорционально его массе. В 1990-е и в начале 2000-х метод MALDI-TOF очень активно использовался для исследований белков<ref>{{cite pmid|1789447}}</ref><ref>{{cite doi|10.1016/j.ijms.2014.07.018}}</ref>. |
||
[[Файл:Bottom-up vs top down.svg|thumb|400px|Сравнение принципов протеомики «снизу вверх» и «сверху вниз»]] |
[[Файл:Bottom-up vs top down.svg|thumb|400px|Сравнение принципов протеомики «снизу вверх» и «сверху вниз»]] |
||
| Строка 43: | Строка 43: | ||
Методы масс-спектрометрии могут быть использованы для направленного обнаружения искомых белков, то есть масс-спектрометр можно настроить таким образом, чтобы он видел только нужный пептид. Для этой цели используют прибор с детектором типа тройного квадруполя, то есть три одинаковых масс-спектрометра, последовательно передающие друг другу ионы. Первый масс-спектрометр отфильтровывает интересующий пептид, во втором он фрагментируется, а третий регистрирует от 3 до 5 заранее выбранных фрагментов. Количественный анализ производится на основе интенсивности фрагментов. Этот метод называется мониторинг множественных реакций ({{lang-en|multiple reaction monitoring, MRM}}), или мониторинг выбранных реакций ({{lang-en|selected reaction monitoring, SRM}})<ref>{{cite doi|10.1074/mcp.M500331-MCP200}}</ref>. |
Методы масс-спектрометрии могут быть использованы для направленного обнаружения искомых белков, то есть масс-спектрометр можно настроить таким образом, чтобы он видел только нужный пептид. Для этой цели используют прибор с детектором типа тройного квадруполя, то есть три одинаковых масс-спектрометра, последовательно передающие друг другу ионы. Первый масс-спектрометр отфильтровывает интересующий пептид, во втором он фрагментируется, а третий регистрирует от 3 до 5 заранее выбранных фрагментов. Количественный анализ производится на основе интенсивности фрагментов. Этот метод называется мониторинг множественных реакций ({{lang-en|multiple reaction monitoring, MRM}}), или мониторинг выбранных реакций ({{lang-en|selected reaction monitoring, SRM}})<ref>{{cite doi|10.1074/mcp.M500331-MCP200}}</ref>. |
||
=== Белковые микрочипы === |
|||
== Сравнительная протеомика == |
|||
{{main|Белковый микрочип}} |
|||
| ⚫ | Сравнение протеомов двух организмов (необязательно близкородственных) позволяет выявить как общие для этих двух организмов белки, так и белки, которые обуславливают различия их фенотипов. Такой анализ может давать информацию, полезную для понимания эволюционного процесса<ref>{{cite |
||
Белковые микрочипы разрабатываются для идентификации определённых белков в образце. По аналогии с ДНК-микрочипами, на твёрдую подложку наносятся очень маленькие капли, содержащие антитела. В каждой капле находятся меченные антитела к одному определённому белку, который добавляется на чип в виде флуоресцентно-меченной пробы. После промывки флуоресценция детектируется только в тех каплях, в которых антитела связали исследуемый белок. Вместо антител можно использовать другие молекулы, специфически взаимодействующие с конкретными белками, например, олигонуклеотиды<ref>{{cite pmid|15113093}}</ref>. Белковые микрочипы также можно использовать для обнаружения белок-белковых взаимодействий и определения функций белков. В настоящее время белковые микрочипы автоматизированы. Они обладают высокой чувствительностью и требуют совсем небольшого количества исследуемого белка, благодаря чему отличаются экономичностью<ref>{{cite doi|10.1038/nbt0302-225}}</ref>. |
|||
== Практические приложения == |
|||
С помощью MALDI-TOF можно определять патогенные микроорганизмы с точностью до родов и видов. Интактные бактериальные клетки наносят на металлическую мишень масс-спектрометра, покрывают матрицей, облучают лазером и получают специфичные профили, которые обученный алгоритм распознаёт по характерным массам<ref>{{cite pmid|19095774}}</ref>. |
|||
Исследуется возможность использования протеомики для диагностики раковых заболеваний, а также определения степени злокачественности опухоли. В этом направлении уже достигнуты некоторые успехи. Например, в США разрешено использование разработанного в 2015 году теста Xpresys Lung, который использует таргетную масс-спектрометрию нескольких белков плазмы крови и оценивает степень злокачественности опухолевых узелков в лёгких<ref>{{cite pmid|26376647}}</ref>. |
|||
| ⚫ | Сравнение протеомов двух организмов (необязательно близкородственных) позволяет выявить как общие для этих двух организмов белки, так и белки, которые обуславливают различия их фенотипов. Такой анализ может давать информацию, полезную для понимания эволюционного процесса<ref>{{cite pmid|11385598}}</ref> , а иногда это позволяет определить ранее неизвестные функции белков. Например, при помощи сравнительной протеомики были выявлены белки Nilaparvata lugens, вовлеченные в процессы, связанные с размножением, чья экспрессия изменяется в ответ на обработку инсектицидами<ref>{{cite pmid|21800909}}</ref>. |
||
== Примечания == |
== Примечания == |
||
Версия от 18:29, 30 августа 2018
Эту страницу в данный момент активно редактирует участник Minina. |
Протео́мика (англ. Proteomics) — область молекулярной биологии, посвящённая идентификации и количественному анализу белков (иными словами, высокопроизводительному исследованию белков). Термин «протеомика» был предложен в 1997 году.
Объектом изучения протеомики являются белки, которые экспрессируются в данной клетке, ткани или организме в данный момент времени (то есть протеом). Хотя первые методы протеомики, например, секвенирование белков по Эдману, появились задолго до геномных технологий, действительно высокопроизводительное изучение белков стало возможным только в постгеномную эпоху, то есть при наличии известных нуклеотидных последовательностей геномов разных организмов.
Методы
Количественный анализ, не требующий информации о структурах белков
Количественный анализ белков с ферментативной активностью можно опосредованно проводить через определение активности этих белков. Ещё в начале XX века подобный анализ можно было осуществить с помощью методов спектрофотометрии. При этом количество катализатора оценивается в условных единицах активности. Условные единицы активности до сих пор используют для описания концентрации в крови таких биомаркеров, как аланиниаминотрансфераза и аспартатаминотрансфераза. В 1975 году были изобретены моноклональные антитела, которые быстро нашли применение в исследовании белков. Например, если известен антиген данного антитела, то с помощью антител можно идентифицировать этот антиген в исследуемом образце. В медицине в качестве биомаркеров и сейчас широко используются антитела, антигены которых неизвестны, но которые связывают у больных людей гораздо больше антигена, чем у здоровых. Например, гликопротеин CA-125 использовали как биомаркер рака яичников с 1981 года, когда были получены антитела к нему. Значительно позднее идентифицировали сам белок — муцин 16[1].
Секвенирование по Эдману
В 1950-х годах шведский химик Пер Эдман изобрёл метод определения аминокислотной последовательности белков (секвенирование). Первым этапом секвенирования по Эдману является обработка исследуемого пептида изотиоцианатом фенила, который взаимодействует с аминогруппой, давая фенилтиокарбомоильный радикал. При умеренном закислении раствора он отщепляется, захватывая вместе с собой N-концевую аминокислоту. В результате в раствор выходит тиазолинон с радикалом, специфичным для данной аминокислоты. Это производное анализируют хроматографически, определяя, какая аминокислота была на N-конце, и цикл повторяется. Если исследуемый белок закреплён на твёрдой подложке, то после каждой обработки изотиоцианатом фенила его можно промывать, удаляя тиазолинон с N-концевой кислотой, и начинать новый цикл. Метод Эдмана позволяет с высокой точностью определять последовательность длиной до 30 аминокислотных остатков. Метод также очень чувствителен: он позволяет секвенировать менее 0,1 нмоль пептида с 99% точностью. В 1960-х был создан автоматический секвенатор, реализующий метод Эдмана. Метод Эдмана изредка используют и сейчас при исследовании организмов, геномные последовательности которых неизвестны[2][3][4].
Двумерный гель-электрофорез


В 1970—1980-х годах достигли расцвета методы выделения и очистки белков. Эти методы сочетали принципы хроматографии, электрофореза и центрифугирования; многие из них давно вышли из употребления, но некоторые используются и по сей день. В 1970-м году швейцарский учёный Ульрих Лэммли предложил метод разделения белков при помощи электрофореза в денатурирующих условиях. Сначала белки подвергали жёсткой денатурации под действием додецилсульфата натрия (англ. sodium dodecyl sulphate, SDS), который в виде слоя покрывал каждую белковую молекулу. Чем больше белок, тем больше SDS связывалось с ним и тем больший отрицательный заряд приобретал их комплекс. Поэтому при нанесении образцов в полиакриламидный гель они начинали двигаться по действием электрического поля, причём скорость движения белковых молекул зависит от их массы (более лёгкие белки перемещаются по гелю быстрее). Метод хорошо подходит для разделения белков с массой от 5 до 250 кДа[5].

Метод Лэммли получил дальнейшее развитие. В 1975 году Патрик О’Фарелл и Йоахим Клозе независимо предложили принцип так называемого двумерного электрофореза: перед разделением по массе с помощью SDS белки предварительно разделяются согласно их изоэлектрической точке. Сначала белки вносят в стеклянную трубку, заполненную особыми полимерами, которые создают в ней неподвижный градиент pH. Белки распределяются по трубке, занимая места, pH которых равен их изоэлектрической точке. Далее содержимое трубки выдавливают и приплавляют в гелю для обычного электрофореза по Лэммли. Таким образом, сначала белки делятся по изоэлектрической точке, а потом по массе. В результате двумерного электрофореза каждому белку соответствует не полоса, как при обычном электрофорезе, а сфокусированное округлое пятно, размер и интенсивность окрашивания которого соответствуют концентрации белка. С помощью двумерного электрофореза можно разделять не только различные белки, но и изоформы одного и того же белка, а также формы белка с разными посттрансляционными модификациями. Были предложены различные усовершенствования методики двумерного электрофореза, некоторые этапы, а также обработка отсканированных гелей, были автоматизированы. По сути, двумерный электрофорез — единственный способ наглядного представления протеома[6][7][8].
Вестерн-блоттинг

Дальнейшее развитие белковый гель-электрофорез получил в виде нового метода для идентификации белков в пробе — вестерн-блоттинг. Сначала белки разделяют в полиакриламидном геле, после чего переносят их на особую мембрану, причём полосы располагаются на мембране так же, как на геле. Далее мембрану обрабатывают последовательно двумя видами антител и выявляют полосы, с которыми связались антитела и, следовательно, соответствующие искомому белку.
Масс-спектрометрия

Масс-спектрометрия включает ряд методов, которые направлены на получение молекулярной массы исследуемых соединений. Она нашла огромное применение и в биологии, в особенности, в протеомике. Сначала белки, находящиеся в образце, ионизируют, потом в условиях вакуума ионы сортируются и детектируются, давая на выходе спектр, который дальше анализируется специальными вычислительными методами. В конечном итоге для каждого иона определяется значение отношения массы к заряду. Если заряд иона равен единице, то отношение численно равно его молекулярной массе. Поначалу использование масс-спектрометрии в биологии было ограничено из-за того, что ионизация была очень жёсткой и приводила к разрушению молекул. В 1980-х годах был разработан метод ионизации молекул лазером при их сокристаллизации со светочувствительным органическим веществом (его называют матрицей). Матрица окружает молекулы исследуемого вещества и под действием лазера ионизирует соседние молекулы. В некоторых условиях ионизацию можно провести без разрушения исследуемых молекул. Этот метод получил название опосредованная матрицей лазерная десорбция-ионизация (англ. matrix-assisted laser desorption ionisation, MALDI). Новый метод ионизации совместили с обычным масс-спектрометрометрическим детектором (времяпролётным, англ. time-of-flight, TOF). В этом детекторе ионы движутся в вакуумной трубке и достигают чувствительной пластины (фотоэлектронного умножителя), которая и является детектором. Время, за которое ион преодолевает длину трубки, обратно пропорционально его массе. В 1990-е и в начале 2000-х метод MALDI-TOF очень активно использовался для исследований белков[9][10].

Из-за особенностей изотопного разделения пики в спектрах больших белков чрезвычайно сложно анализировать. По этой причине перед исследованием их с помощью фермента трипсина разрушают на пептиды массой 500—2500 Да, и затем по данным для пептидов восстанавливают информацию об исходном белке подобно тому, как при секвенировании нуклеиновых кислот нового поколения исходные последовательности собираются из коротких прочтений. Этот подход называется «протеомикой снизу вверх» (англ. bottom-up). Процесс сборки небезошибочен и приводит к большим потерям информации, поэтому в некоторых случаях исследуются целые белки без расщепления с помощью мощных детекторов сверхвысокого разрешения («протеомика сверху вниз», англ. top-down)[11].
Набор молекулярных масс пептидов, которые были получены при обработке белка трипсином, уникален для каждого белка. Это связано в основном с высокой специфичностью трипсина, который вносит разрез только по остатками лизина и аргинина. Сравнивания полученную картину молекулярных масс пептидов для исследуемого белка с пептидными картами белков из баз данных, можно установить, какой именно белок исследовался. Этот подход получил название пептидной дактилоскопии[12]. Поскольку полного соответствия экспериментального распределения масс пептидов и эталонными пептидными картами достичь невозможно, была введена количественная оценка (score) вероятности того, что экспериментальная пептидная карта соответствует данной теоретической. Для пептидной дактилоскопии были разработаны специальные программы, например, MOWSE[13].
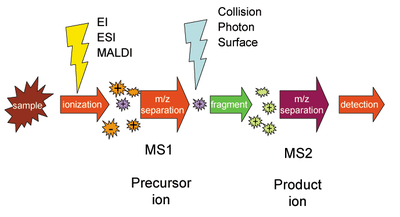
Вместо фрагментации трипсином перед установкой образцов в масс-спектрометр фрагментацию белков на фрагменты можно осуществлять в самом масс-спектрометре при помощи, например, столкновения с молекулами инертных газов. При этом каждый пептид характеризуется массой иона-предшественника и набором масс ионов-фрагментов. Массы фрагментов можно измерить и по ним восстановить информацию об исходном белке, так как молекулярные массы фрагментов можно найти исходя из последовательности пептида. Такой подход получил название тандемной масс-спектрометрии (MS-MS). Как и при пептидной дактилоскопии, в тандемной масс-спектрометрии имеет место вероятностная оценка того, что пептидная карта исследуемого белка соответствует одной из теоретических. В 2007 году для анализа данных тандемной масс-спектрометрии был предложен подход target-decoy. Суть этого подхода заключается в том, что при анализе данных к целевым теоретическим пептидам (target) стали добавлять равное количество бессмысленных, фальшивых (decoy) пептидов. Этот подход позволяет оценить качество анализа. Если анализ в качестве лучших соответствий выдаёт соответствие экспериментального белка с заведомо фальшивым, то он даёт ложноположительный результат, а подход target-decoy позволяет оценить долю ложноположительных результатов[14].

В качестве альтернативны MALDI ионизацию пептидов перед масс-спектрометрией можно осуществлять с помощью метода ионизации электрораспылением, или ионизации электроспреем (англ. electrospray ionisation, ESI). Жидкость, содержащая исследуемые белки, помещается в конический капилляр, и, когда она выходит из капилляра, к ней прилагается сильное напряжение. В результате жидкость превращается в аэрозоль, и при испарении частиц их аэрозоля в потоке инертного газа заряд может переходить на растворённые в аэрозоле биомолекулы, в том числе белки. При таком способе ионизации биомолекулы не разрушаются. Ионизацию электроспреем можно легко совместить с высокоэффективной жидкостной хроматографией: поток хроматографической фазы с колонки можно направить прямо в капилляр для электрораспыления. Таким образом, масс-спектрометр будет определять массы разделяемых в аналитической колонке молекул. Этот метод обозначают аббревиатурой LC-MS (от англ. Шаблон:Liquid chromatography-mass spectrometry)[15]. Идентификация белков в сложном растворе при помощи комбинации масс-спектрометрии и высокоэффективной жидкостной хроматографии получила название протеомики-дробовика, или скорострельной протеомики (англ. shotgun proteomics)[16].
Методы масс-спектрометрии могут быть использованы для направленного обнаружения искомых белков, то есть масс-спектрометр можно настроить таким образом, чтобы он видел только нужный пептид. Для этой цели используют прибор с детектором типа тройного квадруполя, то есть три одинаковых масс-спектрометра, последовательно передающие друг другу ионы. Первый масс-спектрометр отфильтровывает интересующий пептид, во втором он фрагментируется, а третий регистрирует от 3 до 5 заранее выбранных фрагментов. Количественный анализ производится на основе интенсивности фрагментов. Этот метод называется мониторинг множественных реакций (англ. multiple reaction monitoring, MRM), или мониторинг выбранных реакций (англ. selected reaction monitoring, SRM)[17].
Белковые микрочипы
Белковые микрочипы разрабатываются для идентификации определённых белков в образце. По аналогии с ДНК-микрочипами, на твёрдую подложку наносятся очень маленькие капли, содержащие антитела. В каждой капле находятся меченные антитела к одному определённому белку, который добавляется на чип в виде флуоресцентно-меченной пробы. После промывки флуоресценция детектируется только в тех каплях, в которых антитела связали исследуемый белок. Вместо антител можно использовать другие молекулы, специфически взаимодействующие с конкретными белками, например, олигонуклеотиды[18]. Белковые микрочипы также можно использовать для обнаружения белок-белковых взаимодействий и определения функций белков. В настоящее время белковые микрочипы автоматизированы. Они обладают высокой чувствительностью и требуют совсем небольшого количества исследуемого белка, благодаря чему отличаются экономичностью[19].
Практические приложения
С помощью MALDI-TOF можно определять патогенные микроорганизмы с точностью до родов и видов. Интактные бактериальные клетки наносят на металлическую мишень масс-спектрометра, покрывают матрицей, облучают лазером и получают специфичные профили, которые обученный алгоритм распознаёт по характерным массам[20].
Исследуется возможность использования протеомики для диагностики раковых заболеваний, а также определения степени злокачественности опухоли. В этом направлении уже достигнуты некоторые успехи. Например, в США разрешено использование разработанного в 2015 году теста Xpresys Lung, который использует таргетную масс-спектрометрию нескольких белков плазмы крови и оценивает степень злокачественности опухолевых узелков в лёгких[21].
Сравнение протеомов двух организмов (необязательно близкородственных) позволяет выявить как общие для этих двух организмов белки, так и белки, которые обуславливают различия их фенотипов. Такой анализ может давать информацию, полезную для понимания эволюционного процесса[22] , а иногда это позволяет определить ранее неизвестные функции белков. Например, при помощи сравнительной протеомики были выявлены белки Nilaparvata lugens, вовлеченные в процессы, связанные с размножением, чья экспрессия изменяется в ответ на обработку инсектицидами[23].
Примечания
- ↑ Bast R C, Feeney M, Lazarus H, Nadler L M, Colvin R B, Knapp R C. Reactivity of a monoclonal antibody with human ovarian carcinoma. (англ.) // Journal of Clinical Investigation. — 1981. — 1 November (vol. 68, no. 5). — P. 1331—1337. — ISSN 0021-9738. — doi:10.1172/JCI110380.
- ↑ Edman Pehr, Högfeldt Erik, Sillén Lars Gunnar, Kinell Per-Olof. Method for Determination of the Amino Acid Sequence in Peptides. (англ.) // Acta Chemica Scandinavica. — 1950. — Vol. 4. — P. 283—293. — ISSN 0904-213X. — doi:10.3891/acta.chem.scand.04-0283.
- ↑ Edman P., Begg G. A protein sequenator. (англ.) // European Journal Of Biochemistry. — 1967. — March (vol. 1, no. 1). — P. 80—91. — PMID 6059350.
- ↑ Niall Hugh D. [36 Automated edman degradation: The protein sequenator] (англ.) // Methods in Enzymology. — 1973. — P. 942—1010. — ISBN 9780121818906. — ISSN 0076-6879. — doi:10.1016/S0076-6879(73)27039-8.
- ↑ LAEMMLI U. K. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4 (англ.) // Nature. — 1970. — August (vol. 227, no. 5259). — P. 680—685. — ISSN 0028-0836. — doi:10.1038/227680a0.
- ↑ O'Farrell P. H. High resolution two-dimensional electrophoresis of proteins. (англ.) // The Journal Of Biological Chemistry. — 1975. — 25 May (vol. 250, no. 10). — P. 4007—4021. — PMID 236308.
- ↑ Klose J. Protein mapping by combined isoelectric focusing and electrophoresis of mouse tissues. A novel approach to testing for induced point mutations in mammals. (англ.) // Humangenetik. — 1975. — Vol. 26, no. 3. — P. 231—243. — PMID 1093965.
- ↑ Bandow J. E., Baker J. D., Berth M., Painter C., Sepulveda O. J., Clark K. A., Kilty I., VanBogelen R. A. Improved image analysis workflow for 2-D gels enables large-scale 2-D gel-based proteomics studies--COPD biomarker discovery study. (англ.) // Proteomics. — 2008. — August (vol. 8, no. 15). — P. 3030—3041. — doi:10.1002/pmic.200701184. — PMID 18618493.
- ↑ Hillenkamp F., Karas M., Beavis R. C., Chait B. T. Matrix-assisted laser desorption/ionization mass spectrometry of biopolymers. (англ.) // Analytical Chemistry. — 1991. — 15 December (vol. 63, no. 24). — P. 1193—1203. — PMID 1789447.
- ↑ McEwen Charles N., Larsen Barbara S. Fifty years of desorption ionization of nonvolatile compounds (англ.) // International Journal of Mass Spectrometry. — 2015. — February (vol. 377). — P. 515—531. — ISSN 1387-3806. — doi:10.1016/j.ijms.2014.07.018.
- ↑ Durbin K. R., Fornelli L., Fellers R. T., Doubleday P. F., Narita M., Kelleher N. L. Quantitation and Identification of Thousands of Human Proteoforms below 30 kDa. (англ.) // Journal Of Proteome Research. — 2016. — 4 March (vol. 15, no. 3). — P. 976—982. — doi:10.1021/acs.jproteome.5b00997. — PMID 26795204.
- ↑ Shevchenko A., Jensen O. N., Podtelejnikov A. V., Sagliocco F., Wilm M., Vorm O., Mortensen P., Shevchenko A., Boucherie H., Mann M. Linking genome and proteome by mass spectrometry: large-scale identification of yeast proteins from two dimensional gels. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1996. — 10 December (vol. 93, no. 25). — P. 14440—14445. — PMID 8962070.
- ↑ Pappin D. J., Hojrup P., Bleasby A. J. Rapid identification of proteins by peptide-mass fingerprinting. (англ.) // Current Biology : CB. — 1993. — 1 June (vol. 3, no. 6). — P. 327—332. — PMID 15335725.
- ↑ Elias Joshua E, Gygi Steven P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry (англ.) // Nature Methods. — 2007. — March (vol. 4, no. 3). — P. 207—214. — ISSN 1548-7091. — doi:10.1038/nmeth1019.
- ↑ Pitt J. J. Principles and applications of liquid chromatography-mass spectrometry in clinical biochemistry. (англ.) // The Clinical Biochemist. Reviews. — 2009. — February (vol. 30, no. 1). — P. 19—34. — PMID 19224008.
- ↑ Alves P., Arnold R. J., Novotny M. V., Radivojac P., Reilly J. P., Tang H. Advancement in protein inference from shotgun proteomics using peptide detectability. (англ.) // Pacific Symposium On Biocomputing. Pacific Symposium On Biocomputing. — 2007. — P. 409—420. — PMID 17990506.
- ↑ Anderson Leigh, Hunter Christie L. Quantitative Mass Spectrometric Multiple Reaction Monitoring Assays for Major Plasma Proteins (англ.) // Molecular & Cellular Proteomics. — 2005. — 6 December (vol. 5, no. 4). — P. 573—588. — ISSN 1535-9476. — doi:10.1074/mcp.M500331-MCP200.
- ↑ Weston A. D., Hood L. Systems biology, proteomics, and the future of health care: toward predictive, preventative, and personalized medicine. (англ.) // Journal Of Proteome Research. — 2004. — March (vol. 3, no. 2). — P. 179—196. — PMID 15113093.
- ↑ Mitchell Peter. A perspective on protein microarrays (англ.) // Nature Biotechnology. — 2002. — March (vol. 20, no. 3). — P. 225—229. — ISSN 1087-0156. — doi:10.1038/nbt0302-225.
- ↑ Ilina E. N., Borovskaya A. D., Malakhova M. M., Vereshchagin V. A., Kubanova A. A., Kruglov A. N., Svistunova T. S., Gazarian A. O., Maier T., Kostrzewa M., Govorun V. M. Direct bacterial profiling by matrix-assisted laser desorption-ionization time-of-flight mass spectrometry for identification of pathogenic Neisseria. (англ.) // The Journal Of Molecular Diagnostics : JMD. — 2009. — January (vol. 11, no. 1). — P. 75—86. — doi:10.2353/jmoldx.2009.080079. — PMID 19095774.
- ↑ Vachani A., Hammoud Z., Springmeyer S., Cohen N., Nguyen D., Williamson C., Starnes S., Hunsucker S., Law S., Li X. J., Porter A., Kearney P. Clinical Utility of a Plasma Protein Classifier for Indeterminate Lung Nodules. (англ.) // Lung. — 2015. — December (vol. 193, no. 6). — P. 1023—1027. — doi:10.1007/s00408-015-9800-0. — PMID 26376647.
- ↑ Gagneux P., Amess B., Diaz S., Moore S., Patel T., Dillmann W., Parekh R., Varki A. Proteomic comparison of human and great ape blood plasma reveals conserved glycosylation and differences in thyroid hormone metabolism. (англ.) // American Journal Of Physical Anthropology. — 2001. — June (vol. 115, no. 2). — P. 99—109. — doi:10.1002/ajpa.1061. — PMID 11385598.
- ↑ Ge L. Q., Cheng Y., Wu J. C., Jahn G. C. Proteomic analysis of insecticide triazophos-induced mating-responsive proteins of Nilaparvata lugens Stål (Hemiptera: Delphacidae). (англ.) // Journal Of Proteome Research. — 2011. — 7 October (vol. 10, no. 10). — P. 4597—4612. — doi:10.1021/pr200414g. — PMID 21800909.