Комплекс распознавания Ori
| Origin recognition complex subunit 2 | |
|---|---|
| Идентификаторы | |
| Символ | ORC2 |
| Pfam | PF04084 |
| InterPro | IPR007220 |
| Доступные структуры белков | |
| Pfam | структуры |
| PDB | RCSB PDB; PDBe; PDBj |
| PDBsum | 3D-модель |
| Origin recognition complex (ORC) subunit 3 N-terminus | |
|---|---|
| Идентификаторы | |
| Символ | ORC3_N |
| Pfam | PF07034 |
| InterPro | IPR010748 |
| Доступные структуры белков | |
| Pfam | структуры |
| PDB | RCSB PDB; PDBe; PDBj |
| PDBsum | 3D-модель |
| Origin recognition complex subunit 6 (ORC6) | |
|---|---|
| Идентификаторы | |
| Символ | ORC6 |
| Pfam | PF05460 |
| InterPro | IPR008721 |
| Доступные структуры белков | |
| Pfam | структуры |
| PDB | RCSB PDB; PDBe; PDBj |
| PDBsum | 3D-модель |
Комплекс распознавания Ori (англ. Origin recognition complex, сокр. ORC) — представляет собой мультисубъединичный ДНК-связывающий комплекс (включает 6 субъединиц), который во всех эукариотах и археях АТФ-зависимым образом связывается с точками начала репликации (англ. origin, сокр. ori). Субъединицы данного комплекса кодируются соответствующими генами ORC1, ORC2, ORC3, ORC4, ORC5 и ORC6[1][2][3]. ORC является центральным компонентом для репликации эукариотической ДНК и остаётся связанным с хроматином у точек начала репликации на протяжении всего клеточного цикла[4].
ORC направляет репликацию ДНК по всему геному и необходим для её инициации[5][6][7]. ORC и Noc3p, связанные у начала репликации, служат основой для сборки пререпликационного комплекса (pre-RC), в который входят Cdc6, Tah11 (он же Cdt1) и комплекс Mcm2-Mcm7 (белки поддержания минихромосом)[8][9][10][11]. Сборка пре-РК в G1-фазе необходима для лицензирования репликации хромосом перед синтезом ДНК в S-фазе[12][13][14]. Фосфорилирование субъединиц Orc2, Orc6, Cdc6 и MCM, циклин-зависимой протеинкиназой Cdc28 регулирует инициацию репликации ДНК, включая блокирование реинициации (повторной инициации) в G2/M-фазах[4][15][16][17].
ORC присутствует на протяжении всего клеточного цикла, связываясь с точками начала репликации, но активен только в конце митоза и начале G1-фазы.
У дрожжей ORC также играет роль в установлении сайленсинга в локусах спаривания Hidden MAT Left (HML) и Hidden MAT Right (HMR)[5][6][7]. ORC участвует в сборке транскрипционно «молчащего» хроматина в HML и HMR, путём рекрутирования сайленсинг-белка Sir1 к сайленсерам HML и HMR[7][18][19].
И Orc1, и Orc5 связывают молекулы ATP, но только Orc1 обладает АТФазной активностью[20]. Связывание молекул ATP субъединицей Orc1 требуется для связывания всего комплекса ORC с ДНК и необходимо для жизнеспособности клеток[11]. АТФазная активность Orc1 участвует в формировании пре-РК[21][22][23]. Связывание молекул ATP субъединицей Orc5 имеет решающее значение для стабильности ORC в целом. Только субъединицы Orc1-5 требуются для связывания ori; Orc6 необходим для поддержания пре-РК после формирования[24]. Взаимодействия внутри ORC предполагают, что Orc2-3-6 могут образовывать кóровый (сердцевинный) комплекс[4]. В сообщении от 2020 года говорится, что ORC почкующихся дрожжей димеризуется в зависимости от клеточного цикла для контроля разрешающего фактора (лицензирование)[25][26].
Белки, входящие в комплекс[править | править код]
В состав ORC (различных организмов) входят следующие белки:
| S. cerevisiae | S. pombe | D. melanogaster | Позвоночные |
|---|---|---|---|
| ORC 1-6 | ORC 1-6 | ORC 1-6 | ORC 1-6 |
| Cdc6 | Cdc18 | Cdc6 | Cdc6 |
| Cdt1/Tah11/Sid2 | Cdt1 | DUP | Cdt1/RLF-B |
| Mcm2 | Mcm2/Cdc19/Nda1 | Mcm2 | Mcm2 |
| Mcm3 | Mcm3 | Mcm3 | Mcm3 |
| Cdc54/Mcm4 | Cdc21 | DPA | Mcm4 |
| Cdc46/Mcm5 | Mcm5/Nda4 | Mcm5 | Mcm5 |
| Mcm6 | Mcm6/Mis5 | Mcm6 | Mcm6 |
| Cdc47/Mcm7 | Mcm7 | Mcm7 | Mcm7 |
Археи имеют более простую (примитивную) форму ORC, MCM, и, как следствие, объединённый пре-РК. Вместо того чтобы использовать шесть разных белков Mcm для формирования псевдосимметричного гетерогексамера, все шесть субъединиц в MCM у архей одинаковы. Обычно они имеют несколько белков, гомологичных как Cdc6, так и Orc1, некоторые из которых выполняют функции обоих. В отличие от эукариотических ORC, они не всегда образуют комплекс. Фактически, они имеют дивергентные комплексные структуры во время их образования. Sulfolobus islandicus также использует гомолог Cdt1 для распознавания одного из своих точек начала репликации[28].
Автономно реплицирующиеся последовательности[править | править код]
У почкующихся дрожжей[править | править код]
Автономно реплицирующиеся последовательности (от англ. Autonomously Replicating Sequences, сокр. ARS), впервые обнаруженные у почкующихся дрожжей, являются неотъемлемой частью и успешны интегрированы с ORC. Эти последовательности имеют длину 100-200 п.н., которые облегчают репликативную активность в S-фазе. ARS могут быть размещены в любой новой части хромосом почкующихся дрожжей и будут способствовать репликации из этих мест. Высококонсервативная последовательность длиной 11 п.н. (известная как A-элемент) считается необходимой для функционирования origin в почкующихся дрожжах[27]. Комплекс распознавания Ori был первоначально идентифицирован по его способности связываться с A-элементом ARS в почкующихся дрожжах.
У животных[править | править код]
Животные клетки содержат гораздо более «загадочную» версию ARS, в которой пока не обнаружено ни одной консервативной последовательности. Здесь точки начала репликации собираются в пучки, называемые кластерами репликонов. Длина репликонов каждого кластера одинакова, но отдельные кластеры имеют репликоны разной длины. Все эти репликоны имеют сходные оснóвные остатки, с которыми связывается ORC, что во многом имитирует консервативный A-элемент длиной 11 п.н.. Все эти кластеры одновременно активируются в S-фазе[27].
Роль в сборке пре-РК[править | править код]
Комплекс распознавания Ori необходим для загрузки комплексов MCM (Pre-RC) на молекулы ДНК. Этот процесс непосредственно зависит от ORC, Noc3, Cdc6 и Cdt1, включая несколько событий рекрутирования, под контролем молекул ATP. Во-первых, ORC, Noc3p и Cdc6 образуют комплекс на исходной ДНК (отмеченный областями типа ARS). Новые комплексы ORC/Noc3/Cdc6 затем рекрутируют на этот сайт молекулы Cdt1/Mcm2-7. Как только образуется данный массивный комплекс ORC/Noc3/Cdc6/Cdt1/Mcm2-7, молекулы ORC/Noc3/Cdc6/Cdt1 работают вместе, загружая Mcm2-7 на молекулу ДНК, путём гидролиза ATP с помощью Cdc6. Фосфорилирующая активность Cdc6 зависит как от ORC, так и от исходной ДНК. Это приводит к снижению стабильности Cdt1 на ДНК и отсоединению комплекса, что приводит к рекрутированию комплекса Mcm2-7 на молекулу ДНК[29][27][23][11]. Структуры ORC, MCM, а также промежуточного комплекса OCCM были хорошо изучены[30].
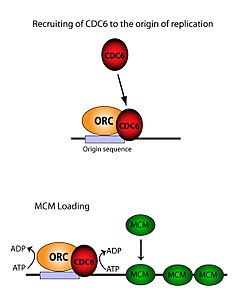

Активность связывания origin[править | править код]
Хотя ORC состоит из шести отдельных субъединиц, только одна из них оказалась наиболее значимой — ORC1. Исследования in vivo показали, что Lys-263 и Arg-367 являются оснóвными остатками, отвечающими за точную сборку ORC. Эти молекулы представляют собой вышеупомянутый ARS[31]. ORC1 взаимодействует с ATP и упомянутыми оснóвными остатками, чтобы связывать весь ORC с исходной ДНК. Установлено, что связывание происходит задолго до репликации и что сам ORC уже связан с origin ДНК к моменту загрузки Mcm2-7[11]. Когда Mcm2-7 впервые рекрутируется, он полностью окружает молекулу ДНК, и активность хеликазы ингибируется. В S-фазе комплекс Mcm2-7 взаимодействует с кофакторами хеликазы Cdc45 и GINS, чтобы отделить одну нить ДНК, раскрутить origin и начать репликацию вниз по хромосоме. Для того чтобы репликация была двунаправленной, этот процесс происходит дважды в origin. Оба события сборки опосредуются одним ORC посредством идентичного процесса, что и первый[32].
Примечания[править | править код]
- ↑ MeSH Origin+Recognition+Complex
- ↑ Dutta A, Bell SP (1997). "Initiation of DNA replication in eukaryotic cells". Annual Review of Cell and Developmental Biology. 13: 293—332. doi:10.1146/annurev.cellbio.13.1.293. PMID 9442876.
- ↑ Multiple functions of the origin recognition complex. — 2007. — Vol. 256. — P. 69–109. — ISBN 9780123737007. — doi:10.1016/S0074-7696(07)56003-1.
- ↑ 1 2 3 Matsuda K, Makise M, Sueyasu Y, Takehara M, Asano T, Mizushima T (December 2007). "Yeast two-hybrid analysis of the origin recognition complex of Saccharomyces cerevisiae: interaction between subunits and identification of binding proteins". FEMS Yeast Research. 7 (8): 1263—9. doi:10.1111/j.1567-1364.2007.00298.x. PMID 17825065.
- ↑ 1 2 Bell SP, Stillman B (May 1992). "ATP-dependent recognition of eukaryotic origins of DNA replication by a multiprotein complex". Nature. 357 (6374): 128—34. Bibcode:1992Natur.357..128B. doi:10.1038/357128a0. PMID 1579162. S2CID 4346767.
- ↑ 1 2 Bell SP, Mitchell J, Leber J, Kobayashi R, Stillman B (November 1995). "The multidomain structure of Orc1p reveals similarity to regulators of DNA replication and transcriptional silencing". Cell. 83 (4): 563—8. doi:10.1016/0092-8674(95)90096-9. PMID 7585959.
- ↑ 1 2 3 Gibson DG, Bell SP, Aparicio OM (June 2006). "Cell cycle execution point analysis of ORC function and characterization of the checkpoint response to ORC inactivation in Saccharomyces cerevisiae". Genes to Cells. 11 (6): 557—73. doi:10.1111/j.1365-2443.2006.00967.x. PMID 16716188. S2CID 22439595.
- ↑ Zhang Y, Yu Z, Fu X, Liang C (June 2002). "Noc3p, a bHLH Protein, Plays an Integral Role in the Initiation of DNA Replication in Budding Yeast". Cell. 109 (7): 849—860. doi:10.1016/s0092-8674(02)00805-x. PMID 12110182.
- ↑ Rao H, Stillman B (March 1995). "The origin recognition complex interacts with a bipartite DNA binding site within yeast replicators". Proceedings of the National Academy of Sciences of the United States of America. 92 (6): 2224—8. Bibcode:1995PNAS...92.2224R. doi:10.1073/pnas.92.6.2224. PMC 42456. PMID 7892251.
- ↑ Rowley A, Cocker JH, Harwood J, Diffley JF (June 1995). "Initiation complex assembly at budding yeast replication origins begins with the recognition of a bipartite sequence by limiting amounts of the initiator, ORC". The EMBO Journal. 14 (11): 2631—41. doi:10.1002/j.1460-2075.1995.tb07261.x. PMC 398377. PMID 7781615.
- ↑ 1 2 3 4 Speck C, Chen Z, Li H, Stillman B (November 2005). "ATPase-dependent cooperative binding of ORC and Cdc6 to origin DNA". Nature Structural & Molecular Biology. 12 (11): 965—71. doi:10.1038/nsmb1002. PMC 2952294. PMID 16228006.
- ↑ Kelly TJ, Brown GW (2000). "Regulation of chromosome replication". Annual Review of Biochemistry. 69: 829—80. doi:10.1146/annurev.biochem.69.1.829. PMID 10966477.
- ↑ Bell SP, Dutta A (2002). "DNA replication in eukaryotic cells". Annual Review of Biochemistry. 71: 333—74. doi:10.1146/annurev.biochem.71.110601.135425. PMID 12045100.
- ↑ Stillman B (February 2005). "Origin recognition and the chromosome cycle". FEBS Letters. 579 (4): 877—84. doi:10.1016/j.febslet.2004.12.011. PMID 15680967. S2CID 33220937.
- ↑ Weinreich M, Liang C, Chen HH, Stillman B (September 2001). "Binding of cyclin-dependent kinases to ORC and Cdc6p regulates the chromosome replication cycle". Proceedings of the National Academy of Sciences of the United States of America. 98 (20): 11211—7. doi:10.1073/pnas.201387198. PMC 58709. PMID 11572976.
- ↑ Nguyen VQ, Co C, Li JJ (June 2001). "Cyclin-dependent kinases prevent DNA re-replication through multiple mechanisms". Nature. 411 (6841): 1068—73. Bibcode:2001Natur.411.1068N. doi:10.1038/35082600. PMID 11429609. S2CID 4393812.
- ↑ Archambault V, Ikui AE, Drapkin BJ, Cross FR (August 2005). "Disruption of mechanisms that prevent rereplication triggers a DNA damage response". Molecular and Cellular Biology. 25 (15): 6707—21. doi:10.1128/MCB.25.15.6707-6721.2005. PMC 1190345. PMID 16024805.
- ↑ Triolo T, Sternglanz R (May 1996). "Role of interactions between the origin recognition complex and SIR1 in transcriptional silencing". Nature. 381 (6579): 251—3. Bibcode:1996Natur.381..251T. doi:10.1038/381251a0. PMID 8622770. S2CID 4309206.
- ↑ Fox CA, Ehrenhofer-Murray AE, Loo S, Rine J (June 1997). "The origin recognition complex, SIR1, and the S phase requirement for silencing". Science. 276 (5318): 1547—51. doi:10.1126/science.276.5318.1547. PMID 9171055.
- ↑ Klemm RD, Austin RJ, Bell SP (February 1997). "Coordinate binding of ATP and origin DNA regulates the ATPase activity of the origin recognition complex". Cell. 88 (4): 493—502. doi:10.1016/S0092-8674(00)81889-9. PMID 9038340.
- ↑ Klemm RD, Bell SP (July 2001). "ATP bound to the origin recognition complex is important for preRC formation". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8361—7. Bibcode:2001PNAS...98.8361K. doi:10.1073/pnas.131006898. PMC 37444. PMID 11459976.
- ↑ Bowers JL, Randell JC, Chen S, Bell SP (December 2004). "ATP hydrolysis by ORC catalyzes reiterative Mcm2-7 assembly at a defined origin of replication". Molecular Cell. 16 (6): 967—78. doi:10.1016/j.molcel.2004.11.038. PMID 15610739.
- ↑ 1 2 Randell JC, Bowers JL, Rodríguez HK, Bell SP (January 2006). "Sequential ATP hydrolysis by Cdc6 and ORC directs loading of the Mcm2-7 helicase". Molecular Cell. 21 (1): 29—39. doi:10.1016/j.molcel.2005.11.023. PMID 16387651.
- ↑ Semple JW, Da-Silva LF, Jervis EJ, Ah-Kee J, Al-Attar H, Kummer L, et al. (November 2006). "An essential role for Orc6 in DNA replication through maintenance of pre-replicative complexes". The EMBO Journal. 25 (21): 5150—8. doi:10.1038/sj.emboj.7601391. PMC 1630405. PMID 17053779.
- ↑ Yin YC, Prasanth SG (July 2021). "Replication initiation: Implications in genome integrity". DNA Repair. 103: 103131. doi:10.1016/j.dnarep.2021.103131. PMC 8296962. PMID 33992866.
- ↑ Amin A, Wu, R, Cheung MH, Scott JF, Wang Z, Zhou Z, Liu C, Zhu G, Wong KC, Yu Z, Liang C (March 2020). "An Essential and Cell-Cycle-Dependent ORC Dimerization Cycle Regulates Eukaryotic Chromosomal DNA Replication". Cell Reports. 30 (10): 3323—3338.e6. doi:10.1016/j.celrep.2020.02.046. PMID 32160540.
- ↑ 1 2 3 4 Morgan, David. The Cell Cycle: Principles of Control. — Primers in Biology, 2007. — P. 62–75. — ISBN 978-0878935086.
- ↑ Ausiannikava D, Allers T (January 2017). "Diversity of DNA Replication in the Archaea". Genes. 8 (2): 56. doi:10.3390/genes8020056. PMC 5333045. PMID 28146124.
- ↑ Fernández-Cid A, Riera A, Tognetti S, Herrera MC, Samel S, Evrin C, et al. (May 2013). "An ORC/Cdc6/MCM2-7 complex is formed in a multistep reaction to serve as a platform for MCM double-hexamer assembly". Molecular Cell. 50 (4): 577—88. doi:10.1016/j.molcel.2013.03.026. hdl:10044/1/19289. PMID 23603117.
- ↑ Yuan Z, Riera A, Bai L, Sun J, Nandi S, Spanos C, et al. (March 2017). "Structural basis of Mcm2-7 replicative helicase loading by ORC-Cdc6 and Cdt1". Nature Structural & Molecular Biology. 24 (3): 316—324. doi:10.1038/nsmb.3372. PMC 5503505. PMID 28191893.
- ↑ Kawakami H, Ohashi E, Kanamoto S, Tsurimoto T, Katayama T (October 2015). "Specific binding of eukaryotic ORC to DNA replication origins depends on highly conserved basic residues". Scientific Reports. 5: 14929. Bibcode:2015NatSR...514929K. doi:10.1038/srep14929. PMC 4601075. PMID 26456755.
- ↑ Chistol G, Walter JC (April 2015). "Single-Molecule Visualization of MCM2-7 DNA Loading: Seeing Is Believing". Cell. 161 (3): 429—430. doi:10.1016/j.cell.2015.04.006. PMID 25910200.