Переворот оснований ДНК
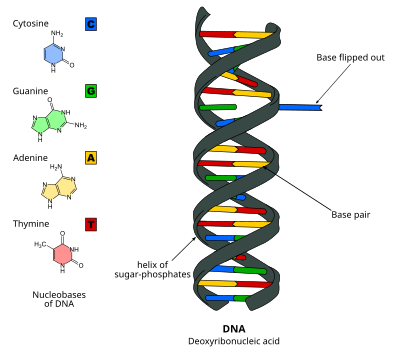
Переворот оснований ДНК, или переворот нуклеотидов, представляет собой механизм, в котором одиночное основание нуклеотида, или азотистое основание, вращается вне двойной спирали нуклеиновой кислоты[1]. Это происходит, когда ферменту, обрабатывающему нуклеиновую кислоту, требуется доступ к основанию для выполнения работы с ним, например, для его вырезания для замены другим основанием во время репарации ДНК. Впервые он был обнаружен в 1994 году с помощью рентгеновской кристаллографии фермента метилтрансферазы, катализирующего метилирование цитозинового основания в ДНК. С тех пор было показано, что он используется различными ферментами во многих биологических процессах, таких как метилирование ДНК, различные механизмы восстановления ДНК и репликация ДНК. Это также может происходить в двойных спиралях РНК[2] или в интермедиатах «ДНК:РНК», образующихся во время транскрипции РНК.
Переворот оснований ДНК происходит путем разрыва водородных связей между основаниями и отделения основания от его соседей. Это может происходить в результате активного процесса, когда фермент связывается с ДНК, а затем способствует вращению основания, или пассивного процесса, когда основание спонтанно вращается, и это состояние распознается и связывается ферментом. Его можно обнаружить с помощью рентгеновской кристаллографии, ЯМР-спектроскопии, флуоресцентной спектроскопии или гибридизационных зондов.
Открытие
[править | править код]Переворот оснований впервые наблюдался в 1994 году, когда исследователи Климасаускас, Кумар, Робертс и Ченг использовали рентгеновскую кристаллографию для наблюдения за промежуточной стадией химической реакции метилтрансферазы, связанной с ДНК[3]. Метилтрансферазой, которую они использовали, была C5-цитозинметилтрансфераза из Haemophilus haemolyticus (далее по тексту M. HhaI). Этот фермент распознает специфическую последовательность ДНК (5'-GCGC-3') и метилирует первое цитозиновое основание последовательности в его положении С5[4]. После кристаллизации комплекса M. HhaI-ДНК они увидели, что целевое цитозиновое основание полностью вывернуто из двойной спирали и расположено в активном центре M. HhaI. Он удерживался на месте благодаря многочисленным взаимодействиям между M. HhaI и ДНК[4].
Авторы предположили, что переворот оснований был механизмом, используемым многими другими ферментами, такими как хеликазы, ферменты рекомбинации, РНК-полимеразы, ДНК-полимеразы и топоизомеразы типа II[4]. В дальнейшем было проведено много исследований, и было обнаружено, что переворот оснований является механизмом, используемым во многих биологических процессах, предложенных авторами[5][6].
Механизм
[править | править код]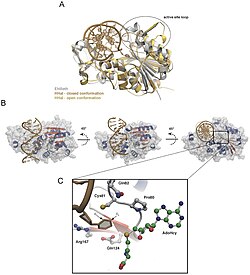
Нуклеотиды ДНК удерживаются вместе водородными связями, которые относительно слабы и легко рвутся. Переворот основания происходит в миллисекундном масштабе[7] путем разрыва водородных связей между основаниями и её отделения от соседей[8]. Основание поворачивается относительно двойной спирали на 180 градусов[9], как правило, через большую бороздку[6] в активный центр фермента. Это событие приводит к небольшим конформационным изменениям в остове ДНК[10], которые быстро стабилизируются усилением взаимодействий фермент-ДНК[6]. Исследования профилей свободной энергии при перевороте оснований показали, что барьер свободной энергии для переворота может быть снижен на 17 ккал / моль для M.HhaI в закрытой конформации.
Существует два механизма переворота оснований ДНК: активный и пассивный[11]. В активном механизме фермент связывается с ДНК, а затем активно вращает основание, в то время как в пассивном механизме поврежденное основание сначала спонтанно вращается, а затем распознается и связывается ферментом[12]. Исследования продемонстрировали оба механизма: урацил-ДНК-гликозилаза следует пассивному механизму[12], а транспозаза Tn10 следует активному механизму[13].
Кроме того, исследования показали, что переключение оснований ДНК используется многими различными ферментами в различных биологических процессах, таких как метилирование ДНК, различных механизмах репарации ДНК, транскрипции РНК и репликации ДНК[14][6][15].
Биологические процессы
[править | править код]Модификация ДНК и восстановление
[править | править код]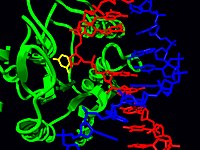
ДНК может иметь мутации, которые вызывают повреждение основания в цепи ДНК. Чтобы гарантировать генетическую целостность ДНК, ферменты должны восстанавливать любые повреждения. Существует много типов репарации ДНК. В основе процесса эксцизионной репарации оснований используется переворачивание оснований для того чтобы перевернуть поврежденное основание из двойной спирали[6] в активный центр гликозилазы, которая гидролизует гликозидную связь и удаляет основание[16]. ДНК-гликозилазы взаимодействуют с ДНК, переворачивая основания для определения несоответствия. Пример эксцизионной репарации оснований происходит, когда цитозиновое основание дезаминируется и становится урациловым основанием. Это вызывает неправильную пару U:G, которая обнаруживается урациловой ДНК-гликозилазой . Основание урацила выбрасывается в активный карман гликозилазы, где оно удаляется из цепи ДНК[17]. Переворот оснований используется для восстановления таких мутаций, как 8-оксогуанин (oxoG)[18] и димеров тимина, созданных УФ-излучением[19][20].
Репликация, транскрипция и рекомбинация
[править | править код]Репликация ДНК и транскрипция РНК используют переворот оснований[6]. ДНК-полимераза — фермент, осуществляющий репликацию. Его можно представить как руку, сжимающую одноцепочечную матрицу ДНК[21]. Когда матрица проходит через область ладони полимеразы, основания матрицы выворачиваются из спирали и удаляются от сайта связывания dNTP[22]. Во время транскрипции РНК-полимераза катализирует синтез РНК. Во время фазы инициации два основания в промоторе высвобождаются из спирали и помещаются в два кармана в РНК-полимеразе. Эти новые взаимодействия стабилизируют промотор и способствуют разделению или расплавлению нитей ДНК[23].
Переворот основания также происходит на последних стадиях рекомбинации[24]. RecA — это белок, который способствует инвазии цепи[19] во время гомологичной рекомбинации. Переворот оснований был предложен как механизм, с помощью которого RecA может позволить одной цепи распознавать гомологию в дуплексной ДНК[25]. Другие исследования показывают, что он также участвует в рекомбинации V(D)J[26].
Метилирование ДНК
[править | править код]
Метилирование ДНК представляет собой процесс, при котором метильная группа добавляется либо к цитозину, либо к аденину[27]. Этот процесс вызывает активацию или инактивацию экспрессии генов, что приводит к регуляции генов в эукариотических клетках. Известно также, что процесс метилирования ДНК участвует в некоторых типах образования рака[28][29][30]. Чтобы произошла эта химическая модификация, необходимо, чтобы целевое основание вывернулось из двойной спирали ДНК, чтобы метилтрансферазы могли катализировать реакцию[31].
Распознавание мишени эндонуклеазами рестрикции
[править | править код]Эндонуклеазы рестрикции, также известные как ферменты рестрикции, представляют собой ферменты, которые расщепляют сахаро-фосфатный остов ДНК в определённых последовательностях нуклеотидов, которые обычно имеют длину от четырёх до шести нуклеотидов[32]. Исследования, проведенные Хортоном и его коллегами, показали, что механизм, с помощью которого эти ферменты расщепляют ДНК, включает переворот оснований, а также изгибание ДНК и расширение малой бороздки[33]. В 2006 году Хортон и его коллеги представили данные рентгеновской кристаллографии, показывающие, что эндонуклеаза рестрикции HinP1I использует переворот оснований для распознавания своей последовательности-мишени. Известно, что этот фермент расщепляет ДНК по палиндромной тетрануклеотидной последовательности G↓CGC.
Экспериментальные подходы к обнаружению
[править | править код]Рентгеновская кристаллография
[править | править код]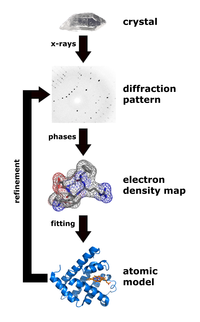
Рентгеновская кристаллография — это метод, который измеряет углы и интенсивности кристаллических атомов, чтобы определить атомную и молекулярную структуру интересующего кристалла. Затем кристаллографы могут создавать трехмерные изображения, на которых можно определить положение атомов, химических связей, а также другие важные характеристики[34]. Климасаукас и его коллеги использовали эту технику для наблюдения первого явления переворота базы, в котором их экспериментальная процедура включала несколько этапов[3]:
- Очищение
- Кристаллизация
- Сбор данных
- Определение и уточнение структуры
Во время очистки метилтрансфераза Haemophilus haemolyticus подвергалась сверхэкспрессии и очищалась с использованием стадии обратной экстракции с высоким содержанием соли для селективного растворения M.HhaI с последующей быстрой белковой жидкостной хроматографией (FPLC), как это было сделано ранее Кумаром и его коллегами[35]. Авторы использовали анионообменную колонку Mono-Q для удаления небольшого количества белковых материалов и нежелательной ДНК перед стадией кристаллизации. После того, как M.HhaI был успешно очищен, образец был выращен с использованием метода, который смешивает раствор, содержащий комплекс, при температуре 16 °C и метод диффузии паров висячей капли для получения кристаллов. Затем авторы смогли собрать рентгеновские данные в соответствии с методом, использованным Ченгом и его коллегами в 1993 году[36]. Этот метод включал измерение интенсивности дифракции на детекторе FAST, где время экспозиции для поворота на 0,1° составляло 5 или 10 секунд. Для определения и уточнения структуры Климасаукас и его коллеги использовали молекулярную замену уточненной структуры апо, описанную Ченгом и его коллегами в 1993 г.[36], где поисковые модели X-PLOR, MERLOT и TRNSUM использовались для решения функций вращения и трансляции[37][38]. Эта часть исследования включает использование различных программ и компьютерных алгоритмов для определения структуры и характеристик интересующего кристалла.
ЯМР-спектроскопия
[править | править код]ЯМР-спектроскопия — это метод, который на протяжении многих лет использовался для изучения важных динамических аспектов переворота оснований. Он позволяет исследователям определять физические и химические свойства атомов и других молекул, используя магнитные свойства атомных ядер[39]. Кроме того, ЯМР может предоставить разнообразную информацию, включая структуру, состояние реакции, химическое окружение молекул и динамику[40][41]. Во время эксперимента по открытию переворота основания ДНК исследователи использовали ЯМР-спектроскопию для исследования индуцированного ферментом переворота основания HhaI-метилтрансферазы. Чтобы выполнить этот эксперимент, два остатка 5-фторцитозина были включены в мишень и контрольное положение с ДНК-субстратом, чтобы можно было выполнить анализ химического сдвига 19F . После оценки анализа химического сдвига 19F был сделан вывод, что комплексы ДНК существуют с множественными формами целевого 5-фторцитозина вдоль пути переключения оснований[42].
Флуоресцентная спектроскопия
[править | править код]Флуоресцентная спектроскопия — это метод, который используется для анализа образца с использованием флуоресцентного зонда. Нуклеотиды ДНК сами по себе не являются хорошими кандидатами для этого метода, потому что они слабо излучают свет при световом возбуждении[43]. Для обнаружения переворачивания базы необходим флуоресцентный маркер. 2-Аминопурин представляет собой основание, которое структурно похоже на аденин, но способное к флуоресценции при его отделении от дуплекса ДНК[44]. Он обычно используется для обнаружения переворачивания основания и имеет возбуждение на уровне 305—320.нм и излучение при 370нм так, чтобы он хорошо отделялся от возбуждений белков и ДНК. Другими флуоресцентными зондами, используемыми для изучения переворота оснований ДНК, являются 6MAP (4-амино-6-метил-7(8H)-птеридон)[45] и пирроло-C (3-[β-D-2-рибофуранозил]-6-метилпирроло [2,3-d]пиримидин-2(3H)-он)[46][47] Флуоресцентная спектроскопия с временным разрешением также используется для получения более подробной картины степени переворота основания, а также конформационной динамики, происходящей во время переворота основания[48].
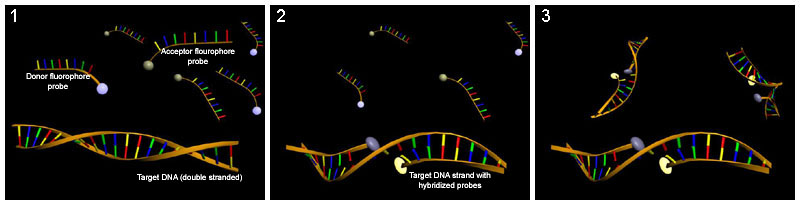
Гибридизационное зондирование
[править | править код]Гибридизационные зонды можно использовать для обнаружения переворачивания оснований. В этом методе используется молекула, которая имеет последовательность, комплементарную последовательности, которую вы хотите обнаружить, так что она связывается с одноцепочечной ДНК или РНК. Несколько зондов гибридизации использовались для обнаружения переворачивания оснований. Перманганат калия используется для обнаружения остатков тимина, которые были высвобождены цитозин-С5 и аденин-N6 метилтрансферазами[49]. Хлорацетальдегид используется для обнаружения остатков цитозина, высвобождаемых ДНК-цитозин-5-метилтрансферазой HhaI (M. HhaI)[50].
См. также
[править | править код]- Восстановление ДНК
- Эксцизионная репарация
- Репликация ДНК
- Транскрипция РНК
- Метилирование ДНК
- ДНК-метилтрансфераза
- Генетическая рекомбинация
- Гомологическая рекомбинация
- ДНК
- Эпигенетика
- Эпигеномика
Использованная литература
[править | править код]- ↑ Roberts, RJ (1998). "Base flipping". Annual Review of Biochemistry. 67 (1): 181—198. doi:10.1146/annurev.biochem.67.1.181. PMID 9759487.
- ↑ Reiter, NJ (2004). "Dynamics in the U6 RNA intramolecular stem-loop: a base flipping conformational change". Biochemistry. 43 (43): 13739—47. doi:10.1021/bi048815y. PMID 15504036.
- ↑ 1 2 Saulius Klimasauskas, Sanjay Kumar, Richard J. Roberts, Xiaodong Cheng. Hhal methyltransferase flips its target base out of the DNA helix (англ.) // Cell. — 1994-01. — Vol. 76, iss. 2. — P. 357–369. — doi:10.1016/0092-8674(94)90342-5. Архивировано 15 июня 2022 года.
- ↑ 1 2 3 Klimasauskas, Saulius (January 1994). "Hhal methyltransferase flips its target base out of the DNA helix". Cell. 76 (2): 357—369. doi:10.1016/0092-8674(94)90342-5. PMID 8293469.
- ↑ ATDBio - Base flipping. atdbio.com. Дата обращения: 19 августа 2022. Архивировано 19 августа 2022 года.
- ↑ 1 2 3 4 5 6 Niu Huang, Nilesh K. Banavali, Alexander D. MacKerell. Protein-facilitated base flipping in DNA by cytosine-5-methyltransferase (англ.) // Proceedings of the National Academy of Sciences. — 2003-01-07. — Vol. 100, iss. 1. — P. 68–73. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.0135427100. Архивировано 19 августа 2022 года.
- ↑ Bouvier, Benjamin (August 2007). "A Molecular Dynamics Study of Slow Base Flipping in DNA using Conformational Flooding" (PDF). Biophysical Journal. 93 (3): 770—786. Bibcode:2007BpJ....93..770B. doi:10.1529/biophysj.106.091751. PMID 17496048. Архивировано из оригинала (PDF) 9 августа 2017. Дата обращения: 18 августа 2022.
- ↑ Laurent Larivière, Solange Moréra. Structural Evidence of a Passive Base-flipping Mechanism for β-Glucosyltransferase (англ.) // Journal of Biological Chemistry. — 2004-08. — Vol. 279, iss. 33. — P. 34715–34720. — doi:10.1074/jbc.M404394200.
- ↑ DNA and RNA modification enzymes : structure, mechanism, function, and evolution. — Austin, Tex.: Landes Bioscience, 2009. — 1 online resource (653 pages) с. — ISBN 978-1-4416-1558-9, 1-4416-1558-X, 978-1-4337-1155-8, 1-4337-1155-9, 978-1-58706-361-9, 1-58706-361-1, 978-1-4987-1315-3, 1-4987-1315-7.
- ↑ Giudice, E. (1 March 2003). "Base pair opening within B-DNA: free energy pathways for GC and AT pairs from umbrella sampling simulations". Nucleic Acids Research. 31 (5): 1434—1443. doi:10.1093/nar/gkg239. PMID 12595551.
- ↑ O'Neil, Lauren. Base Flipping in DNA: Detection, Structures and Energetics, A Dissertation.. — 2014. — ISBN 9780549590743..
- ↑ 1 2 Lariviere, L. (23 June 2004). "Structural Evidence of a Passive Base-flipping Mechanism for Glucosyltransferase". Journal of Biological Chemistry. 279 (33): 34715—34720. doi:10.1074/jbc.M404394200. PMID 15178685.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Bischerour, Julien (10 July 2009). "Base Flipping in Tn10 Transposition: An Active Flip and Capture Mechanism". PLOS ONE. 4 (7): e6201. Bibcode:2009PLoSO...4.6201B. doi:10.1371/journal.pone.0006201. PMID 19593448.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ Brown. Nucleic Acids Book. Дата обращения: 26 февраля 2014. Архивировано 17 февраля 2021 года.
- ↑ Grubmüller. DNA Base Flipping. Дата обращения: 26 февраля 2014. Архивировано из оригинала 4 февраля 2017 года.
- ↑ Molecular biology of the gene. — Seventh edition. — Boston, 2014. — xxxiv, 872 pages с. — ISBN 978-0-321-76243-6, 0-321-76243-6, 978-0-321-90537-6, 0-321-90537-7, 978-0-321-90264-1, 0-321-90264-5, 978-0-321-85149-9, 0-321-85149-8. Архивировано 29 сентября 2021 года.
- ↑ Krokan, Hans E (16 December 2002). "Uracil in DNA – occurrence, consequences and repair". Oncogene. 21 (58): 8935—8948. doi:10.1038/sj.onc.1205996. PMID 12483510.
- ↑ Banerjee, Anirban (31 March 2005). "Structure of a repair enzyme interrogating undamaged DNA elucidates recognition of damaged DNA". Nature. 434 (7033): 612—618. Bibcode:2005Natur.434..612B. doi:10.1038/nature03458. PMID 15800616.
- ↑ 1 2 Julien Bischerour, Ronald Chalmers. Base flipping in tn10 transposition: an active flip and capture mechanism // PloS One. — 2009-07-10. — Т. 4, вып. 7. — С. e6201. — ISSN 1932-6203. — doi:10.1371/journal.pone.0006201. Архивировано 18 августа 2022 года.
- ↑ Fuxreiter, Monika (November 2002). "Role of Base Flipping in Specific Recognition of Damaged DNA by Repair Enzymes". Journal of Molecular Biology. 323 (5): 823—834. doi:10.1016/S0022-2836(02)00999-3. PMID 12417196.
- ↑ Anirban Banerjee, Wei Yang, Martin Karplus, Gregory L. Verdine. Structure of a repair enzyme interrogating undamaged DNA elucidates recognition of damaged DNA (англ.) // Nature. — 2005-03. — Vol. 434, iss. 7033. — P. 612–618. — ISSN 1476-4687 0028-0836, 1476-4687. — doi:10.1038/nature03458. Архивировано 18 августа 2022 года.
- ↑ Patel, Premal H. (May 2001). "Prokaryotic DNA polymerase I: evolution, structure, and "base flipping" mechanism for nucleotide selection". Journal of Molecular Biology. 308 (5): 823—837. doi:10.1006/jmbi.2001.4619. PMID 11352575.
- ↑ Lim, H. M. (4 December 2001). "A "master" in base unpairing during isomerization of a promoter upon RNA polymerase binding". Proceedings of the National Academy of Sciences. 98 (26): 14849—14852. Bibcode:2001PNAS...9814849L. doi:10.1073/pnas.261517398. PMID 11734629.
- ↑ Voloshin, Oleg N. (September 2004). "Synaptic Complex Revisited". Molecular Cell. 15 (6): 846—847. doi:10.1016/j.molcel.2004.09.010. PMID 15383274.
- ↑ Folta-Stogniew, E (Sep 24, 2004). "Exchange of DNA base pairs that coincides with recognition of homology promoted by E. coli RecA protein". Molecular Cell. 15 (6): 965—75. doi:10.1016/j.molcel.2004.08.017. PMID 15383285.
- ↑ Bischerour, J. (31 August 2009). "Base Flipping in V(D)J Recombination: Insights into the Mechanism of Hairpin Formation, the 12/23 Rule, and the Coordination of Double-Strand Breaks". Molecular and Cellular Biology. 29 (21): 5889—5899. doi:10.1128/MCB.00187-09. PMID 19720743.
- ↑ Klose, Robert J. (2006). "Genomic DNA methylation: the mark and its mediators". Trends in Biochemical Sciences. 31 (2): 89—97. doi:10.1016/j.tibs.2005.12.008. ISSN 0968-0004. PMID 16403636.
- ↑ Nakao, M (2001). "Epigenetics: Interaction of DNA Methylation and Chromatin". Gene. 278 (1—2): 25—31. doi:10.1016/s0378-1119(01)00721-1. PMID 11707319.
- ↑ Plass, C (2002). "DNA Methylation, Imprinting and Cancer". Eur J Hum Genet. 10 (1): 6—16. doi:10.1038/sj.ejhg.5200768. PMID 11896451.
- ↑ Esteller, M (2002). "Cancer as an Epigenetic Disease: DNA Methylation and Chromatin Alterations in Human Tumours". J Pathol. 196 (1): 1—7. doi:10.1002/path.1024. PMID 11748635.
- ↑ Huang, Niu (January 7, 2003). "Protein-facilitated base flipping in DNA by cytosine-5-methyltransferase". PNAS. 100 (1): 68—73. Bibcode:2003PNAS..100...68H. doi:10.1073/pnas.0135427100. PMID 12506195.
- ↑ Archived copy. Дата обращения: 3 апреля 2014. Архивировано из оригинала 18 апреля 2014 года.
- ↑ Horton, John R. (2006). "DNA Nicking by HinP1I Endonuclease: Bending, Base Flipping and Minor Groove Expansion". Nucleic Acids Research. 34 (3): 939—948. doi:10.1093/nar/gkj484. PMID 16473850.
- ↑ X-ray crystallography
- ↑ Kumar, S (1992). "Purification, Crystallization, and Preliminary X-ray Diffraction Analysis of an M.HhaI-AdoMet Complex". Biochemistry. 31 (36): 8648—8653. doi:10.1021/bi00151a035. PMID 1390649.
- ↑ 1 2 Cheng, X (1993). "Crystal Structure of the HhaI DNA Methyltransferase Complexed with S-adenosyl-L-methionine". Cell. 74 (2): 299—307. doi:10.1016/0092-8674(93)90421-l. PMID 8343957.
- ↑ Brunger A.T. (1992)"X-PLOR, Version 3.1 : A system for x-ray crystallography and NMR"(New Haven, Connecticut: Yale University Press)
- ↑ Fitzgerald, P.M.D. (1988). "MERLOT, an integral package of computer programs for the determination of crystal structure by molecular replacement". J. Appl. Crystallogr. 21 (3): 273—288. doi:10.1107/s0021889887012299.
- ↑ NMR spectroscopy
- ↑ Gueron, M., and J. L. Leroy. 1995. Studies of basepair kinetics by NMR measurement of proton exchange. In Nuclear Magnetic Resonance And Nucleic Acids. Academic Press, San Diego, CA.
- ↑ Leijon, M. (1992). "Effects of sequence and length on imino proton-exchange and basepair opening kinetics in DNA oligonucleotide duplexes". Nucleic Acids Res. 20 (20): 5339—5343. doi:10.1093/nar/20.20.5339. PMID 1331987.
- ↑ Klimasaukas, Salius, and Zita Liutkeviciute. «Experimental Approaches to Study DNA Base Flipping.» DNA and RNA Modification Enzymes: Structure, Mechanism, Function and Evolution. Landes Bioscience, 2009. 37-50. Web. 16 Mar. 2014. <https://www.landesbioscience.com/pdf/04GrosjeanKlimasauskas.pdf Архивировано 7 апреля 2014 года.>.
- ↑ Grosjean, [edited by] Henri. DNA and RNA modification enzymes : structure, mechanism, function and evolution. — ISBN 978-1-58706-329-9. Архивная копия от 7 апреля 2014 на Wayback Machine
- ↑ Holz, B (15 February 1998). "2-Aminopurine as a fluorescent probe for DNA base flipping by methyltransferases". Nucleic Acids Research. 26 (4): 1076—1083. doi:10.1093/nar/26.4.1076. PMID 9461471.
- ↑ Yang, K (Sep 6, 2007). "6MAP, a fluorescent adenine analogue, is a probe of base flipping by DNA photolyase". The Journal of Physical Chemistry B. 111 (35): 10615—25. doi:10.1021/jp071035p. PMID 17696385.
- ↑ Yang, K (May-Jun 2008). "The extent of DNA deformation in DNA photolyase-substrate complexes: a solution state fluorescence study". Photochemistry and Photobiology. 84 (3): 741—9. doi:10.1111/j.1751-1097.2007.00251.x. PMID 18086248.
{{cite journal}}: Википедия:Обслуживание CS1 (формат даты) (ссылка) - ↑ Berry, David A. (March 2004). "Pyrrolo-dC and pyrrolo-C: fluorescent analogs of cytidine and 2′-deoxycytidine for the study of oligonucleotides". Tetrahedron Letters. 45 (11): 2457—2461. doi:10.1016/j.tetlet.2004.01.108.
- ↑ Neely, R. K. (8 September 2009). "Time-resolved fluorescence studies of nucleotide flipping by restriction enzymes". Nucleic Acids Research. 37 (20): 6859—6870. doi:10.1093/nar/gkp688. PMID 19740769.
- ↑ Serva, S (1 August 1998). "Chemical display of thymine residues flipped out by DNA methyltransferases". Nucleic Acids Research. 26 (15): 3473—3479. doi:10.1093/nar/26.15.3473. PMID 9671807.
- ↑ Daujotyte, D. (15 April 2008). "Chemical mapping of cytosines enzymatically flipped out of the DNA helix". Nucleic Acids Research. 36 (10): e57. doi:10.1093/nar/gkn200. PMID 18450817.
На эту статью не ссылаются другие статьи Википедии. |