Белки скользящего зажима
Белки́ скользя́щего зажима, или скользя́щий зажи́м (англ. DNA clamp) — белки, которые выполняют функцию усилителя процессивности при репликации ДНК.
Белки скользящего зажима являются важным компонентом холофермента ДНК-полимеразы III и предотвращают диссоциацию фермента от матрицы ДНК. Так как лимитирующей стадией в реакции синтеза ДНК является связывание ДНК-полимеразы с матрицей, присутствие белка скользящего зажима значительно увеличивает количество присоединяемых к растущей цепи нуклеотидов за один акт присоединения фермента к матрице. Это объясняется тем, что белок-белковое взаимодействие сильнее и является более специфическим, чем взаимодействие полимеразы и матрицы ДНК. Белки скользящего зажима увеличивают скорость синтеза ДНК до тысячи раз в сравнении с непроцессивной полимеразой[2].
Структура
[править | править код]Белки скользящего зажима (застёжки) представляют собой α+β-белки, которые собираются в мультимерные структуры, полностью окружающие кольцом двойную спираль ДНК, когда ДНК-полимераза добавляет нуклеотиды на растущую цепь[3]. Они окружают ДНК в области репликационной вилки и «скользят» по ДНК вместе с продвигающейся вперёд полимеразой. Скольжение облегчается наличием слоя молекул воды в центральной поре зажима; этот слой разделяет поверхность белка и ДНК, играя роль смазки. Из-за тороидальной формы мультимера зажим не может диссоциировать от ДНК без распада на мономеры.
Белки скользящего зажима выявлены у бактерий, архей, эукариот и некоторых вирусов. У бактерий застёжка представляет собой гомодимер, состоящий из двух идентичных β-субъединиц ДНК-полимеразы III, и поэтому называется β-зажим. У архей[4] и эукариот застёжка является тримером из трёх молекул PCNA[англ.]. Фаг Т4 также имеет скользящую застёжку. Она называется gp45 и представляет собой тример, по структуре схожий с тримером архей и эукариот, однако входящие в его состав мономеры не обнаруживают гомологии последовательности аминокислот как с PCNA, так и с β-субъединицами[3].
| Царство | Белки скользящего зажима | Агрегационное состояние | Связанная ДНК-полимераза |
|---|---|---|---|
| Бактерии | β-субъединицы ДНК-полимеразы III | димер | ДНК-полимераза III |
| Археи | PCNA[англ.] архей | тример | ДНК-полимераза ε |
| Эукариоты | PCNA | тример | ДНК-полимераза δ[англ.] |
| Вирусы | gp43 / gp45 | тример | RB69 ДНК-полимераза / T4 ДНК-полимераза |
Бактерии
[править | править код]
Как уже говорилось, у бактерий скользящая застёжка представляет собой димер из двух β-субъединиц холофермента ДНК-полимеразы III (β-зажим). Две β-субъединицы собираются около ДНК при помощи γ-субъединицы и за счёт энергии гидролиза АТФ. После сборки димера вокруг ДНК сродство β-субъединиц к γ-субъединице сменяется сродством к α- и ε-субъединицам; таким образом формируется полный холофермент[6][7][8]. ДНК-полимераза III является важнейшим ферментативным комплексом, задействованным в репликации ДНК у бактерий.
γ-комплекс ДНК-полимеразы III, образованный субъединицами γδδ'χψ, катализирует гидролиз АТФ и направляет полученную энергию на сборку β-димера вокруг ДНК, выступая, таким образом, в роли шаперона. Связавшись с ДНК, β-димер может свободно скользить по двойной спирали ДНК. α-субъединица обеспечивает полимеразную активность ДНК-полимеразы, а ε-субъединица играет роль 3'—5'-экзонуклеазы[8].
β-субъединица бактериальной ДНК-полимеразы III состоит из трёх топологически неэквивалентных доменов (С-концевого, центрального и N-концевого). Две β-субъединицы плотно взаимодействуют друг с другом, формируя замкнутое кольцо вокруг двойной спирали ДНК.
Эукариоты и археи
[править | править код]
У эукариот скользящая застёжка состоит из специфических субъединиц ДНК-полимеразы δ, называемых ядерным антигеном пролиферирующих клеток (англ. Proliferating cell nuclear antigen, PCNA[англ.]). С-концевые и N-концевые домены PCNA топологически идентичны. Три молекулы PCNA плотно взаимодействуют друг с другом, формируя замкнутое кольцо вокруг двойной спирали ДНК.
Аминокислотная последовательность PCNA достаточно консервативна среди животных и растений. Это иллюстрирует давление естественного отбора, способствующее консервативности структуры, а также подтверждает, что такой тип репликации ДНК является общим для всех эукариот[10].
Белки, гомологичные PCNA, были также выявлены у архей (Euryarchaeota и Crenarchaeota), у вируса Paramecium bursaria Chlorella virus 1 (PBCV-1), а также у ядерных полиэдрозных вирусов[англ.].
Вирусы
[править | править код]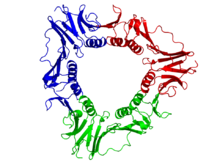
Субъединица вирусного белка скользящего зажима — gp45 — включает 2 домена. Каждый домен состоит из двух α-спиралей и двух β-слоёв. Таким образом, эта субъединица содержит 2 топологически идентичных фолда и имеет внутреннюю псевдосимметрию относительно них. 3 молекулы gp45 плотно взаимодействуют друг с другом, формируя замкнутое кольцо вокруг двойной спирали ДНК[12].
Сборка
[править | править код]Белки скользящего зажима доставляются на соответствующую двойную спираль ДНК особым белком, известным как репликационный фактор С[англ.] (белки-погрузчики белков скользящего зажима[13]), которые также разбирают комплекс застёжки после того, как репликация завершится. Сайты связывания этих инициаторных белков (погрузчиков) перекрываются с сайтами связывания ДНК-полимеразы, поэтому белки застёжки не могут одновременно быть связанными с погрузчиками и ДНК-полимеразой. Поэтому комплекс застёжки не будет разбираться, пока они остаются связанными с ДНК-полимеразой. Белки скользящего зажима также связываются с другими факторами, участвующими в поддержании гомеостаза ДНК и генома, например, факторами сборки нуклеосом, лигазами, сшивающими фрагменты Оказаки, а также белками репарации ДНК. У всех этих белков сайты связывания на белках зажима также перекрываются с сайтами связывания погрузчиков. Это тоже гарантирует то, что застёжка не будет разобрана, пока любой из этих ферментов продолжает работать. Для работы белков-погрузчиков необходима энергия гидролиза АТФ, чтобы замкнуть белки застёжки вокруг ДНК.
Примечания
[править | править код]- ↑ PDB 1W60; Kontopidis G., Wu S. Y., Zheleva D. I., Taylor P., McInnes C., Lane D. P., Fischer P. M., Walkinshaw M. D. Structural and biochemical studies of human proliferating cell nuclear antigen complexes provide a rationale for cyclin association and inhibitor design (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2005. — February (vol. 102, no. 6). — P. 1871—1876. — doi:10.1073/pnas.0406540102. — PMID 15681588. — PMC 548533.
- ↑ V. Mizrahi, R. N. Henrie, J. F. Marlier, K. A. Johnson, S. J. Benkovic. Rate-limiting steps in the DNA polymerase I reaction pathway (англ.) // Biochemistry : journal. — 1985. — Vol. 24, no. 15. — P. 4010—4018. — doi:10.1021/bi00336a031. Архивировано 25 сентября 2019 года.
- ↑ 1 2 Bruck I., O'Donnell M. The ring-type polymerase sliding clamp family (англ.) // Genome Biol.[англ.] : journal. — 2001. — Vol. 2, no. 1. — P. REVIEWS3001. — doi:10.1186/gb-2001-2-1-reviews3001. — PMID 11178284. — PMC 150441.
- ↑ Matsumiya S., Ishino Y., Morikawa K. Crystal structure of an archaeal DNA sliding clamp: Proliferating cell nuclear antigen from Pyrococcus furiosus (англ.) // Protein Sci.[англ.] : journal. — 2001. — January (vol. 10, no. 1). — P. 17—23. — doi:10.1110/ps.36401. — PMID 11266590. — PMC 2249843.
- ↑ PDB 1MMI; Oakley A. J., Prosselkov P., Wijffels G., Beck J. L., Wilce M. C., Dixon N. E. Flexibility revealed by the 1.85 Å crystal structure of the beta sliding-clamp subunit of Escherichia coli DNA polymerase III (англ.) // Acta Crystallogr. D Biol. Crystallogr.[англ.] : journal. — International Union of Crystallography, 2003. — July (vol. 59, no. Pt 7). — P. 1192—1199. — doi:10.1107/S0907444903009958. — PMID 12832762.
- ↑ Lewin, Benjamin. Genes VI (англ.). — Oxford [Oxfordshire]: Oxford University Press, 1997. — P. 484—487. — ISBN 0-19-857779-6.
- ↑ Lehninger, Albert L. Biochemistry: The Molecular Basis of Cell Structure and Function (англ.). — New York: Worth Publishers, 1975. — P. 894. — ISBN 0-87901-047-9.
- ↑ 1 2 Stukenberg P. T., Studwell-Vaughan P. S., O'Donnell M. Mechanism of the sliding beta-clamp of DNA polymerase III holoenzyme (англ.) // J. Biol. Chem. : journal. — 1991. — June (vol. 266, no. 17). — P. 11328—11334. — PMID 2040637. Архивировано 7 июля 2019 года.
- ↑ PDB 1AXC; Gulbis J. M., Kelman Z., Hurwitz J., O'Donnell M., Kuriyan J. Structure of the C-terminal region of p21(WAF1/CIP1) complexed with human PCNA (англ.) // Cell : journal. — Cell Press, 1996. — October (vol. 87, no. 2). — P. 297—306. — doi:10.1016/S0092-8674(00)81347-1. — PMID 8861913.
- ↑ Suzuka I., Hata S., Matsuoka M., Kosugi S., Hashimoto J. Highly conserved structure of proliferating cell nuclear antigen (DNA polymerase delta auxiliary protein) gene in plants (англ.) // Eur. J. Biochem.[англ.] : journal. — 1991. — January (vol. 195, no. 2). — P. 571—575. — doi:10.1111/j.1432-1033.1991.tb15739.x. — PMID 1671766.
- ↑ PDB 1CZD; Moarefi I., Jeruzalmi D., Turner J., O'Donnell M., Kuriyan J. Crystal structure of the DNA polymerase processivity factor of T4 bacteriophage (англ.) // J. Mol. Biol.[англ.] : journal. — 2000. — March (vol. 296, no. 5). — P. 1215—1223. — doi:10.1006/jmbi.1999.3511. — PMID 10698628.
- ↑ Steitz T. A., Shamoo Y. Building a replisome from interacting pieces: sliding clamp complexed to a peptide from DNA polymerase and a polymerase editing complex (англ.) // Cell : journal. — Cell Press, 1999. — Vol. 99, no. 2. — P. 155—166. — doi:10.1016/S0092-8674(00)81647-5. — PMID 10535734.
- ↑ Калинин, с. 35.
Литература
[править | править код]- Калинин В. Л. Репликация генома. — 120 с.
См. также
[править | править код]
Эта статья входит в число добротных статей русскоязычного раздела Википедии. |

