Нанопоровое секвенирование: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
м Добавлены ссылки, к неподтвержденным утверждениям. |
|||
| Строка 110: | Строка 110: | ||
==== Пост-обработка данных полученных с помощью Oxford Nanopore ==== |
==== Пост-обработка данных полученных с помощью Oxford Nanopore ==== |
||
После использования продукции Oxford Nanopore на выходе имеются сырые данные формата FAST5. Формат FAST5<ref>{{Статья|ссылка=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4722697/|автор=Camilla L.C. Ip, Matthew Loose, John R. Tyson, Mariateresa de Cesare, Bonnie L. Brown|заглавие=MinION Analysis and Reference Consortium: Phase 1 data release and analysis|год=2015-10-15|издание=F1000Research|том=4|issn=2046-1402|doi=10.12688/f1000research.7201.1}}</ref>, используемый [[:en:Oxford Nanopore Technologies|Oxford Nanopore]] — это вариант стандарта [[Hierarchical Data Format|HDF5]] с иерархической внутренней структурой, предназначенной для хранения [[Метаданные|метаданных]], связанных с последовательностью ДНК и событий (агрегированные измерения общего тока) предварительно обработанных вашим рабочим устройством(прим. MinION). Результаты обработки отображаются в реальном времени в графическом интерфейсе MinKNOW, а данные записываются в формате файлов [[FASTQ]] или .fast5. Далее нужно произвести процесс base-calling’а который обработает сырые данные формата FAST5 в [[FASTQ]] формат (в программе MinKNOW этот процесс можно запустить во время прочтения ридов). Также можно использовать такие программы как [https://github.com/arq5x/poretools/tree/master/poretools poreTools], [https://github.com/nanoporetech/pyguppyclient Guppy]. |
После использования продукции Oxford Nanopore на выходе имеются сырые данные формата FAST5. Формат FAST5<ref>{{Статья|ссылка=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4722697/|автор=Camilla L.C. Ip, Matthew Loose, John R. Tyson, Mariateresa de Cesare, Bonnie L. Brown|заглавие=MinION Analysis and Reference Consortium: Phase 1 data release and analysis|год=2015-10-15|издание=F1000Research|том=4|issn=2046-1402|doi=10.12688/f1000research.7201.1}}</ref>, используемый [[:en:Oxford Nanopore Technologies|Oxford Nanopore]] — это вариант стандарта [[Hierarchical Data Format|HDF5]] с иерархической внутренней структурой, предназначенной для хранения [[Метаданные|метаданных]], связанных с последовательностью ДНК и событий (агрегированные измерения общего тока) предварительно обработанных вашим рабочим устройством(прим. MinION). Результаты обработки отображаются в реальном времени в графическом интерфейсе MinKNOW, а данные записываются в формате файлов [[FASTQ]] или .fast5. Далее нужно произвести процесс base-calling’а который обработает сырые данные формата FAST5 в [[FASTQ]] формат (в программе MinKNOW этот процесс можно запустить во время прочтения ридов). Также можно использовать такие программы как <ref>{{Статья|ссылка=https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-018-5381-7|автор=Sarah Goldstein, Lidia Beka, Joerg Graf, Jonathan L. Klassen|заглавие=Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing|год=2019-12|язык=en|издание=BMC Genomics|том=20|выпуск=1|страницы=23|issn=1471-2164|doi=10.1186/s12864-018-5381-7}}</ref>[https://github.com/arq5x/poretools/tree/master/poretools poreTools], [https://github.com/nanoporetech/pyguppyclient Guppy]. |
||
Далее нужно очистить полученные последовательности, чтобы избавиться от данных со слишком большим шумом. Для этой задачи |
Далее нужно очистить полученные последовательности, чтобы избавиться от данных со слишком большим шумом. Для этой задачи используется такая программа как <ref>{{Статья|ссылка=http://genomea.asm.org/lookup/doi/10.1128/MRA.00156-20|автор=Maria Stein, Erik Brinks, Jana Rathje, Gyu-Sung Cho, Charles M. A. P. Franz|заглавие=Complete Genome Sequence of Tetracycline-Resistant Serratia liquefaciens S1, Isolated from Mixed Greens, Obtained Using Illumina MiSeq and Oxford Nanopore MinION Sequencing|год=2020-05-07|ответственный=David A. Baltrus|язык=en|издание=Microbiology Resource Announcements|том=9|выпуск=19|страницы=e00156–20, /mra/9/19/MRA.00156–20.atom|issn=2576-098X|doi=10.1128/MRA.00156-20}}</ref>[https://pypi.org/project/NanoFilt/ NanoFilt]. Как только данные будут очищены полученные данные дальше можно использовать для последующей [[Сборка генома|сборки]] и анализа данных. |
||
== Примечания == |
== Примечания == |
||
Версия от 21:10, 11 мая 2020
Нанопоровое секвенирование — семейство высокоэффективных методов секвенирования ДНК или РНК третьего поколения[1]. Метод основан на использовании белковых, твердотельных или иных пор диаметром в несколько нанометров, чувствительных к ДНК.
Нанопоровое секвенирование позволяет избежать стадий ПЦР-амплификации и химического мечения образца ДНК или РНК. Это является существенным преимуществом по сравнению с другими методами секвенирования, которые используют хотя бы одну из этих стадий. Возможности метода включают относительно дешёвое генотипирование, высокую мобильность, быстрый анализ и отображение результатов в реальном времени. Было описано использование метода в быстром выявлении вирусных патогенов[2], отслеживании бактериальной резистентности[3], секвенировании генома человека[4][5] и растений[6], гаплотипировании[7], отслеживании вируса эболы[8] и других областях.
История
В 1995 году впервые была предложена идея нанопорового секвенирования — определение свойств линейного полимера при его протаскивании через пору в мембране. При прохождении через пору полимер определённым образом с ней взаимодействует, что позволяет определить его свойства[9]. Спустя год — в 1996 году — появилась первая работа, описывающая возможность применения нанопор (в качестве нанопоры был использован альфа-гемолизин) для определения характеристик нуклеиновых кислот[10].
В 1999—2000 годах была показано, что используя в качестве нанопоры альфа-гемолизин стафилококка, можно отличить одноцепочечную РНК от одноцепочечной ДНК[11][12].
В 2001 году впервые была проведена работа, в которой с помощью нанопор определяли наличие коротких последовательностей ДНК[13]. Только к 2009 году удалось показать необходимую для создания методов секвенирования возможность различать нанопорами все основания в последовательности ДНК[14].
В 2012 году компанией Oxford Nanopore были продемонстрированы первые нанопоровые секвенаторы: GridION и MinION[15].
Тогда же была показана принципиальная возможности применения данного метода — был секвенирован геном бактериофага phiX длиной 5,4 килобазы[16].
Принцип работы
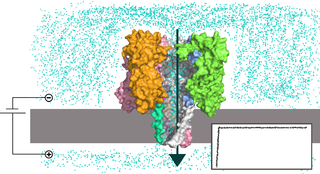
Нанопоровые системы представляют собой реакционную камеру, внутри которой находится раствор электролита. Камера разделена на две части липидной мембраной или иной тонкой непроводящей поверхностью, в которую внедрена единичная нанопора. К частям камеры прикладывают напряжение, из-за чего возникает ток ионов через пору. Когда исследуемые молекулы проходят через пору по направлению действия поля, они уменьшают сечение, доступное для ионов, и сила тока падает. Анализируя изменение силы тока, можно определить свойства молекулы, проходящей через пору [17].
Диаметр используемых для нуклеиновых кислот нанопор составляет несколько нанометров, из-за чего ДНК и РНК способны проходить сквозь пору только в одноцепочечной форме, но не в двухцепочечной. При прохождении молекулы нуклеиновой кислоты через пору отдельные нуклеотиды задерживаются в определённых сайтах внутри поры, в результате чего происходит измеримое падение силы тока [17].
Достоинства нанопорового секвенирования
По сравнению с уже существующими методами секвенирования применение нанопор обладает следующими преимуществами[18]:
- Дешевизна и простота использования (достигается отсутствием необходимости приготовления образца и использования реактивов);
- Высокая чувствительность, вплоть до секвенирования без амплификации ДНК из крови или слюны;
- Высокая длина прочтений, вплоть до десятков тысяч оснований.
Недостатки нанопорового секвенирования
- Низкое качество прочтения по сравнению с технологиями секвенирования с короткими чтениями, однако эта ситуация меняется в лучшую сторону с появлением новых алгоритмов[19];
- Потеря функциональных свойств биологических пор c течением времени;
- Влияние факторов среды на скорость прочтения последовательности и, следовательно, на его качество;
Варианты нанопорового секвенирования
В зависимости от того, сохраняют ли секвенируемые молекулы нуклеиновых кислот свою химическую целостность, выделяют следующие методы[20]
Секвенирование целых цепочек
В данном методе цепи нуклеиновых кислот не расщепляются. Перенос целых молекул ДНК и РНК через пору может осуществляться следующими способами:
Транспорт под действием напряжения
Так как ДНК и РНК несут на себе отрицательный заряд, то самым простым способом транспорта молекулы нуклеиновой кислоты через пору является их электрофоретический перенос вместе с ионами. Проблемой данного метода является то, что для измерения падения тока ионов через пору изначально требуется большой ток, чтобы получить хорошее соотношение сигнал/шум. Но при увеличении приложенного напряжения увеличивается и скорость, с которой молекула нуклеиновой кислоты преодолевает пору, а значит уменьшается время распознавания каждого отдельного основания, из-за чего качество распознавания падает.
Транспорт под действием напряжения с расплетанием дуплексов
Уменьшить скорость прохождения одноцепочечной ДНК через пору можно, образуя с ней двухцепочечные участки с помощью комплементарных фрагментов ДНК. Тогда в ходе транспорта будет происходить расплетание данного участка, что и позволит дольше задерживать отдельные нуклеотиды в поре. Тем не менее, поскольку расплетание происходит не понуклеотидно, то время задержки нуклеотида в поре не является постоянным для всей последовательности.
Транспорт с использованием ферментов
Для того, чтобы каждый нуклеотид задерживался в поре на фиксированное время, можно использовать различные ферменты, которые будут пропускать нуклеотиды через пору по одному. Примером такого фермента является ДНК-полимераза. За счёт приложенного напряжения комплекс ДНК-фермент изначально притянут к поре. Но теперь, прежде, чем очередной нуклеотид молекулы ДНК пройдёт через пору, должен произойти один шаг синтеза второй цепи ДНК. Возникающая задержка оснований внутри поры позволяет более точно различать их.
Экзонуклеазное секвенирование
В данном методе цепь нуклеиновой кислоты нарезается на единичные нуклеотиды экзонуклеазой, расположенной в непосредственной близости от поры. Под действием поля отрицательно заряженные нуклеотиды самостоятельно попадают в пору, где происходит определение оснований.
Типы нанопор

Для секвенирования используют белковые нанопоры и синтетические твердотельные нанопоры[20].
Белковые нанопоры
Альфа-гемолизин
Альфа-гемолизин Staphylococcus aureus — это водоростваримый мономер, который в мембране самопроизвольно образует гептамер. Трансмембранный домен состоит из ствола и головки поры. Головка поры содержит полость диаметром около 4,5 нм. В месте соединения ствола и головки находится сужение канала шириной 1,5 нм. Ствол поры состоит из 14 антипараллельных бета-тяжей, формирующих сквозной канал шириной около 2 нм. При нейтральном pH многие аминокислотные остатки в поре заряжены (например, положительно заряженные лизины K147 и негативно заряженные глутаматы E111). В 1М KCl растворе на поре держится потенциал в 120мВ (от ствола к головке), что обуславливает ток 120пА[21]. Внутри ствола находится три сайта распознавания нуклеотидов, что в теории делает возможным распознавание одного нуклеотида более чем одним сайтом (что увеличит точность прочтения)[22]. Время пребывания разных нуклеотидов на этих сайтах разнится, что и позволяет отличать одни основания от других.
MspA
Порин A Mycobacterium smegmatis (Mycobacterium smegmatis porin A, MspA) — нанопора, диаметром 1,2 нм. Обладает структурными особенностями (формой и диаметром поры), которые улучшают соотношение сигнал/шум при секвенировании ДНК по сравнению с альфа-гемолизином[23]. Однако у MspA есть и существенный недостаток: негативно заряженное ядро мешает продвижению одноцепочечной ДНК внутри поры. Поэтому для секвенирования в изначальном белке три негативно заряженные остатка аспартата были заменены на нейтральные остатки аспарагина[24].
Моторный белок упаковки ДНК бактериофага phi29
Этот белок участвует в упаковке ДНК в капсид вирусов, а также в выходе ДНК из капсида при инфицировании. Ключевым отличием от вышеупомянутых белков является то, что имея больший диаметр канала (от 3.6 нм до 6 нм), он способен пропускать двухцепочечную ДНК. Из-за своей природы phi29, в отличие от других пор, в своей исходной форме не встраивается в мембрану, однако эта проблема решается модификацией белка[25]. Другие модификации позволяют белку пропускать одноцепочечную ДНК или одноцепочечную РНК[26].
Твердотельные нанопоры
Помимо белковых нанопор также используются твердотельные нанопоры небиологической природы. Для анализа нуклеиновых кислот используют нанопоры в подложках из кремния, нитрида кремния и полиэтиленимина[27]. Обычно поры формируются пучками ионов или электронов, что позволяет легко варьировать их размер[28]. Отдельно стоит выделить материалы, формирующие очень тонкие — «2D»-поры: графен, дисульфид мольбдена и другие[27]. Графен обладает и предельно маленькой толщиной, что способствует увеличению пространственного разрешения вдоль ДНК, и одновременно с этим прочностью, химической инертностью и электропроводимостью. Эти свойства делают его крайне перспективным материалом в нанопоровом секвенировании[28].
Флуоресцентная детекция
В 2010 году был разработан метод твердотельного наносеквенирования, основанный на детекции флуоресцентного сигнала. Сначала нужная ДНК конвертируется в ДНК, в которой каждому изначальному основанию соответствует короткая последовательность. На эти короткие последовательности гибридизуются флуоресцентные зонды (molecular beacons), при этом конец одного зонда гасит флуоресценцию флуорофора на начале другого зонда. При этом для кодирования четырёх оснований нужно всего два типа зондов: каждому основанию (а точнее соответствующей ему короткой последовательности) соответствует два флуоресцентных сигнала (00, 01, 10 или 11, где 0 соответствует одному цвету, а 1 — другому). При прохождении через пору получившаяся двухцепочечная ДНК расплетается, зонд отделяется, соответственно флуорофор на следующем зонде начинает светиться[29][30].
К преимуществам метода относится точность сигнала — камеры регистрирует сигнал гораздо точнее других имеющихся техник. Однако метод требует предварительной обработки образца: конвертации каждого нуклеотида в примерно 12 нуклеотидов (что также удлиняет саму ДНК)[29].
Сравнение твердотельных и биологических нанопор
Твердотельные нанопоры лишены некоторых недостатков биологических нанопор: чувствительности к pH, температуре, концентрациям электролита, механическим воздействиям и т. д. Помимо этого, они стабильнее, дольше служат, получить разнообразие форм и размеров таких пор значительно проще, а технология производства сходна с производством полупроводников, что сильно облегчает процесс получения таких пор и делает потенциально возможным совмещение с другими наноустройствами. К преимуществам биологических нанопор можно отнести возможность химической или генетической модификации, химическую специфичность к ДНК или РНК и относительно низкую скорость прохождения ДНК или РНК сквозь пору[28][31].
Прочие нанопоры
Для получения нанопор может быть использована технология ДНК-оригами. Впервые такая возможность была продемонстрирована в 2012 году, когда с помощью ДНК-оригами была получена структура, похожая на альфа-гемолизин. Полученная структура самопроизвольно встраивалась в мембраны[27].
В 2010 году было показано, что однослойные углеродные нанотрубки также могут встраиваться в мембраны и пропускать ДНК[27].
На данный момент твердотельные нанопоры не обладают химической специфичностью белков, поэтому активно изучается возможность интеграции белковых нанопор в твердотельных подложках[28].
Другим перспективным направлением является использование твердотельных нанопор с сенсорами (ёмкостными датчиками, туннельными электронными и другими детекторами)[28].
Коммерческое применение
Oxford Nanopore Techonologies
В феврале 2012 года на конференции AGBT во Флориде компания Oxford Nanopore Technologies представила прототипы двух платформ для высокопроизводительного секвенирования длинных фрагментов, основанных на нанопоровом секвенировании целых цепочек: GridION и MinION. В качестве демонстрации был секвенирован геном бактериофага Phi X длиной 5386 оснований[16]. На данный момент компания выпускает несколько устройств. Все они позволяют анализировать данные в реальном времени.
MinION
MinION — секвенатор небольшого размера с одноразовой ячейкой, спроектированный для использования в домашних условиях, с запланированной ценой около 900$. Секвенатор снабжён USB 3.0 разъём для подключения к компьютеру. Содержит 512 нанопор со сходными характеристиками[18]. Ячейка позволяют отсеквенировать до 30 Гб ДНК (примерно за двое суток можно оцифровать 10-20 Гб ДНК)[32]. Хотя формат «карманного секвенатора» и удобен, однако соотношение цены и секвенируемой длины был выбран не самым удачным образом: 30 Гб недостаточно для секвенирования генома человека, а для большинства клинико-диагностических целей это слишком много[33]. Поэтому в 2019 году компания начала выпускать Flongle — адаптор к MinION или GridION, который позволяет работать с менее производительными (~1 Gb, 126 нанопор вместо 512), но существенно более дешёвыми ($90) ячейками[33][34].
GridION
GridION — устройство, спроектированное для полногеномного секвенирования (по сути своей — MinION с увеличенной производительностью). Прототип имел 2000 отдельных нанопор, каждая из которых способна получать риды длиной до 5100 килобаз со скоростью 150 мегабаз/ч в течение 6 часов[18]. GridION Mk1 стоит $49,955 и содержит 5 независимых ячеек. С помощью него за один эксперимент можно отсеквенировать до 150 Гб ДНК[35].
PrometION
Самый высокопроизводительный секвенатор этой компании, позволяет секвенировать за один эксперимент несколько Тб ДНК. PromethION 24 содержит 24 ячейки и способен за трое суток оцифровать 3.8 Тб ДНК, PromethION 48 содержит 48 ячеек и способен за трое суток оцифровать 7.6 Тб ДНК. Ячейки секвенатора содержат 3000 нанопор[33][36]. Поток данных с такого количества нанопор не может анализироваться обычным компьютером, поэтому для использования этого секвенатора необходим суперкомпьютер (впрочем, если запускать только одну ячейку, то справится и обычный компьютер)[33].
Другие разработки
Компания планирует выпустить ещё два устройства: SmidgION — секвенатор, который подключается к смартфону, и Plongle — секвенатор, который содержит 96 независимых, но малопроизводительных ячеек, и, соответственно, рассчитан на частые секвенирования большого объёма коротких ДНК[37].
Пост-обработка данных полученных с помощью Oxford Nanopore
После использования продукции Oxford Nanopore на выходе имеются сырые данные формата FAST5. Формат FAST5[38], используемый Oxford Nanopore — это вариант стандарта HDF5 с иерархической внутренней структурой, предназначенной для хранения метаданных, связанных с последовательностью ДНК и событий (агрегированные измерения общего тока) предварительно обработанных вашим рабочим устройством(прим. MinION). Результаты обработки отображаются в реальном времени в графическом интерфейсе MinKNOW, а данные записываются в формате файлов FASTQ или .fast5. Далее нужно произвести процесс base-calling’а который обработает сырые данные формата FAST5 в FASTQ формат (в программе MinKNOW этот процесс можно запустить во время прочтения ридов). Также можно использовать такие программы как [39]poreTools, Guppy.
Далее нужно очистить полученные последовательности, чтобы избавиться от данных со слишком большим шумом. Для этой задачи используется такая программа как [40]NanoFilt. Как только данные будут очищены полученные данные дальше можно использовать для последующей сборки и анализа данных.
Примечания
- ↑ Thomas P. Niedringhaus, Denitsa Milanova, Matthew B. Kerby, Michael P. Snyder, Annelise E. Barron. Landscape of Next-Generation Sequencing Technologies (англ.) // Analytical Chemistry. — 2011-06-15. — Vol. 83, iss. 12. — P. 4327–4341. — ISSN 1520-6882 0003-2700, 1520-6882. — doi:10.1021/ac2010857.
- ↑ Alexander L. Greninger, Samia N. Naccache, Scot Federman, Guixia Yu, Placide Mbala. Rapid metagenomic identification of viral pathogens in clinical samples by real-time nanopore sequencing analysis (англ.) // Genome Medicine. — 2015-12. — Vol. 7, iss. 1. — P. 99. — ISSN 1756-994X. — doi:10.1186/s13073-015-0220-9.
- ↑ Minh Duc Cao, Devika Ganesamoorthy, Alysha G. Elliott, Huihui Zhang, Matthew A. Cooper. Streaming algorithms for identification of pathogens and antibiotic resistance potential from real-time MinIONTM sequencing (англ.) // GigaScience. — 2016-12. — Vol. 5, iss. 1. — P. 32. — ISSN 2047-217X. — doi:10.1186/s13742-016-0137-2.
- ↑ nanopore-wgs-consortium/NA12878. — 2020-03-01.
- ↑ Human Genome on a MinION (англ.). Oxford Nanopore Technologies (20 октября 2016). Дата обращения: 12 марта 2020.
- ↑ Solanum pennellii (acc. LA5240) - PlabiPD. www.plabipd.de. Дата обращения: 12 марта 2020.
- ↑ Ron Ammar, Tara A. Paton, Dax Torti, Adam Shlien, Gary D. Bader. Long read nanopore sequencing for detection of HLA and CYP2D6 variants and haplotypes (англ.) // F1000Research. — 2015-05-20. — Vol. 4. — P. 17. — ISSN 2046-1402. — doi:10.12688/f1000research.6037.2.
- ↑ Nick Loman. How a small backpack for fast genomic sequencing is helping combat Ebola (англ.). The Conversation. Дата обращения: 12 марта 2020.
- ↑ Characterization of individual polymer molecules based on monomer-interface interactions (англ.). Дата обращения: 12 марта 2020.
- ↑ Kasianowicz, JJ; Brandin E., Branton D., Deamer D. W. Characterization of individual polynucleotide molecules using a membrane channel. (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1996. — 26 November (vol. 93, no. 24). — P. 13770—13773. — doi:10.1073/pnas.93.24.13770. — PMID 8943010. — PMC 19421.
- ↑ Akeson, M.; Branton, D., Kasianowicz, J. J., Brandin, E., and Deamer, D. W. Microsecond time-scale discrimination among polycytidylic acid, polyadenylic acid and polyuridylic acid as homopolymers or as segments within single RNA molecules. (англ.) // Biophys J[англ.] : journal. — 1999. — Vol. 77. — P. 3227—3233. — PMID 10585944.
- ↑ Meller, A.; Nivon, L., Brandin, E., Golovchenko, J., and Branton, D. Rapid nanopore discrimination between single polynucleotide molecules (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2000. — Vol. 97. — P. 1079—1084. — PMC 15527.
- ↑ Howorka, S.; Cheley, S., Bayley, H. Sequence-specific detection of individual DNA strands using engineered nanopores. (англ.) // Nature Biotechnology : journal. — Nature Publishing Group, 2001. — Vol. 19. — P. 636—639. — doi:10.1038/90236.
- ↑ Stoddart, D.; Heron, A., Mikhailova, E., Maglia, G., and Bayley, H. Single nucleotide discrimination in immobilized DNA oligonucleotides with a biological nanopore. (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2009. — Vol. 106. — P. 7702—7707. — doi:10.1073/pnas.0901054106.
- ↑ Oxford Nanopore introduces DNA 'strand sequencing' on the high-throughput GridION platform and presents MinION, a sequencer the size of a USB memory stick (англ.). Oxford Nanopore Technologies (17 февраля 2012). Дата обращения: 12 марта 2020.
- ↑ 1 2 Eisenstein, Michael. Oxford Nanopore announcement sets sequencing sector abuzz (англ.) // Nature Biotechnology : journal. — Nature Publishing Group, 2012. — Vol. 30, no. 04. — P. 295—296. — doi:10.1038/nbt0412-295.
- ↑ 1 2 J. J. Kasianowicz, E. Brandin, D. Branton, D. W. Deamer. Characterization of individual polynucleotide molecules using a membrane channel (англ.) // Proceedings of the National Academy of Sciences. — 1996-11-26. — Vol. 93, iss. 24. — P. 13770–13773. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.93.24.13770.
- ↑ 1 2 3 Maitra, RD; Kim J., Dunbar W. B. Recent advances in nanopore sequencing (неопр.) // Elps. — 2012. — Т. 33, № 23. — С. 3418—3428. — doi:10.1002/elps.201200272. — PMID 23138639.
- ↑ Damla Senol Cali, Jeremie S. Kim, Saugata Ghose, Can Alkan, Onur Mutlu. Nanopore sequencing technology and tools for genome assembly: computational analysis of the current state, bottlenecks and future directions (англ.) // Briefings in Bioinformatics. — 2019-07-19. — Vol. 20, iss. 4. — P. 1542–1559. — doi:10.1093/bib/bby017.
- ↑ 1 2 Wanunu, Meni. Nanopores: A journey towards DNA sequencing (англ.) // Phys Life Rev[англ.] : journal. — 2012. — Vol. 9, no. 2. — P. 125—158. — doi:10.1016/j.plrev.2012.05.010.
- ↑ Jonathan J Nakane, Mark Akeson, Andre Marziali. Nanopore sensors for nucleic acid analysis // Journal of Physics: Condensed Matter. — 2003-08-20. — Т. 15, вып. 32. — С. R1365–R1393. — ISSN 1361-648X 0953-8984, 1361-648X. — doi:10.1088/0953-8984/15/32/203.
- ↑ David Stoddart, Giovanni Maglia, Ellina Mikhailova, Andrew J. Heron, Hagan Bayley. Multiple Base-Recognition Sites in a Biological Nanopore: Two Heads are Better than One (англ.) // Angewandte Chemie International Edition. — 2010-01-12. — Vol. 49, iss. 3. — P. 556–559. — doi:10.1002/anie.200905483.
- ↑ Elizabeth A. Manrao, Ian M. Derrington, Mikhail Pavlenok, Michael Niederweis, Jens H. Gundlach. Nucleotide Discrimination with DNA Immobilized in the MspA Nanopore (англ.) // PLoS ONE / Jerome Mathe. — 2011-10-04. — Vol. 6, iss. 10. — P. e25723. — ISSN 1932-6203. — doi:10.1371/journal.pone.0025723.
- ↑ T. Z. Butler, M. Pavlenok, I. M. Derrington, M. Niederweis, J. H. Gundlach. Single-molecule DNA detection with an engineered MspA protein nanopore (англ.) // Proceedings of the National Academy of Sciences. — 2008-12-30. — Vol. 105, iss. 52. — P. 20647–20652. — ISSN 1091-6490 0027-8424, 1091-6490. — doi:10.1073/pnas.0807514106.
- ↑ David Wendell, Peng Jing, Jia Geng, Varuni Subramaniam, Tae Jin Lee. Translocation of double-stranded DNA through membrane-adapted phi29 motor protein nanopores (англ.) // Nature Nanotechnology. — 2009-11. — Vol. 4, iss. 11. — P. 765–772. — ISSN 1748-3395 1748-3387, 1748-3395. — doi:10.1038/nnano.2009.259.
- ↑ Bing-Yuan Guo, Tao Zeng, Hai-Chen Wu. Recent advances of DNA sequencing via nanopore-based technologies (англ.) // Science Bulletin. — 2015-02. — Vol. 60, iss. 3. — P. 287–295. — doi:10.1007/s11434-014-0707-6.
- ↑ 1 2 3 4 Bing-Yuan Guo, Tao Zeng, Hai-Chen Wu. Recent advances of DNA sequencing via nanopore-based technologies (англ.) // Science Bulletin. — 2015-02. — Vol. 60, iss. 3. — P. 287–295. — doi:10.1007/s11434-014-0707-6.
- ↑ 1 2 3 4 5 Types of nanopores (англ.). Oxford Nanopore Technologies. Дата обращения: 12 марта 2020.
- ↑ 1 2 Ben McNally, Alon Singer, Zhiliang Yu, Yingjie Sun, Zhiping Weng. Optical Recognition of Converted DNA Nucleotides for Single-Molecule DNA Sequencing Using Nanopore Arrays (англ.) // Nano Letters. — 2010-06-09. — Vol. 10, iss. 6. — P. 2237–2244. — ISSN 1530-6992 1530-6984, 1530-6992. — doi:10.1021/nl1012147.
- ↑ Gautam V. Soni, Alon Singer, Zhiliang Yu, Yingjie Sun, Ben McNally. Synchronous optical and electrical detection of biomolecules traversing through solid-state nanopores (англ.) // Review of Scientific Instruments. — 2010-01. — Vol. 81, iss. 1. — P. 014301. — ISSN 1089-7623 0034-6748, 1089-7623. — doi:10.1063/1.3277116.
- ↑ Zewen Liu, Yifan Wang, Tao Deng, Qi Chen. Solid-State Nanopore-Based DNA Sequencing Technology (англ.) // Journal of Nanomaterials. — 2016. — Vol. 2016. — P. 1–13. — ISSN 1687-4129 1687-4110, 1687-4129. — doi:10.1155/2016/5284786.
- ↑ MinION (англ.). Oxford Nanopore Technologies. Дата обращения: 13 апреля 2020.
- ↑ 1 2 3 4 Задачи отечественного секвенаторостроения (июнь, 2019 г.). habr.com. Дата обращения: 13 апреля 2020.
- ↑ Flongle adapter (англ.). Oxford Nanopore Technologies. Дата обращения: 13 апреля 2020.
- ↑ GridION Mk1 (англ.). Oxford Nanopore Technologies. Дата обращения: 13 апреля 2020.
- ↑ PromethION (англ.). Oxford Nanopore Technologies. Дата обращения: 13 апреля 2020.
- ↑ Products (англ.). Oxford Nanopore Technologies. Дата обращения: 13 апреля 2020.
- ↑ Camilla L.C. Ip, Matthew Loose, John R. Tyson, Mariateresa de Cesare, Bonnie L. Brown. MinION Analysis and Reference Consortium: Phase 1 data release and analysis // F1000Research. — 2015-10-15. — Т. 4. — ISSN 2046-1402. — doi:10.12688/f1000research.7201.1.
- ↑ Sarah Goldstein, Lidia Beka, Joerg Graf, Jonathan L. Klassen. Evaluation of strategies for the assembly of diverse bacterial genomes using MinION long-read sequencing (англ.) // BMC Genomics. — 2019-12. — Vol. 20, iss. 1. — P. 23. — ISSN 1471-2164. — doi:10.1186/s12864-018-5381-7.
- ↑ Maria Stein, Erik Brinks, Jana Rathje, Gyu-Sung Cho, Charles M. A. P. Franz. Complete Genome Sequence of Tetracycline-Resistant Serratia liquefaciens S1, Isolated from Mixed Greens, Obtained Using Illumina MiSeq and Oxford Nanopore MinION Sequencing (англ.) // Microbiology Resource Announcements / David A. Baltrus. — 2020-05-07. — Vol. 9, iss. 19. — P. e00156–20, /mra/9/19/MRA.00156–20.atom. — ISSN 2576-098X. — doi:10.1128/MRA.00156-20.
Статья является кандидатом в добротные статьи с 14 апреля 2020. |