Малые ядрышковые РНК: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
Shureg (обсуждение | вклад) оставляю англ текст до завтра |
Shureg (обсуждение | вклад) |
||
| Строка 42: | Строка 42: | ||
The majority of vertebrate snoRNA genes are encoded in the introns of proteins involved in ribosome synthesis or translation, and are synthesized by RNA polymerase II, but snoRNAs can also be transcribed from their own promoters by RNA polymerase II or III. |
The majority of vertebrate snoRNA genes are encoded in the introns of proteins involved in ribosome synthesis or translation, and are synthesized by RNA polymerase II, but snoRNAs can also be transcribed from their own promoters by RNA polymerase II or III. |
||
== Другие функции мякРНК == |
|||
== Other functions of snoRNA == |
|||
| ⚫ | Недавно было обнаружено, что мякРНК имеют функции, не связанные с рРНК. Одна из таких функций состоит в регуляции [[альтернативный сплайсинг|альтернативного сплайсинга]] ''транс''-транскриптов{{термин?}}, осуществляемой мякРНК под названием [[HBII-52]] (SNORD115).<ref name=Kishore> {{cite journal | author = Kishore S, Stamm S | year = 2006| title = The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C | journal = Science | volume = 311| issue = 5758 | pages = 230–231 | pmid = 16357227| doi = 10.1126/science.1118265 }} </ref> |
||
| ⚫ | |||
== Ссылки == |
== Ссылки == |
||
Версия от 03:05, 5 ноября 2008
Эту страницу в данный момент активно редактирует участник Shureg. |
[[Категория:Википедия:Ошибка выражения: неожидаемый оператор <, редактируемые прямо сейчас]]
Малые ядрышковые РНК (мякРНК, англ. snoRNA) — класс малых РНК, участвующих в химических модификациях (метилировании и псевдоуридилировании) рибосомных РНК, а также тРНК и малых ядерных РНК. По классификации MeSH малые ядрышковые РНК считаются подгруппой малых ядерных РНК. мякРНК обычно считаются гидовыми РНК, однако их нельзя путать с гидовыми РНК, направляющими редактирование РНК у трипаносоматид.
Модификации, направляемые мякРНК
После транскрипции свободные молекулы рРНК (называемые пре-рРНК), для того, чтобы превратиться в зрелые рРНК, должны быть подвергнуты серии шагов процессирования. Процессирование включает метилирование и псевдоуридилирование, направляемые мякРНК.
- Метилирование — это присоединение метильных групп к различным субстратам. рРНК человека содержат приблизительно 115 модификаций, являющихся метилированием. Большая часть таких модификаций — метилирование по 2’O атому рибозы.[1]
- Псевдоуридилирование — это изомеризация уридина в псевдоуридин(Ψ). Зрелые рРНК человека содержат около 95 псевдоуридинов.[1]
Каждая молекула мякРНК действует в качестве «направляющей» только для одной-двух модификаций целевой РНК. При этом каждая молекула мякРНК связана по меньшей мере с четырьмя молекулами белка, образуя РНК-белковый комплексы, называемые малыми ядрышковыми рибонуклопротеидами (англ. snoRNP). То, какие белки входят в состав комплекса, зависит от типа мякРНК(см. ниже). Молекула мякРНК содержит последовательность из 10-20 нуклеотидов, комплементарную последовательности, в состав которой входит модифицируемый нуклеотид, что позволяет мякРНК специфично связываться с необходимым участком процессируемой рРНК. После связывания мякРНК с процессируемым сайтом белки, входящие в состав комплекса, осуществляют катализ химической модификации основания.
Семейства мякРНК
Два типа модификаций рРНК (метилирование и псевдоуридилирование) направляются двумя различными семействами мякРНК. These families of snoRNAs are referred to as antisense C/D box and H/ACA box snoRNAs based on the presence of conserved sequence motifs in the snoRNA. There are exceptions but as a general rule C/D box members guide methylation and H/ACA members guide pseudouridylation. The members of each family may vary in biogenesis, structure and function but each family is classified by the following generalised characteristics. For more detail see review [2].
C/D box

C/D box snoRNAs contain two short conserved sequence motifs, C (UGAUGA) and D (CUGA) located near the 5' and 3' ends of the snoRNA respectively. Short regions (~ 5 nucleotides) located upstream of the C box and downstream of the D box are usually base complementary and form a stem-box structure which brings the C and D box motifs into close proximity. This stem-box structure has been shown to be essential for correct snoRNA synthesis and nucleolar localization [3]. Many C/D box snoRNA also contain an additional less well conserved copy of the C and D motifs (referred to as C' and D') located in the central portion of the snoRNA molecule. A conserved region of 10-21 nucleotides upstream of the D box is complementary to the methylation site of the target RNA and enables the snoRNA to form and RNA duplex with the RNA [4]. The nucleotide to be modified in the target RNA is usually located at the 5th position upstream from the D box (or D' box) [5] [6]. Box C/D snoRNAs associate with four evolutionary conserved and essential proteins (Fibrillarin (Nop1p), Nop56p, Nop58p and Snu13) which make up the core C/D box snoRNP [2].
H/ACA box
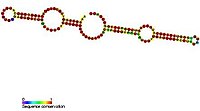
H/ACA box snoRNAs have a common secondary structure consisting of a two hairpins and two single stranded regions termed a hairpin-hinge-hairpin-tail structure [2]. H/ACA snoRNAs also contain conserved sequence motifs known as H box (consensus ANANNA) and the ACA box (ACA). Both motifs are usually located in the single stranded regions of the secondary structure. The H motif is located in the hinge and the ACA motif is located in the tail region, 3 nucleotides from the 3' end of the sequence [7]. The hairpin regions contain internal bulges known as recognition loops in which the antisense guide sequences (bases complementary to the target sequence) are located. This recognition sequence is bipartite (constructed from the two different arms of the loop region) and forms complex pseudo-knots with the target RNA. H/ACA box snoRNAs associate with four evolutionary conserved and essential proteins (dyskerin (Cbf5p), Gar1p, Nhp2p and Nop10p) which make up the core of the H/ACA box snoRNP [2].
Composite H/ACA and C/D box
An unusual guide snoRNA U85 was identified that functions in both 2'-O-ribose methylation and pseudouridylation of small nuclear RNA (snRNA) U5 [8]. This composite snoRNA contains both C/D and H/ACA box domains and associates with the proteins specific to each class of snoRNA (fibrillaring and Gar1p respectively. More composite snoRNAs have now been characterised [9].
These composite snoRNAs have been found to accumulate in a subnuclear organelle called the Cajal body and are referred to as Cajal body specific RNAs. This is in contrast to the majority of C/D box or H/ACA box snoRNAs which localise to the nucleolus. These Cajal body specific RNAs and are proposed to be involved in the modification of RNA polymerase II transcribed spliceosomal RNAs U1, U2, U4, U5 and U12[9]. Not all snoRNAs that have been localised to Cajal bodies are composite C/D and H/ACA box snoRNAs.
snoRNA targets
The targets for newly identified snoRNAs are predicted on the basis of sequence complementarity between putative target RNAs and the antisense elements or recognition loops in the snoRNA sequence. However, there are an increasing number of 'orphan' guides without any known RNA targets, which suggests that there might be more proteins or transcripts involved in rRNA than previously and/or that some snoRNAs have different functions not concerning rRNA.[10]
Target modifications
The precise effect of the methylation and pseudouridylation modifications on the function of the mature RNAs is not yet known. The modifications do not appear to be essential but are known to subtly enhance the RNA folding and interaction with ribosomal proteins. In support of their importance, target site modifications are exclusively located within conserved and functionally important domains of the mature RNA and are commonly conserved amongst distant eukaryotes [2].
- 2'-O-methylated ribose causes an increase in the 3'-endo conformation
- Pseudouridine (psi/Ψ) adds another option for H-bonding.
- Heavily methylated RNA is protected from hydrolysis. rRNA acts as a ribozyme by catalyzing its own hydrolysis and splicing.
Genomic organisation
The majority of vertebrate snoRNA genes are encoded in the introns of proteins involved in ribosome synthesis or translation, and are synthesized by RNA polymerase II, but snoRNAs can also be transcribed from their own promoters by RNA polymerase II or III.
Другие функции мякРНК
Недавно было обнаружено, что мякРНК имеют функции, не связанные с рРНК. Одна из таких функций состоит в регуляции альтернативного сплайсинга транс-транскриптов[неизвестный термин], осуществляемой мякРНК под названием HBII-52 (SNORD115).[11]
Ссылки
- ↑ 1 2 Maden BE, Hughes JM (1997). "Eukaryotic ribosomal RNA: the recent excitement in the nucleotide modification problem". Chromosoma. 105 (7–8): 391—400. PMID 9211966.
- ↑ 1 2 3 4 5 Bachellerie, JP (2002). "The expanding snoRNA world". Biochimie. 84: 775—790. doi:10.1016/S0300-9084(02)01402-5. PMID 12457565.
{{cite journal}}: Неизвестный параметр|coauthors=игнорируется (|author=предлагается) (справка) - ↑ Samarsky, DA (1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". EMBO. 17: 3747—3757. doi:10.1093/emboj/17.13.3747. PMID 9649444.
{{cite journal}}: Неизвестный параметр|coauthors=игнорируется (|author=предлагается) (справка) - ↑ Kiss-László Z, Henry Y, Kiss T (1998). "Sequence and structural elements of methylation guide snoRNAs essential for site-specific ribose methylation of pre-rRNA". EMBO J. 17 (3): 797—807. doi:10.1093/emboj/17.3.797. PMID 9451004.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Cavaillé J, Nicoloso M, Bachellerie JP (1996). "Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides". Nature. 383 (6602): 732—5. doi:10.1038/383732a0. PMID 8878486.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Kiss-László Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M, Kiss T (1996). "Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs". Cell. 85 (7): 1077—88. PMID 8674114.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Ganot P, Caizergues-Ferrer M, Kiss T (1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes Dev. 11 (7): 941—56. PMID 9106664.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Jády BE, Kiss T (2001). "A small nucleolar guide RNA functions both in 2'-O-ribose methylation and pseudouridylation of the U5 spliceosomal RNA". EMBO J. 20 (3): 541—51. doi:10.1093/emboj/20.3.541. PMID 11157760.
- ↑ 1 2 Darzacq X, Jády BE, Verheggen C, Kiss AM, Bertrand E, Kiss T (2002). "Cajal body-specific small nuclear RNAs: a novel class of 2'-O-methylation and pseudouridylation guide RNAs". EMBO J. 21 (11): 2746—56. doi:10.1093/emboj/21.11.2746. PMID 12032087.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Gingeras, Thomas R. (2007). "Origin of phenotypes: Genes and transcripts". Genome Research. 17 (6): 682—690. doi:10.1101/gr.6525007. PMID 17567989.
- ↑ Kishore S, Stamm S (2006). "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C". Science. 311 (5758): 230—231. doi:10.1126/science.1118265. PMID 16357227.
Это заготовка статьи по молекулярной биологии. Помогите Википедии, дополнив её. |