5′-Нетранслируемая область

5′-Нетранслируемая область (5′-НТО, произносится как пять-штрих-нетранслируемая область, англ. 5′-untranslated region, 5′-UTR), или лидерная последовательность[1] — некодирующий участок мРНК, располагающийся сразу после кэпа, но перед кодирующей областью. Такое же название имеет участок ДНК, соответствующий 5′-НТО транскрипта[2]. В 5′-НТО располагаются различные элементы, принимающие участие в регуляции эффективности трансляции[3].
Структура[править | править код]
Длина и нуклеотидный состав[править | править код]
Общая длина 5′-НТО, чаще всего, приблизительно одинакова для всех таксономических групп эукариот и составляет около 100 - 200 нуклеотидов, но может достигать и нескольких тысяч[4][5]. Так, у дрожжей Schizosaccharomyces pombe длина 5′-НТО в транскрипте ste11 составляет 2273 нуклеотида[6][7]. Средняя длина 5′-НТО у человека — около 210 нуклеотидов (в то же время средняя длина 3′-НТО — 800 нуклеотидов[8]). Наиболее длинная известная человеческая 5′-НТО — у онкогена Tre, её длина составляет 2858 нуклеотида, а длина наиболее короткой человеческой 5′-НТО составляет 18 нуклеотидов[1].
Состав оснований также различается в 3′- и 5′-НТО. Так, в 5′-НТО выше содержание G+C, чем в 3′-НТО. Особенно это различие заметно в мРНК теплокровных позвоночных, у которых содержание G+C в 5′-НТО составляет 60 %, а в 3′-НТО — 45 %[9].
Интроны[править | править код]
Внутри участков ДНК, соответствующих 5′-НТО транскрипта, имеются интроны, как и в участках ДНК, соответствующих кодирующей области мРНК. Около 30 % генов Metazoa имеют участки, соответствующие 5′-НТО, состоящие только из экзонов[4]. У человека же около 35 % генов имеют интроны в 5′-НТО. Интроны в 5′-НТО отличаются от таковых в кодирующей области и в 3′-НТО по нуклеотидному составу, длине и плотности[10]. Известно, что отношение общей длины интронов к длине экзонов в 5′-НТО меньше, чем в кодирующей области, однако плотность интронов в 5′-НТО выше (по другим данным, наоборот, ниже[11]), при этом интроны в 5′-НТО приблизительно в два раза длиннее интронов в кодирующей области. В 3′-НТО интроны встречаются значительно реже, чем в 5′-НТО[12].
Эволюция и функции интронов в 5′-НТО остаются в значительной мере неизученными. Тем не менее, установлено, что у активно экспрессируемых генов чаще всего бывают короткие интроны в 5′-НТО, чем длинные или они отсутствуют вовсе. Хотя связи между длиной и количеством интронов и тканью к настоящему моменту не установлено, обнаружена некоторая корреляция между числом интронов в генах и их функциями. Так, особенно много интронов было выявлено в генах, выполняющих регуляторные функции[10]. Вообще, наличие хотя бы одного интрона в 5′-НТО усиливает экспрессию гена, усиливая транскрипцию (в данном случае идёт речь об участке ДНК, соответствующем 5′-НТО транскрипта) или стабилизируя зрелую мРНК. Например, экспрессия гена убиквитина С (UbC) зависит от наличия интрона в 5′-НТО. При утрате интрона активность промотора резко падает, и дальнейшие исследования показали, что в области 5′-НТО ДНК связываются транскрипционные факторы Sp1 и Sp3[11].
Вторичная структура[править | править код]
Структурный и нуклеотидный состав 5′-НТО имеет важное значение для регуляции экспрессии генов; более того, были показаны различия в структуре 5′-НТО мРНК генов «домашнего хозяйства» и генов, задействованных в регуляции онтогенеза. 5′-НТО генов, экспрессия которых сопровождается образованием большого количества белка, как правило, имеют небольшую длину, для них характерно низкое содержание G+C, отсутствие выраженных элементов вторичной структуры и внутренних кодонов AUG (старт-кодонов), расположенных до основного старт-кодона. Напротив, 5′-НТО генов, дающих начало небольшому количеству белка, имеют большую длину, более высокое содержание GC и обладают большим числом характерных элементов вторичной структуры. Высокоструктурированные 5′-НТО нередко присущи мРНК генов, участвующих в регуляции развития; более того, эти образование этих мРНК часто характеризуется тканевой и возрастной специфичностью[13].
Установлено, что в 5′-НТО, оказывающих подавляющее действие на трансляцию, имеются компактные структуры вокруг старт-кодона. Хотя конкретные механизмы такой репрессии неизвестны, считается, что нуклеотидные и структурные особенности 5′-НТО обусловливают связывание с ней различных белковых факторов, активирующих или подавляющих трансляцию[13].

Важными и хорошо изученными элементами вторичной структуры 5′-НТО являются G-квадруплексы. Они образуются тогда, когда последовательности, обогащённые гуанином, сворачиваются в чрезвычайно стабильную неканоническую структуру из четырёх цепей; такие структуры оказывают строго подавляющее действие на трансляцию. Биоинформатический анализ позволил установить, что G-квадруплексы нередко высококонсервативны и имеются в приблизительно 3000 мРНК человека[14]. Примерами таких мРНК человека могут служить мРНК рецептора эстрогена[15], внеклеточной металлопротеиназы[англ.][16], NRAS-протоонкогена[14]. Помимо 5′-НТО, G-квадруплексы обнаружены в промоторах, теломерах и 3′-НТО. Особенно много G-квадруплексов в мРНК белков, участвующих в регуляции трансляции и онтогенеза. Подавляющее действие G-квадруплексов на трансляцию той мРНК, на которой они находятся, может быть обусловлено как их вторичной структурой самой по себе, так и их взаимодействием с белками и другими факторами[17].
Сканирующая модель инициации трансляции предполагает, что малая субъединица рибосомы движется по мРНК («сканирует») в направлении от 5′- к 3′-концу в поисках подходящего старт-кодона AUG и с него начинает трансляцию. При этом также считалось, что наличие стабильных элементов вторичной структуры (например, шпилек) в 5′-НТО оказывает подавляющее действие на трансляцию, поскольку через них рибосома пройти неспособна. Однако недавние исследования показали, что так происходит далеко не всегда. Трансляция мРНК с длинной, высокоструктурированной 5′-НТО может идти не хуже, чем мРНК с короткой и неструктурированной 5′-НТО. Объясняется это тем, что подавляющий эффект самой по себе вторичной структуры часто не выражен, поскольку он определяется прежде всего взаимодействующими с ней белками. Господствовавшая ранее вышеописанная ошибочная точка зрения появилась из-за того, что ранее исследователи использовали систему лизата ретикулоцитов кролика (англ. rabbit reticulocyte lysate (RRL)), и эта система имела ряд недостатков и не соответствовала условиям in vivo[18].
Альтернативные 5′-НТО[править | править код]
Существует несколько механизмов образования альтернативных 5′-НТО при одной и той же кодирующей последовательности:
- использование альтернативных промоторов;
- образование различных 5′-НТО в ходе альтернативного сплайсинга;
- использование альтернативных сайтов начала транскрипции в пределах одного промотора[19].
Наличие различных 5′-НТО в мРНК одного и того же гена даёт дополнительные возможности для регуляции его экспрессии, поскольку даже небольшие различия во вторичной структуре 5′-НТО могут коренным образом повлиять на регуляцию трансляции. Анализ транскриптомов млекопитающих показал, что экспрессия альтернативных 5′-НТО есть распространённый феномен и потенциально большая часть генов может использовать такой механизм регуляции. Белковые продукты генов, постоянно использующих альтернативные 5′-НТО, обычно задействованы в таких процессах, как транскрипция и сигнальные пути. Например, ген рецептора эстрогена β (ERβ) имеет 3 мРНК с альтернативными 5′-НТО, дающими начало изоформам одного и того же белка, и зачастую сбои в их активности наблюдаются при раковых заболеваниях[19].
Функции[править | править код]
Внутри 5′-НТО локализуются важные функциональные элементы, участвующие в инициации трансляции и контроле экспрессии генов. Об этом свидетельствует, во-первых, то, что скорость трансляции не зависит от длины и структуры 5′-НТО как в кэпированных, так и некэпированных мРНК, а также то, что некоторые гены способны экспрессироваться в условиях стресса[20]. К важнейшим из таких функциональных элементов относят участки внутренней посадки рибосомы (IRES), внутренние открытые рамки считывания uORFs, железозависимый элемент (IRE) и др.
IRES[править | править код]
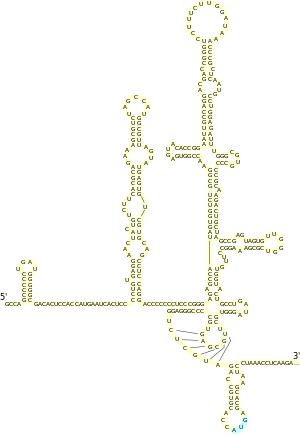
Участок внутренней посадки рибосомы (англ. internal ribosome entry site, IRES) — это регуляторный мотив мРНК, осуществляющий кэп-независимый механизм инициации трансляции, при котором посадка рибосомы происходит внутри 5′-НТО, но рядом с сайтом начала трансляции. Механизм IRES используется как кэпированными, так и некэпированными мРНК в условиях, когда кэп-зависимая инициация трансляции подавлена по причине стресса, в определённой стадии клеточного цикла и при апоптозе, обеспечивая продолжительную экспрессию необходимых белков. Ряд генов, использующих IRES, например, гены c-Myc, APAF1, Bcl-2, при нормальных условиях экспрессируются мало и активируются за счёт IRES в условиях стресса. Предполагается, что IRES может также принимать участие в поддержании в нормальных условиях низкого уровня экспрессии ряда белков, забирая на себя рибосомы и не давая им начать трансляцию с основного сайта инициации. Механизм внутренней инициации трансляции пока плохо понятен, хотя точно известно, что эффективность IRES в значительной мере находится под влиянием транс'′-регуляторных белковых факторов, что даёт возможность для клеточно-специфического использования IRES в трансляции[20].
Структура эукариотических IRES очень различна, и на данный момент не было установлено никаких характерных для них консервативных мотивов. Для некоторых генов для работы IRES необходимы специфичные стабильные элементы вторичной структуры мРНК, у других генов, напротив, они оказывают подавляющее действие на трансляцию. Было высказано предположение, что IRES не являются статичными структурами и подвергаются перемещениям, значительно изменяя свою активность. Элементы IRES могут также давать начало различным изоформам белков, что даёт дополнительные возможности получения различных белковых продуктов с одного и того же гена[21].
uORF[править | править код]
Короткие открытые рамки считывания (англ. upstream open reading frames, uORF) располагаются в 5′-НТО и характеризуются тем, что их внутрирамковый стоп-кодон располагается после внутреннего старт-кодона (англ. upstream AUG, uAUG), но перед основным старт-кодоном, который находится уже в транслируемой (кодирующей) области. uORF обнаружены в приблизительно 50 % 5′-НТО человеческих мРНК, и их наличие обусловливает снижение экспрессии генов, уменьшая количество функциональной мРНК на 30 %, а образование белка — на 30—80 %. Рибосомы, связывающиеся с uAUG, начинают трансляцию uORF, что может отрицательно сказаться на эффективности трансляции основной рамки считывания (то есть кодирующей области). Если не происходит эффективного связывания рибосомы со старт-кодоном в кодирующей области (то есть инициации трансляции), то в результате снижается образование белка, а значит, и уровень экспрессии соответствующего гена. Может произойти и обратная ситуация: трансляция uORF продолжится в трансляцию кодирующей области, и в итоге образуется слишком длинный белок, который может быть вредоносным для организма. Снижение эффективности трансляции из-за наличия uORF в 5′-НТО является хорошо изученным эффектом; одним из примеров, иллюстрирующих его, может служить ген поли(А)-полимеразы α[англ.] (англ. poly(A)-polymerase α, PAPOLA), чья мРНК содержит две высококонсервативные uORF в 5′-НТО. Мутация проксимального uAUG вызывает повышение эффективности трансляции этой мРНК, что свидетельствует о том, что uORF значительно снижает экспрессию этого гена. Другим примером является рецептор тироидного гормона, оказывающий активирующее или репрессирующее действие на транскрипцию ряда генов-мишеней; сильная репрессия его трансляции осуществляется uORF длиной 15 нуклеотидов внутри 5′-НТО его мРНК[22].
Широко распространена точка зрения, что uORF снижают эффективность трансляции, поскольку после терминации трансляции uORF рибосома не может вновь начать трансляцию и транслировать кодирующую область (англ. coding sequence, CDS). Тем не менее, недавние исследования более чем 500 локусов генов, содержащих 5′-НТО, показали, что не существует никакой определённой связи между влиянием uORF на экспрессию нижележащего гена и расстоянием между uORF и кодирующей последовательностью. При этом авторы исследования предполагают, что в генах, содержащих единственную uORF, вероятнее всего, трансляция CDS осуществляется после сканирования uORF рибосомой без её диссоциации, а не через реинициацию трансляции. Это предположение сильно отличается от выводов Козак (1987) и вообще всех представлений о uORF. Более того, эксперименты с клетками, лишёнными Rent1 (фактора, участвующего в процессе направленного разрушения дефектных мРНК — англ. nonsense-mediated decay, NMD), показали, что в отсутствие NMD успешно шла трансляция транскриптов, содержащих uORF. Это показывает, что NMD также играет важную роль в регуляции функционирования этих транскриптов. Вероятнее всего, существует несколько вариантов развития событий после взаимодействия uORF и рибосомы: продолжение трансляции, продолжение сканирования или реинициация трансляции кодирующей области, причём то, какой из них произойдёт, зависит от ряда факторов[22].
Установлено, что, помимо AUG, в качестве сайта начала трансляции могут использоваться также кодоны, отличающиеся от AUG одним нуклеотидом, и эффективность инициации в каждом случае будет определяться окружением нестандартного старт-кодона[23].
Хотя большая часть uORF отрицательно влияет на экспрессию генов, существуют случаи, когда наличие uORF усиливает трансляцию. Примером может служить бицистронная мРНК vpu-env вируса ВИЧ-1, содержащая консервативную очень маленькую uORF. Эта uORF располагается лишь за 5 нуклеотидов до AUG vpu и вскоре заканчивается стоп-кодоном, перекрывающимся с AUG vpu. Было установлено, что эта uORF оказывает значительный положительный эффект на трансляцию env и при этом не мешает трансляции vpu. Были получены мутанты, у которых расстояние между uORF и основным AUG было увеличено на 5 нуклеотидов, и было показано, что uORF не задействована в инициации vpu. На основании этого авторами исследования было высказано предположение, что эта маленькая uORF может служить местом задержки рибосомы, в ходе которой рибосома взаимодействует с РНК-структурами, способствующими её продвижению, то есть физически преодолевает часть 5′-НТО, чтобы достичь основного инициаторного кодона[24].
Помимо вышеперечисленных, известны также следующие механизмы действия uORF:
- нуклеотиды, находящиеся uORF, могут образовывать шпильку, которая препятствует продвижению рибосомы;
- не только цис-, но и транс-регуляция трансляции кодирующей области;
- взаимодействие с IRES[25].
Значение uORF как регуляторных элементов, участвующих в регуляции связывания рибосомы и трансляции, хорошо изучено, однако функция и даже судьба кодируемых uORF пептидов часто неизвестна, возможно, по причине сложностей в анализе уровня экспрессии и локализации пептидов[26].
IRE[править | править код]

В 5′-НТО мРНК белков, связанных с метаболизмом железа, часто имеется особый регуляторный элемент — железозависимый элемент. Он имеется в 5′-НТО мРНК таких белков, как ферритин, трансферриновый рецептор[англ.], эритроидная аминолевулинатсинтаза[англ.], митохондриальная аконитаза, ферропортин[англ.], переносчик двухвалентных металлов[англ.] (англ. divalent metal transporter 1 (DMT1))[27] (впрочем, он встречается и в мРНК белков, не связанных с метаболизмом железа, например, в мРНК белкового продукта гена CDC42BPA — киназы, участвующей в реорганизации цитоскелета[28]). IRE представляет собой шпильку, взаимодействующую с особыми регуляторными белками[англ.] — IRP1 и IRP2 (англ. iron-regulatory proteins). Когда концентрация железа мала, с IRE связываются IRP1 и IRP2, создавая преграды для рибосомы и делая трансляцию мРНК-мишени невозможной[29]. При высокой концентрации железа между этими белками и шпилькой нет жёсткого связывания, и идёт трансляция белков, задействованных в метаболизме железа. Кроме того, установлено, что трансляция белка-предшественника бета-амилоида также контролируется IRE, причём его IRE тоже способен связываться с IRP1 и IRP2, поэтому не исключено, что IRE может играть определённую роль в развитии болезни Альцгеймера[30].
Другие взаимодействия с белками[править | править код]
В начала трансляции у эукариот на 5′-конце транскрипта в области кэпа собирается белковый комплекс eIF4F[англ.], причём две его субъединицы — eIF4E[англ.] и eIF4G[англ.] — присоединяются в области 5′-НТО, ограничивая тем самым скорость, с которой может происходить инициация трансляции[31]. Однако роль 5′-НТО в образовании преинициаторного комплекса этим не ограничивается. В некоторых случаях с 5′-НТО связываются белки, препятствующие сборке инициаторного комплекса. В качестве примера можно рассмотреть регуляцию гена msl-2 (англ. male-specific lethal 2 — мужская специфическая леталь 2), участвующего в определении пола у дрозофилы. С интроном, локализованным в 5′-НТО первичного транскрипта msl-2, связывается белковый продукт гена SXL (англ. sex lethal — половая леталь), в результате чего этот интрон не удаляется в ходе сплайсинга[29]. Он способствует одновременному связыванию с 5′-НТО и 3′-НТО белков, не позволяющих собраться инициаторному комплексу. Впрочем, SXL может подавлять трансляцию мРНК, лишённых поли(А)-хвоста или вообще 3′-НТО[32]. В мРНК орнитиндекарбоксилазы[англ.], участвующей в метаболизме полиаминов[англ.], и мРНК c-myc в 5′-НТО имеются шпилечные структуры, стабилизируемые белком-репрессором, препятствующие посадке на них рибосомы и сборке инициаторного комплекса. Варьирования в количестве белков-репрессоров обусловливают различную степень стабилизации этих шпилек и, соответственно, доступность этих 5′-НТО для инициаторных белков и рибосомы может быть различной[33].
С 5′-НТО некоторых может связываться не только белок-репрессор, препятствующей сборке инициаторного комплекса и посадке рибосомы, но и белки-репрессоры, стабилизирующие различные структурные барьеры на пути сканирующего рибосомного комплекса. Например, трансляционная репрессия мРНК тимидилатсинтазы[англ.] человека осуществляется продуктом её трансляции — тимидилатсинтазы — по принципу отрицательной обратной связи; тимидилатсинтаза взаимодействует с 30-нуклеотидной шпилькой в 5′-НТО, стабилизируя её и препятствуя продвижению рибосомы[34].
Взаимодействие 5′-НТО и 3′-НТО[править | править код]

Известно, что мРНК способна замыкаться в кольцо (циркуляризация) за счёт взаимодействия специальных белков, связывающихся с поли(А)-хвостом, способствующих связыванию фактора eIF4F с кэпом. В результате мРНК приобретает замкнутую форму, стимулируется инициация трансляции и повышается эффективность трансляции. Однако в некоторых случаях связываться друг с другом могут 5′-НТО и 3′-НТО одной и той же мРНК. Так, у мРНК человеческого гена p53 имеются участки в 5′-НТО и 3′-НТО, комплементарные друг другу. Связываясь друг с другом и с трансляционным фактором RPL26[англ.], они тем самым способствуют повышению эффективность трансляции белка р53 в ответ на повреждения ДНК[35].
Анализ мРНК различных генов человека показал, что в 5′-НТО присутствует мотив, специфически взаимодействующий с 3′-концами микроРНК, при этом во многих таких микроРНК на 5′-конце имеется сайт, комплементарный 3′-НТО. Дальнейшие исследования показали, что связывание 5′-НТО и 3′-НТО с одной и той же микроРНК облегчает связь 5′-конца мРНК с 3′-концом, подобно мостику, и мРНК, активность которых значительно определяется микроРНК, имеют предсказуемые сайты связывания на обеих НТО. Такие мРНК получили название miBridge. Далее было установлено, что утрата этих сайтов связывания уменьшала репрессию трансляции транскрипта, управляемую микроРНК. Так было выяснено, что сайты связывания НТО друг с другом необходимы для подавления трансляции мРНК. Это свидетельствует о том, что комплементарное взаимодействие 5′-НТО и 3′-НТО необходимо для точной регуляции экспрессии генов[36].
5′-НТО прокариот и вирусов[править | править код]
Бактерии[править | править код]
В мРНК бактерий также имеются 5′- и 3′-нетранслируемые области[38][39]. Длина 5′-НТО бактерий значительно меньше таковой у эукариот и составляет обычно 3—10 нуклеотидов. Например, длина 5′-НТО транскрипта лактозного оперона Escherichia coli составляет лишь 7 нуклеотидов[40]. В 5′-НТО бактерий локализована последовательность Шайна — Дальгарно (AGGAGG)[41], служащая для связывания рибосомы и отделённая спейсером от старт-кодона AUG. Хотя 5′-НТО бактерий и эукариот различны, было показано, что добавление нуклеотидов СС в спейсер мРНК гена Ner бактериофага Mu, хорошо экспрессирующегося в клетках Escherichia coli и Streptomyces, привело к успешной экспрессии этого гена в клетках ретикулоцитов кролика[42].
Элементы вторичной структуры, локализованные в 5′-НТО, как правило, оказывают подавляющее действие на трансляцию[43]. В частности, именно в 5′-НТО обычно располагаются аттенюаторы[англ.] — элементы оперонов, вызывающие преждевременную терминацию трансляции[44] (наиболее известным примером аттенюации является работа триптофанового оперона).
Кроме того, в 5′-НТО бактерий располагается большая часть рибопереключателей[45] — регуляторных элементов мРНК, способных связываться с малыми молекулами, что приводит к изменению образования белка, кодируемого этой мРНК[46].
Археи[править | править код]
Нетранслируемые области имеются и в мРНК многих архей. В частности, в 5′- и 3′-НТО мРНК метаногенной археи Methanococcus jannaschii[англ.] (как и у других представителей порядков Methanopyrales и Methanococcales) локализован элемент SECIS, ответственный за вставку аминокислоты селеноцистеина в полипептидную цепь[47].
Установлено, что мРНК большинства галоархей, а также Pyrobaculum[англ.] и Sulfolobus[англ.] лишены выраженной 5′-НТО, но у мРНК архей-метаногенов имеются длинные 5′-НТО. В связи с этим предполагается, что механизм инициации трансляции метаногенных архей может быть отличен от такового у остальных представителей этого домена[43][48].
В 5′-НТО архей располагается TPP-рибопереключатель[англ.], связывающийся с тиаминпирофосфатом (TPP) (такие рибопереключатели имеются также у бактерий и эукариот)[49].
Вирусы[править | править код]

У многих вирусов инициация трансляции происходит по кэп-независимому механизму и осуществляется через уже упоминавшиеся элементы IRES, локализованные в 5′-НТО[50]. Например, так происходит у ВИЧ, вирусов гепатита A и С[51]. Такой механизм инициации трансляции удобен тем, что в его случае нет необходимости в сканировании длинного фрагмента 5′-НТО[40].
Клиническое значение[править | править код]
Мутации, затрагивающие 5′-НТО, нередко приводят к появлению различных заболеваний, поскольку они нарушают работу тонкой системы регуляции тех или иных генов. В представленной ниже схеме собраны сведения о мутациях, затрагивающих различные регуляторные элементы 5′-НТО, и развивающихся при этом заболеваниях[1] (следует уточнить, что синдром наследственной гиперферритинемии/катаракты развивается при мутации в IRE[1][52]).

Примечания[править | править код]
- ↑ 1 2 3 4 Sangeeta Chatterjee, Jayanta K. Pal. Role of 5- and 3-untranslated regions of mRNAs in human diseases // Biol. Cell. — 2009. — С. 251—262. — doi:10.1042/BC20080104. (недоступная ссылка)
- ↑ Barrett et. al., 2013, p. 9.
- ↑ Molecular biology glossary: 5′ Untranslated Region (5′ UTR). Дата обращения: 1 июня 2014. Архивировано 5 июня 2014 года.
- ↑ 1 2 Flavio Mignone, Carmela Gissi, Sabino Liuni, Graziano Pesole. Untranslated regions of mRNAs // Genome Biol.. — 2002. — Т. 3, № 3. Архивировано 19 июня 2020 года.
- ↑ Lodish, Havery. Molecular Cell Biology (англ.). — New York, New York: W.H. Freeman and Company[англ.], 2004. — P. 113. — ISBN 0-7167-4366-3.
- ↑ Rhind, Nicholas; Chen, Zehua; Yassour, Moran; Thompson, Dawn A.; Haas, Brian J.; Habib, Naomi; Wapinski, Ilan; Roy, Sushmita; Lin, Michael F.; Heiman, David I.; Young, Sarah K.; Furuya, Kanji; Guo, Yabin; Pidoux, Alison; Chen, Huei Mei; Robbertse, Barbara; Goldberg, Jonathan M.; Aoki, Keita; Bayne, Elizabeth H.; Berlin, Aaron M.; Desjardins, Christopher A.; Dobbs, Edward; Dukaj, Livio; Fan, Lin; Fitzgerald, Michael G.; French, Courtney; Gujja, Sharvari; Hansen, Klavs; Keifenheim, Dan; Levin, Joshua Z. Comparative Functional Genomics of the Fission Yeasts (англ.) // Science : journal. — 2011. — Vol. 332, no. 6032. — P. 930—936. — doi:10.1126/science.1203357. — PMID 21511999. — PMC 3131103.
- ↑ Здесь и далее в разделах «Структура» и «Функции» приводится информация по эукариотическим клеточным 5′-UTR. Данные по 5′-UTR бактерий, архей и вирусов рассматриваются в соответствующем разделе.
- ↑ Mignone, Flavio; Graziano Pesole. mRNA Untranslated Regions (UTRs) (англ.). — 2011. — 15 August. — doi:10.1002/9780470015902.a0005009.pub2.
- ↑ Pesole G, Liuni S, Grillo G, Saccone C. Structural and compositional features of untranslated regions of eukaryotic mRNAs (англ.) // Gene[англ.]. — Elsevier, 1997. — Vol. 205, no. 1—2. — P. 95—102.
- ↑ 1 2 Cenik C., Derti A., Mellor J. C., Berriz G. F., Roth F. P. Genome-wide functional analysis of human 5' untranslated region introns. — 2010. — Т. 11, № 3. — doi:10.1186/gb-2010-11-3-r29. Архивировано 30 октября 2013 года.
- ↑ 1 2 Barrett et. al., 2013, p. 21.
- ↑ Xin Hong, Douglas G. Scofield, Michael Lynch. Intron Size, Abundance, and Distribution within Untranslated Regions of Genes // Molecular Biology and Evolution[англ.]. — Oxford University Press, 2006. — Т. 23, № 12. — С. 2392—2404. — doi:10.1093/molbev/msl11. Архивировано 7 июня 2014 года.
- ↑ 1 2 Barrett et. al., 2013, p. 10.
- ↑ 1 2 Kumari S., Bugaut A., Huppert J. L., Balasubramanian S. An RNA G-quadruplex in the 5' UTR of the NRAS proto-oncogene modulates translation. (англ.) // Nature chemical biology. — 2007. — Vol. 3, no. 4. — P. 218—221. — doi:10.1038/nchembio864. — PMID 17322877.
- ↑ Balkwill G. D., Derecka K., Garner T. P., Hodgman C., Flint A. P., Searle M. S. Repression of translation of human estrogen receptor alpha by G-quadruplex formation. (англ.) // Biochemistry. — 2009. — Vol. 48, no. 48. — P. 11487—11495. — doi:10.1021/bi901420k. — PMID 19860473.
- ↑ Morris M. J., Basu S. An unusually stable G-quadruplex within the 5'-UTR of the MT3 matrix metalloproteinase mRNA represses translation in eukaryotic cells. (англ.) // Biochemistry. — 2009. — Vol. 48, no. 23. — P. 5313—5319. — doi:10.1021/bi900498z. — PMID 19397366.
- ↑ Barrett et. al., 2013, p. 11.
- ↑ Barrett et. al., 2013, p. 12.
- ↑ 1 2 Barrett et. al., 2013, p. 13.
- ↑ 1 2 Barrett et. al., 2013, p. 14.
- ↑ Barrett et. al., 2013, p. 15.
- ↑ 1 2 Barrett et. al., 2013, p. 16.
- ↑ Barrett et. al., 2013, p. 17.
- ↑ Barrett et. al., 2013, p. 17—18.
- ↑ Somers, Joanna; Pöyry, Tuija; Willis, Anne E. A perspective on mammalian upstream open reading frame function (англ.) // The International Journal of Biochemistry & Cell Biology[англ.] : journal. — 2013. — Vol. 45, no. 8. — P. 1690—1700. — doi:10.1016/j.biocel.2013.04.020. — PMID 23624144.
- ↑ Barrett et. al., 2013, p. 18.
- ↑ Paul Piccinelli, Tore Samuelsson. Evolution of the iron-responsive element // RNA. — 2007. — Т. 13, № 7. — С. 952—966. — doi:10.1261/rna.464807.
- ↑ T. Leung, X. Q. Chen, I. Tan, E. Manser & L. Lim. Myotonic dystrophy kinase-related Cdc42-binding kinase acts as a Cdc42 effector in promoting cytoskeletal reorganization (англ.) // Molecular and cellular biology[англ.] : journal. — 1998. — January (vol. 18, no. 1). — P. 130—140. — PMID 9418861. — PMC 121465.
- ↑ 1 2 Araujo, Patricia R.; Yoon, Kihoon; Ko, Daijin; Smith, Andrew D.; Qiao, Mei; Suresh, Uthra; Burns, Suzanne C.; Penalva, Luiz O. F. Before It Gets Started: Regulating Translation at the 5′ UTR (англ.) // Comparative and Functional Genomics : journal. — 2012. — Vol. 2012. — P. 1. — doi:10.1155/2012/475731.
- ↑ Rogers, Jack T.; Bush, Ashley I.; Cho, Hyan-Hee; Smith, Deborah H.; Thomson, Andrew M.; Friedlich, Avi L.; Lahiri, Debomoy K.; Leedman, Peter J.; Huang, Xudong; Cahill, Catherine M. Iron and the translation of the amyloid precursor protein (APP) and ferritin mRNAs: Riboregulation against neural oxidative damage in Alzheimer's disease (англ.) // Biochemical Society Transactions[англ.] : journal. — 2008. — Vol. 36, no. 6. — P. 1282—1287. — doi:10.1042/BST0361282. — PMID 19021541. — PMC 2746665.
- ↑ Kang, Min-Kook; Han, Seung-Jin. Post-transcriptional and post-translational regulation during mouse oocyte maturation (англ.) // BMB Reports : journal. — 2011. — Vol. 44, no. 3. — P. 147—157. — doi:10.5483/BMBRep.2011.44.3.147. — PMID 21429291.
- ↑ Penalva, L. O. F.; Sanchez, L. RNA Binding Protein Sex-Lethal (Sxl) and Control of Drosophila Sex Determination and Dosage Compensation (англ.) // Microbiology and Molecular Biology Reviews[англ.] : journal. — American Society for Microbiology[англ.], 2003. — Vol. 67, no. 3. — P. 343—359. — doi:10.1128/MMBR.67.3.343-359.2003. — PMID 12966139. — PMC 193869.
- ↑ Спирин, 2011, с. 414—415.
- ↑ Спирин, 2011, с. 416.
- ↑ Barrett et. al., 2013, с. 32.
- ↑ Barrett et. al., 2013, с. 32—33.
- ↑ Edwards T. E., Ferré-D'Amaré A. R. Crystal structures of the thi-box riboswitch bound to thiamine pyrophosphate analogs reveal adaptive RNA-small molecule recognition (англ.) // Structure : journal. — 2006. — Vol. 14, no. 9. — P. 1459—1468. — doi:10.1016/j.str.2006.07.008. — PMID 16962976.
- ↑ Льюин Б. Гены. — БИНОМ, 2012. — С. 144. — 896 с. — ISBN 978-5-94774-793-5.
- ↑ Н. В. Равин, С. В. Шестаков. Геном прокариот // Вавиловский журнал генетики и селекции. — 2013. — Т. 17, № 4/2. — С. 972—984. Архивировано 31 мая 2014 года.
- ↑ 1 2 Brown, T.A. Genomes 3 (англ.). — New York, New York: Garland Science Publishing, 2007. — P. 397. — ISBN 0 8153 4138 5.
- ↑ John W. Pelley. Elsevier’s Integrated Review Biochemistry. — 2nd Edition. — 2012. — ISBN 978-0-32307-446-9.
- ↑ A 5′ untranslated region which directs accurate and robust translation by prokaryotic and mammalian ribosomes.
- ↑ 1 2 Jian Zhang. Gene expression in Archaea: Studies of transcriptional promoters, messenger RNA processing, and five prime untranslated regions in Methanocaldococcus jannashchii. — 2009. Архивировано 31 мая 2014 года.
- ↑ Magali Naville, Daniel Gautheret. Transcription attenuation in bacteria: theme and variations // Brief Funct Genomic Proteomic. — 2009. — Т. 8. — С. 482—492. Архивировано 4 июня 2014 года.
- ↑ Riboswitches: A Common RNA Regulatory Element. Дата обращения: 5 июня 2014. Архивировано 31 мая 2014 года.
- ↑ Nudler E., Mironov A. S. The riboswitch control of bacterial metabolism (англ.) // Trends Biochem Sci[англ.] : journal. — 2004. — Vol. 29, no. 1. — P. 11—7. — doi:10.1016/j.tibs.2003.11.004. — PMID 14729327.
- ↑ R. Wilting, S. Schorling, B. C. Persson, A. Bock. Selenoprotein Synthesis in Archaea: Identification of an mRNA Element of Methanococcus jannaschii Probably Directing Selenocysteine Insertion // J. Mol. Biol.. — 1997. — Т. 266. — С. 637—641. Архивировано 23 сентября 2015 года.
- ↑ Brenneis M., Hering O., Lange C., Soppa J. Experimental characterization of Cis-acting elements important for translation and transcription in halophilic archaea // PLoS Genet.. — 2007. — Т. 3, № 12. — doi:10.1371/journal.pgen.0030229.
- ↑ Kosuke Fujishima, Akio Kanai. Diversity, Function, and Processing of Archaeal Non-Coding RNAs // Sakura Y. Kato Archaea: Structure, Habitats and Ecological Significance. — Nova Science Publishers, Inc., 2011. — С. 69—94. — ISBN 978-1-61761-932-8. Архивировано 31 мая 2014 года.
- ↑ Thompson, Sunnie R. Tricks an IRES uses to enslave ribosomes (англ.) // Trends in Microbiology[англ.] : journal. — Cell Press, 2012. — Vol. 20, no. 11. — P. 558—566. — doi:10.1016/j.tim.2012.08.002. — PMID 22944245. — PMC 3479354.
- ↑ Jeffrey S. Kieft. Viral IRES RNA structures and ribosome interactions (англ.) // Trends in Biochemical Sciences. — Cell Press, 2008. — Vol. 33, no. 6. — P. 274—283. — doi:10.1016/j.tibs.2008.04.007. Архивировано 8 мая 2024 года.
- ↑ Barrett et. al., 2013, p. 19.
Литература[править | править код]
- Спирин А. С. Молекулярная биология. Рибосомы и биосинтез белка. — М.: Издательский центр «Академия», 2011. — 496 с. — ISBN 978-5-7695-6668-4.
- Коничев А. С., Севастьянова Г. А. Молекулярная биология. — Издательский центр «Академия», 2012. — 400 с. — ISBN 978-5-7695-9147-1.
- Lucy W. Barrett, Sue Fletcher, Steve D. Wilton. Untranslated Gene Regions and Other Non-coding Elements. — SpringerBriefs in Biochemistry and Molecular Biology, 2013. — 57 p. — ISBN 978-3-0348-0679-4.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |