SARS-CoV-2: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
оформление кода при помощи скрипта |
|||
| Строка 58: | Строка 58: | ||
=== Структурная биология === |
=== Структурная биология === |
||
[[Файл:Virus_penetration_mechanism.jpg |мини| Механизм проникновения вируса в клетку и два способа этому воспрепятствовать: 1. Путем ингибирования (например с помощью [[камостат]]а) [[Фурин (фермент)|фурин подобной протеазы]] [[:en:TMPRSS2|TMPRSS2]] активирующей гликопротеин шипа, и 2. путем ингибирования (например с помощью антител) связывания шипа с клеточным рецептором АПФ2<ref>[https://medicalxpress.com/news/2020-03-sars-coronavirus-humans.html Preventing spread of SARS coronavirus-2 in humans]</ref><ref> Hoffmann, M et al. (2020). SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically-proven protease inhibitor. Cell {{DOI|10.1016/j.cell.2020.02.052}}</ref><ref>Smriti Mallapaty (2020). [https://www.nature.com/articles/d41586-020-00660-x Why does the coronavirus spread so easily between people?]. Nature 579, 183 {{doi|10.1038/d41586-020-00660-x}}</ref><ref> Coutard, B., Valle, C., de Lamballerie, X., Canard, B., Seidah, N. G., & Decroly, E. (2020). The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Research, 104742. {{Doi|10.1016/j.antiviral.2020.104742}}</ref> ]] |
|||
Белковое моделирование, осуществлённое на основе расшифрованного генома вируса, показало, что рецептор-связывающий S-белок вируса может иметь достаточно высокую аффинность к белку человека [[ангиотензинпревращающий фермент 2]] (АПФ2) и использовать его как точку входа в клетку<ref name=":5">{{публикация|статья |язык=en |автор=Xu |автор имя=X. |автор2=Chen |автор2 имя=P. |автор3=Wang |автор3 имя=J. |автор4=Feng |автор4 имя=J. |автор5=H. Zhou, X. Li, W. Zhong, P. Hao |заглавие=Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission |ссылка= http://engine.scichina.com/publisher/scp/journal/SCLS/doi/10.1007/s11427-020-1637-5 |издание=Science China Life Sciences |doi=10.1007/s11427-020-1637-5 |тип=j |pmid=32009228}}</ref>. В конце января 2020 года две группы в Китае и США независимо друг от друга экспериментально показали, что именно АПФ2 является рецептором для вируса SARS-CoV-2<ref name=Letco22Jan2020>{{статья |заглавие=Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV |ссылка= https://www.biorxiv.org/content/10.1101/2020.01.22.915660v1 |издание=BiorXiv |страницы=2020.01.22.915660 |doi=10.1101/2020.01.22.915660 |accessdate=2020-01-24 |язык=en |тип=journal |автор=Letko, Michael; Munster, Vincent |число=22 |месяц=1 |год=2020}}</ref><ref name=Zhou2020>{{статья |заглавие=Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin |ссылка=https://www.biorxiv.org/content/10.1101/2020.01.22.914952v2 |издание=BiorXiv |страницы=2020.01.22.914952 |doi=10.1101/2020.01.22.914952 |accessdate=2020-01-24 |язык=en |тип=journal |автор=Zhou, Peng; Shi, Zheng-Li |год=2020}}</ref><ref name=Gralinski2020>{{публикация|статья |язык=en |автор=Gralinski |автор имя=Lisa E. |автор2=Menachery |автор2 имя=Vineet D. |заглавие=Return of the Coronavirus |подзаголовок=2019-nCoV |издание=Viruses |volume=12 |issue=2 |pages=135 |doi=10.3390/v12020135 |pmid=31991541 |год=2020 |месяц=01 |день=24 |page=E135}}</ref>, так же как и для вируса [[Тяжёлый острый респираторный синдром|SARS-CoV]]{{sfn|European Centre for Disease Prevention and Control|loc=Novel coronavirus (2019-nCoV) infections|p=8}}. |
Белковое моделирование, осуществлённое на основе расшифрованного генома вируса, показало, что рецептор-связывающий S-белок вируса может иметь достаточно высокую аффинность к белку человека [[ангиотензинпревращающий фермент 2]] (АПФ2) и использовать его как точку входа в клетку<ref name=":5">{{публикация|статья |язык=en |автор=Xu |автор имя=X. |автор2=Chen |автор2 имя=P. |автор3=Wang |автор3 имя=J. |автор4=Feng |автор4 имя=J. |автор5=H. Zhou, X. Li, W. Zhong, P. Hao |заглавие=Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission |ссылка= http://engine.scichina.com/publisher/scp/journal/SCLS/doi/10.1007/s11427-020-1637-5 |издание=Science China Life Sciences |doi=10.1007/s11427-020-1637-5 |тип=j |pmid=32009228}}</ref>. В конце января 2020 года две группы в Китае и США независимо друг от друга экспериментально показали, что именно АПФ2 является рецептором для вируса SARS-CoV-2<ref name=Letco22Jan2020>{{статья |заглавие=Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV |ссылка= https://www.biorxiv.org/content/10.1101/2020.01.22.915660v1 |издание=BiorXiv |страницы=2020.01.22.915660 |doi=10.1101/2020.01.22.915660 |accessdate=2020-01-24 |язык=en |тип=journal |автор=Letko, Michael; Munster, Vincent |число=22 |месяц=1 |год=2020}}</ref><ref name=Zhou2020>{{статья |заглавие=Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin |ссылка=https://www.biorxiv.org/content/10.1101/2020.01.22.914952v2 |издание=BiorXiv |страницы=2020.01.22.914952 |doi=10.1101/2020.01.22.914952 |accessdate=2020-01-24 |язык=en |тип=journal |автор=Zhou, Peng; Shi, Zheng-Li |год=2020}}</ref><ref name=Gralinski2020>{{публикация|статья |язык=en |автор=Gralinski |автор имя=Lisa E. |автор2=Menachery |автор2 имя=Vineet D. |заглавие=Return of the Coronavirus |подзаголовок=2019-nCoV |издание=Viruses |volume=12 |issue=2 |pages=135 |doi=10.3390/v12020135 |pmid=31991541 |год=2020 |месяц=01 |день=24 |page=E135}}</ref>, так же как и для вируса [[Тяжёлый острый респираторный синдром|SARS-CoV]]{{sfn|European Centre for Disease Prevention and Control|loc=Novel coronavirus (2019-nCoV) infections|p=8}}. |
||
Версия от 19:41, 14 марта 2020
| SARS-CoV-2 | ||||||||
|---|---|---|---|---|---|---|---|---|
 | ||||||||
| Научная классификация | ||||||||
|
Реалм: Царство: Тип: Класс: Порядок: Подпорядок: Cornidovirineae Семейство: Подсемейство: Род: Подрод: Sarbecovirus Штамм: SARS-CoV-2 |
||||||||
| Международное научное название | ||||||||
| SARS-CoV-2 | ||||||||
| Синонимы | ||||||||
|
||||||||
| Группа по Балтимору | ||||||||
| IV: (+)оцРНК-вирусы | ||||||||
| ||||||||
SARS-CoV-2 (Severe acute respiratory syndrome coronavirus 2)[2], ранее 2019-nCoV (англ. 2019 novel coronavirus, также Wuhan coronavirus, Wuhan seafood market pneumonia virus[3][4]) — новый штамм коронавирусов, выявленный в конце 2019 года и вызывающий опасное инфекционное заболевание — COVID-19. Представляет собой одноцепочечный РНК-содержащий вирус[5][6].
Коронавирусом SARS-CoV-2 обусловлена продолжающаяся пандемия COVID-19. В январе 2020 года Всемирная организация здравоохранения объявила вспышку эпидемии, связанной с SARS-CoV-2, чрезвычайной ситуацией в области здравоохранения международного значения[7], а в марте 2020 года охарактеризовала принявшее мировой масштаб распространение болезни как пандемию[8][9].
Общие сведения
Срок в 14 дней можно считать достаточным сроком для мониторинга и отслеживания предшествующих контактов в случае обнаружения заболевшего[10]. На стадии инкубационного периода выявление заболевших тепловизорами неэффективно[11].
По данным Центров по контролю и профилактике заболеваний США предполагается, что передача инфекции происходит воздушно-капельным путём, но неизвестно, может ли она происходить через прикосновения к предметам или поверхностях, на которых есть вирус, с последующим прикосновениям к рту, носу или глазам[12]. Однако в клинических рекомендациях Министерства здравоохранения Российской Федерации уже заявили, что передаётся как воздушно-капельным (при кашле, чихании и разговоре) и воздушно-пылевым путями, так и контактным[13].
Согласно известным данным о коронавирусах, они не способны долго выживать на различных объектах. В частности, нет какого-либо риска заражения при получении посылок или писем из Китая[14].
История изучения
Штамм SARS-CoV-2 впервые обнаружен в декабре 2019 года в результате анализа нуклеиновой кислоты у пациента с пневмонией[15]. 31 декабря 2019 года Всемирная организация здравоохранения была оповещена о нескольких случаях вирусной пневмонии, вызванной неизвестным патогеном. 7 января 2020 года информация о новом вирусе была подтверждена, а сам вирус был отнесён к коронавирусам[16]. Геном вируса первыми полностью расшифровали службы здравоохранения Китая[17], 10 января его сделали публично доступным[18]. До 12 января 5 геномов были зарегистрированы в базе данных GenBank[19][20], к 26 января их количество выросло до 28[21]. За исключением самого раннего генома, геномы находятся под эмбарго в GISAID. Филогенетический анализ доступен через Nextrain[22]. 20 января 2020 года в китайской провинции Гуандун была подтверждена передача вируса от человека к человеку[23].
Коронавирусы, к которым относится SARS-CoV-2, обычно вызывают простуду, но к этому же семейству относятся опасные вирусы SARS-CoV и MERS-CoV, вызывающие тяжёлый острый респираторный синдром и ближневосточный респираторный синдром соответственно[16]. Коронавирусная инфекция является зооантропонозной, то есть возможна передача от животных к человеку. Выяснено, что источником SARS-CoV были циветы, а MERS-CoV — одногорбые верблюды[24]. Возможно, что и в случае SARS-CoV-2 источником инфекции являются животные — генетический анализ вируса выявил схожесть с коронавирусами, распространёнными среди подковоносых летучих мышей, однако пока достоверно неизвестно, являются ли они изначальным источником инфекции. Сейчас же основным способом распространения вируса является передача от человека к человеку[25].
Учёные из разных стран проанализировали геном вируса и подтверждают тот факт, что вирус имеет естественное природное происхождение. Различные же теории заговоров создают атмосферу страха, слухи и предрассудки, подобные теории осуждаются научным сообществом. Вместе с генеральным директором ВОЗ учёные призывают продвигать научные доказательства вместо дезинформации[26].
Эпидемиология
Репродуктивное число[англ.] по данным Китайского центра по контролю и профилактике заболеваний оценивается между 2 и 3, что соответствует количеству людей, которые заражаются от одного инфицированного[27], одно из исследований оценило среднее значение по состоянию на 22 января 2020 года в 2.2[28]. Другое ранее проведённое исследование показывало диапазон 3,3—5,47[29]. В общем случае, значения данного числа, большие 1, означают, что эпидемия будет распространяться, а уменьшить число помогают меры противодействия распространению инфекции[28].
Распространение вируса

Первые случаи заболевания COVID-19 были зарегистрированы в декабре 2019 года в китайском городе Ухань. Большинство заболевших было связано с местным оптовым рынком морепродуктов Хуанань, где продавались живые животные[30]. На ранних этапах количество заражённых удваивалось примерно каждые 7,5 дней[31]; к середине января 2020 года вирус проник и в другие провинции Китая — этому способствовал статус Ухани как важного транспортного узла и возросшее число поездок в связи с приближающимся китайским Новым годом[32]. Зимой 2019—2020 годов большинство новых случаев заболевания и смертей приходилось на Хубэй — провинцию Китая, центром которой является Ухань; однако уже 26 февраля количество новых случаев COVID-19 за пределами Китая превысило количество заражений в пределах этой страны[33]. В конце января 2020 года Всемирная организация здравоохранения присвоила распространению заболевания статус «чрезвычайной ситуации международного значения»[7], а в марте охарактеризовала его как пандемию[8].
На 12 марта 2020 года заболевание затронуло 125 стран по всему миру; помимо Китая, наибольшее количество новых случаев заболевания пришлось на Италию, Иран и Южную Корею. В общей сложности число заболевших по всему миру превысило 133 тысячи, смертей — 4900[34][35][36]. По состоянию на 13 марта 2020 года, в России отмечено 34[37] случаев заболевания, большая часть приходится на Москву, также случаи заражения отмечены в Санкт-Петербурге, Липецке, Калининграде, Краснодарском Крае и Нижнем Новгороде, основная часть заразившихся привезла вирус из Италии[38][39].
Инфекция
Инфекция может протекать как в форме лёгкой острой респираторной вирусной инфекции[40], так и в тяжёлой форме[41]. Осложнения тяжёлых случаев могут включать пневмонию или дыхательную недостаточность с риском смерти[42][43].
Вирусология
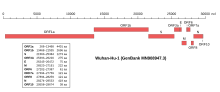
Последовательности бетакоронавируса[англ.] обнаруживают сходство с бетакоронавирусами, выявленными у летучих мышей из Китая. Однако вирус генетически отличается от других коронавирусов, которые вызывают[44]:
- тяжёлый острый респираторный синдром (SARS-CoV), известный также как атипичная пневмония;
- ближневосточный респираторный синдром (MERS-CoV).
SARS-CoV-2, как и SARS-CoV, является членом линии Beta-CoV B[44]. По состоянию на 27 января выделено 26 геномов вируса SARS-CoV-2, в котором уже заметны тенденции к эволюционированию. Как минимум 7 мутаций относятся к одному предку[45].
Длина РНК-последовательности коронавируса составляет около 30 000 нуклеотидов.
РНК варианта Wuhan-Hu-1[19] (номер GenBank MN908947, RefCeq NC_045512[21]) SARS-CoV-2 содержит 29 903 нуклеотида с нетранслируемыми участками длиной 281 и 325 нуклеотидов. Предполагаемые кодирующие области распределены по 10 белкам.

Генетически вирус на 80 % идентичен SARS-CoV[46].
Структурная биология
Белковое моделирование, осуществлённое на основе расшифрованного генома вируса, показало, что рецептор-связывающий S-белок вируса может иметь достаточно высокую аффинность к белку человека ангиотензинпревращающий фермент 2 (АПФ2) и использовать его как точку входа в клетку[51]. В конце января 2020 года две группы в Китае и США независимо друг от друга экспериментально показали, что именно АПФ2 является рецептором для вируса SARS-CoV-2[52][53][54], так же как и для вируса SARS-CoV[55].
Штаммы
С начала вспышки болезни в Китае до марта 2020 года, на основе анализа 103 публично доступных генома SARS-CoV-2 было обнаружено не менее 149 изменений в штамме. Как показало исследование, коронавирус выделился в два подтипа: наиболее часто встречающийся L (70 %) и S (30 %). Подтип L чаще встречался на ранних этапах вспышки в Ухане, однако к началу января 2020 года его частота снизилась. Человеческое вмешательство оказало сильное селективное давление на этот подтип, который может быть более агрессивен и распространяется быстрее. С другой стороны, сравнительная распространённость подтипа S, который эволюционно старее и менее агрессивен, вероятно, возросла в силу более слабого селективного давления.[56]
Иммунитет
Главный внештатный специалист по инфекционным болезням Минздрава России Елена Малинникова на I конгрессе инфекционистов УрФО в Екатеринбурге, заявила, что иммунитет излечившегося от коронавирусной инфекции человека не стойкий, и возможно повторное заражение[57]
Источник вируса
22 января 2020 года медицинский журнал «Journal of Medical Virology[англ.]» опубликовал исследование китайских учёных, в котором пять геномов вируса SARS-CoV-2 сопоставили с 276 известными геномными последовательностями коронавирусов, поражающих людей и различных животных. По мнению учёных, построенное филогенетическое древо коронавирусов показывает, что новые вирусы появились ориентировочно два года назад от одного общего предка, путём гомологичной рекомбинации между коронавирусом летучей мыши и, возможно, коронавирусом китайских змей — южнокитайского многополосного крайта или китайской кобры (обе разновидности змей продавались на рынке в Ухане в качестве экзотической пищи[англ.])[58][59].
Однако ряд исследователей в статье, опубликованной в журнале Nature, оспаривает подобный вывод китайских учёных[60][61] и утверждает, что, с их точки зрения, маловероятно, что в качестве источника заражения могли выступить змеи, где наиболее вероятными кандидатами на эту роль являются млекопитающие и птицы. По мнению Пауло Эдуардо Брандао, вирусолога из Университета Сан-Паулу, китайские учёные не предоставили доказательств того, что змеи могут заражаться новым коронавирусом и служат для него хозяином, в том числе поскольку нет надёжных доказательств наличия коронавирусов у хозяев, кроме млекопитающих и птиц. Цуй Цзе, вирусолог из Шанхайского института Пастера, входивший в группу, выявившую в 2017 году вирусы, связанные с атипичной пневмонией у летучих мышей, сообщает, что полевые работы после вспышки атипичной пневмонии в 2002—2003 годах обнаружили такие вирусы только у млекопитающих[источник не указан 1538 дней].
Другая группа китайских учёных предположила, что источником вируса SARS-CoV-2 являются летучие мыши, поскольку РНК образцов SARS-CoV-2 на 96 % совпала с РНК вируса, который ранее находили у азиатских подковоносов (лат. Rhinolophus affinis). Кроме того, коронавирус SARS-CoV-2 на 79,5 % похож на вирус атипичной пневмонии, эпидемия которой началась в Китае в 2002 году[62]. Исследователи из Южно-Китайского сельскохозяйственного университета в Гуанчжоу считают, что источником нового коронавируса могли стать панголины[63][64].
Примечания
- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ Communicable disease threats report, 9-15 February 2020, week 7. ECDC (10 февраля 2020).
- ↑ Wu et al., 2020.
- ↑ Severe acute respiratory syndrome coronavirus 2 (англ.). Taxonomy browser. NCBI.
- ↑ Surveillance case definitions for human infection with novel coronavirus (nCoV) : Interim guidance v2 : [англ.]. — World Health Organization, 2020. — 15 January. — 1 p. — WHO/2019-nCoV/Surveillance/v2020.2.
- ↑ Novel coronavirus (2019-nCoV), Wuhan, China : [англ.]. — CDC USA, 2020. — 23 January. — Дата обращения: 2020-01-23Коронавирусная инфекция COVID-19.
- ↑ 1 2 Novel Coronavirus(2019-nCoV) Situation Report - 11. World Health Organisation (31 января 2020).
- ↑ 1 2 WHO Director-General’s opening remarks at the media briefing on COVID-19 — 11 March 2020
- ↑ ВОЗ объявила о пандемии коронавируса.
- ↑ European Centre for Disease Prevention and Control, Third update, ECDC risk assessment for the EU/EEA, General assessment, p. 3.
- ↑ Снять «корону»: в РФ создают тест для выявления «китайской» пневмонии.
- ↑ Transmission of Novel Coronavirus (2019-nCoV) (англ.). U. S. CDC (31 января 2020). Дата обращения: 1 февраля 2020.
- ↑ Временные методические рекомендации. Минздрав России (29 января 2020).
- ↑ Novel Coronavirus (2019-nCoV) advice for the public: Myth busters (англ.). World Health Organization. Дата обращения: 30 января 2020.
- ↑ New-type coronavirus causes pneumonia in Wuhan : expert : [англ.] // News. — Xinhua. — Дата обращения: 09.01.2020.
- ↑ 1 2 WHO, Novel Coronavirus (2019-nCoV).
- ↑ CDC, Novel Coronavirus 2019 Situation Summary.
- ↑ European Centre for Disease Prevention and Control, Event background, p. 2.
- ↑ 1 2 Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome (англ.). — 2020-01-23.
- ↑ Novel 2019 coronavirus genome (англ.). Virological (11 января 2020). Дата обращения: 1 февраля 2020.
- ↑ 1 2 2019-nCoV Sequences (Wuhan coronavirus) (англ.). National Center for Biotechnology Information (USA). Дата обращения: 1 февраля 2020.
- ↑ Genomic epidemiology of novel coronavirus (nCoV) using data generated by Fudan University, China CDC, Chinese Academy of Medical Sciences, Chinese Academy of Sciences, Zhejiang Provincial Center for Disease Control and Prevention and the Thai National Institute of Health shared via GISAID. nextstrain.org. Дата обращения: 1 февраля 2020.
- ↑ Associated Press. China confirms human-to-human transmission of new coronavirus (англ.). CBC News (20 января 2020). Дата обращения: 21 января 2020.
- ↑ WHO, Coronavirus.
- ↑ Questions and Answers on 2019novel-Coronavirus. Scientific expertise (англ.). OIE. World Organisation for Animal Health. Дата обращения: 26 января 2020.
- ↑ Charles Calisher, Dennis Carroll, Rita Colwell, Ronald B Corley, Peter Daszak et al. Statement in support of the scientists, public health professionals, and medical professionals of China combatting COVID-19 (англ.) // The Lancet : Correspondence. — Elsevier, 2020. — 18 February. — doi:10.1016/S0140-6736(20)30418-9.
- ↑ European Centre for Disease Prevention and Control, Third update, Novel coronavirus (2019-nCoV) infections, p. 2.
- ↑ 1 2 Qun Li, Xuhua Guan, Peng Wu, Xiaoye Wang, Lei Zhou. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia // New England Journal of Medicine. — 2020-01-29. — Т. 0, вып. 0. — С. null. — ISSN 0028-4793. — doi:10.1056/NEJMoa2001316.
- ↑ Zhao, Shi; Ran, Jinjun; Musa, Salihu Sabiu; Yang, Guangpu; Lou, Yijun; Gao, Daozhou; Yang, Lin; He, Daihai. Preliminary estimation of the basic reproduction number of novel coronavirus (англ.) // biorxiv. — 2019. — 24 January.
- ↑ Novel Coronavirus Pneumonia Emergency Response Epidemiology Team. [The epidemiological characteristics of an outbreak of 2019 novel coronavirus diseases (COVID-19) in China] (кит.) // Zhonghua Liu Xing Bing Xue Za Zhi = Zhonghua Liuxingbingxue Zazhi. — 2020. — 2月 (第41卷, 第2数). — 第145—151 页. — doi:10.3760/cma.j.issn.0254-6450.2020.02.003. — PMID 32064853.
- ↑ Li Q., Guan X., Wu P., Wang X., Zhou L., Tong Y., Ren R., Leung K. S., Lau E. H., Wong J. Y., Xing X., Xiang N., Wu Y., Li C., Chen Q., Li D., Liu T., Zhao J., Li M., Tu W., Chen C., Jin L., Yang R., Wang Q., Zhou S., Wang R., Liu H., Luo Y., Liu Y., Shao G., Li H., Tao Z., Yang Y., Deng Z., Liu B., Ma Z., Zhang Y., Shi G., Lam T. T., Wu J. T., Gao G. F., Cowling B. J., Yang B., Leung G. M., Feng Z. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia (англ.) // The New England Journal of Medicine : journal. — 2020. — January. — doi:10.1056/NEJMoa2001316. — PMID 31995857.

- ↑ WHO-China Joint Mission. Report of the WHO-China Joint Mission on Coronavirus Disease 2019 (COVID-19). World Health Organization (16 февраля 2020). Дата обращения: 8 марта 2020.
- ↑ WHO Director-General's opening remarks at the mission briefing on COVID-19 – 26 February 2020. World Health Organization (26 февраля 2020).
- ↑ Coronavirus Update (Live) for COVID-19 Wuhan China Virus Outbreak – Worldometer. www.worldometers.info.
- ↑ Coronavirus COVID-19 Global Cases by Johns Hopkins CSSE. ArcGIS. Johns Hopkins CSSE (6 марта 2020). Дата обращения: 6 марта 2020.
- ↑ Карта распространения COVID-19.
- ↑ В трех регионах России выявили шесть случаев заражения коронавирусом. 13.03.2020 05:02
- ↑ В России выявили шесть новых случаев коронавируса 06.03.2020 РИА Новости
- ↑ В России выявили четыре новых случая коронавируса РИА Новости 07.03.2020
- ↑ Временные методические рекомендации. Минздрав России. Министерство здравоохранения Российской Федерации (29 января 2020).
- ↑ {{Cite web 2| url=https://www.who.int/publications-detail/clinical-management-of-severe-acute-respiratory-infection-when-novel-coronavirus-(ncov)-infection-is-suspected |title=Clinical management of severe acute respiratory infection when novel coronavirus (nCoV) infection is suspected |date=2020-01-28
- ↑ Symptoms // 2019 Novel Coronavirus, Wuhan, China. — Centers For Disease Control and Prevention (CDC).
- ↑ Novel Coronavirus(2019-nCoV). Situation Report - 8. World Health Organization (20 января 2020).
- ↑ 1 2 Phylogeny of SARS-like betacoronaviruses (англ.). nextstrain. Дата обращения: 18 января 2020.
- ↑ Nextstrain.
- ↑ Redacción Cómo se compara el actual coronavirus con el SARS y qué aprendió China de la mortífera epidemia de hace 17 años (исп.). BBC News Mundo (25 января 2020). Дата обращения: 27 января 2020.
- ↑ Preventing spread of SARS coronavirus-2 in humans
- ↑ Hoffmann, M et al. (2020). SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically-proven protease inhibitor. Cell doi:10.1016/j.cell.2020.02.052
- ↑ Smriti Mallapaty (2020). Why does the coronavirus spread so easily between people?. Nature 579, 183 doi:10.1038/d41586-020-00660-x
- ↑ Coutard, B., Valle, C., de Lamballerie, X., Canard, B., Seidah, N. G., & Decroly, E. (2020). The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Research, 104742. doi:10.1016/j.antiviral.2020.104742
- ↑ Xu, X. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission : [англ.] / X. Xu, P. Chen, J. Wang … [et al.] // Science China Life Sciences : j. — doi:10.1007/s11427-020-1637-5. — PMID 32009228.
- ↑ Letko, Michael; Munster, Vincent. Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV (англ.) // BiorXiv : journal. — 2020. — 22 January. — P. 2020.01.22.915660. — doi:10.1101/2020.01.22.915660.
- ↑ Zhou, Peng; Shi, Zheng-Li. Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin (англ.) // BiorXiv : journal. — 2020. — P. 2020.01.22.914952. — doi:10.1101/2020.01.22.914952.
- ↑ Gralinski, Lisa E. Return of the Coronavirus : 2019-nCoV : [англ.] / Lisa E. Gralinski, Vineet D. Menachery // Viruses. — 2020. — Vol. 12, no. 2 (24 January). — P. 135. — doi:10.3390/v12020135. — PMID 31991541.
- ↑ European Centre for Disease Prevention and Control, Novel coronavirus (2019-nCoV) infections, p. 8.
- ↑ «Китайские ученые рассказали о мутациях коронавируса». ПЕКИН, 4 марта 2020 — РИА Новости.
- ↑ Главный инфекционист Минздрава дала прогноз по коронавирусу/ Lenta.RU, 12 марта 2020
- ↑ Ji et al., 2020.
- ↑ Лосева, Полина Китайский коронавирус оказался гибридом вирусов змеи и летучей мыши. N+1 (23 января 2020).
- ↑ Callaway & Cyranoski, 2020.
- ↑ Megan, Molteni Snakes?! The Slippery Truth of a Flawed Wuhan Virus Theory. One paper advanced a controversial theory about the disease's origin. Other scientists aren't biting. Wired! (23 января 2020).
- ↑ Zhou et al., 2020.
- ↑ Mystery deepens over animal source of coronavirus
- ↑ От кого коронавирус?
Литература
- Wu, F. Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome : GenBank MN908947.3 : [англ.] / F. Wu, Zhao,S., Yu,B. … [et al.] // GenBank : journal. — Bethesda, MD, 2020. — 12 January.
- Ji, W. Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human : [англ.] / W. Ji, W. Wang, X. Zhao … [et al.] // Journal of Medical Virology[англ.]. — Hoboken, New Jersey : Wiley-Blackwell[англ.], 2020. — 22 January. — P. 1—29. — ISSN 1096-9071. — doi:10.1002/jmv.25682. — PMID 31967321.
- Callaway, E. Why snakes probably aren’t spreading the new China virus : One genetic analysis suggests reptilian reservoir — but researchers doubt that the coronavirus could have originated in animals other than birds or mammals : [англ.] / E. Callaway, D. Cyranoski // Nature : журн. — Nature Publishing Group, 2020. — 23 January. — doi:10.1038/d41586-020-00180-8.
- Zhou, Peng. Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin : [preprint] : [англ.] / Peng Zhou, Xing-Lou Yang, Xian-Guang Wang … [et al.] // bioRxiv?! : [сайт]. — 2020. — 23 January. — 18 p. — doi:10.1101/2020.01.22.914952.
- Genomic epidemiology of novel coronavirus (nCoV) // Real-time tracking of pathogen evolution. — Nextstrain. ([Система отслеживания эволюции вируса в реальном времени].)
- Wuhan Coronavirus (2019-nCoV) Global Cases (by JHU CSSE) (англ.). Johns Hopkins University of Medicine. — [Карта зарегистрированных случаев инфицирования 2019-nCoV на сайте Университета Джона Хопкинса].
Ссылки
- Geographical distribution of 2019-nCov cases globally. — European Centre for Disease Prevention and Control (ECDC). (Актуальная информация по глобальному распространению вируса, включая распределение по миру, количество заболевший и смертей.)
- Outbreak of acute respiratory syndrome associated with a novel coronavirus, China; First cases imported in the EU/EEA; second update. — European Centre for Disease Prevention and Control (ECDC), 2020. — 26 January. (Обобщение данных по вирусу от Европейского центра профилактики и контроля заболеваний.)
- Risk assessment: Outbreak of acute respiratory syndrome associated with a novel coronavirus, China: first local transmission in the EU/EEA − third update. — European Centre for Disease Prevention and Control (ECDC), 2020. — 31 January. (Обобщение данных по вирусу от Европейского центра профилактики и контроля заболеваний.)