Ротавирусы
| Ротавирусы | ||||||||
|---|---|---|---|---|---|---|---|---|
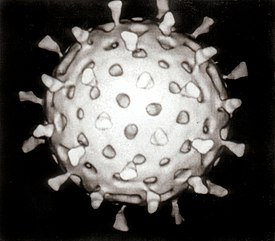 Компьютерная реконструкция ротавируса, основанная на нескольких микрографах | ||||||||
| Научная классификация | ||||||||
|
Реалм: Царство: Тип: Класс: Resentoviricetes Порядок: Reovirales Семейство: Подсемейство: Род: Ротавирусы |
||||||||
| Международное научное название | ||||||||
| Rotavirus | ||||||||
| Виды[2] | ||||||||
|
||||||||
| Группа по Балтимору | ||||||||
| III: дцРНК-вирусы | ||||||||
| ||||||||
Ротавирусы[3] (лат. Rotavirus) — род вирусов с двунитевой сегментированной РНК, принадлежащий к семейству реовирусов (Reoviridae), возбудители ротавирусной инфекции.
Внешний вид частиц напоминает «колесо с широкой ступицей, короткими спицами и чётко очерченным ободком»[4], из-за чего они и получили своё название (от лат. rota — «колесо»).
Виды
[править | править код]Известно 9 видов данного рода, обозначаемых латинскими буквами A—J[2]. Человек может инфицироваться видами A, B и C, при этом возбудителем более 90 % ротавирусных инфекций является наиболее часто встречаемый вид — ротавирус A. Виды с A по D могут вызывать болезни у других животных[5]. К виду Ротавирус A относится несколько серотипов[6]. Как и в случае с вирусом гриппа, здесь применяется двойная классификация по подтипам поверхностных белков: серотипы G определяются вариациями гликопротеина VP7, а серотипы P — протеазочувствительным белком VP4[7]. Поскольку гены, определяющие G- и P-типы, наследуются независимо друг от друга, встречаются различные их комбинации[8].
Структура
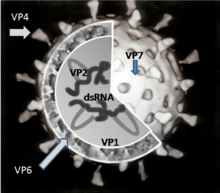
[править | править код]Геном ротавируса состоит из 11 уникальных двунитевых молекул РНК, состоящих в общей сложности из 18 555 нуклеотидов. Нити нумеруются от 1 до 11 в порядке уменьшения длины, каждая представляет собой один ген. Каждый ген кодирует один белок, за исключением гена 9, кодирующего два белка[9]. РНК окружена трёхслойным белковым капсидом в форме усечённого икосаэдра. Каждый из слоёв сложен отдельным вирусным белком. Внутренний и средний слои перфорированы каналами. Средний слой визуально содержит «спицы колеса» (белок VP6) и является важнейшим компонентом вириона.[10] Размер вируса — 76,5 нм в диаметре[11][12], суперкапсида нет[5].
Белки
[править | править код]Вирион сформирован шестью структурными вирусными протеинами (VP), которые обозначаются как VP1, VP2, VP3, VP4, VP6 и VP7. Инфицированная вирусом клетка продуцирует также шесть неструктурных белков (NSP), не являющихся частью вирусной частицы. Они обозначаются: NSP1, NSP2, NSP3, NSP4, NSP5 и NSP6.
По крайней мере шесть из двенадцати кодируемых геномом вируса белков связываются с РНК[13]. Роль этих белков в репликации вируса до конца не выяснена; их функции, как считается, относятся к синтезу РНК и его упаковке в вирион, транспортировке мРНК к месту репликации, трансляции мРНК и регуляции экспрессии генов[14].
Структурные белки
[править | править код]VP1 располагается в ядре вирусной частицы и представляет собой фермент — РНК-полимеразу[15]. В инфицированной клетке фермент осуществляет синтез мРНК для дальнейшего производства вирусных белков, а также синтез сегментов РНК вирусного генома для новых вирионов.
VP2 формирует ядро вируса и связывает вирусный геном[16].
VP3 также составляет ядро вириона и является ферментом гуанилил-трансферазой. Данный фермент катализирует образование 5'-кэп во время процессинга пре-мРНК[17]. Кэп стабилизирует вирусную мРНК, защищая её от утилизации нуклеазами[18].
VP4 расположен на поверхности вириона и выступает с неё в виде шипа[19]. Он связывается с рецепторами на поверхности клеток и управляет внедрением вируса в клетку[20]. Для того, чтобы вирус смог вызывать инфекцию, VP4 должен быть модифицирован находящимся в кишечнике ферментом трипсином в VP5* и VP8*[21]. Именно VP4 определяет вирулентность вируса. VP4 используется для серотипической классификации ротавирусов наряду с VP7.
VP6 формирует толщу капсида. Этот белок крайне антигенен и может использоваться для определения вида ротавируса[22]. Этот белок используется для определения инфекции, вызванной ротавирусом А, в лабораторных тестах[23].
VP7 — структурный гликопротеин, формирующий наружную поверхность вириона. Он определяет G-тип серологической классификации и вместе с VP4 участвует в формировании иммунитета к инфекции[11].
Неструктурные белки
[править | править код]NSP1, продукт гена 5, является неструктурным РНК-связывающим белком[24]. NSP1 также блокирует продукцию интерферона, части врождённой иммунной системы, защищающей клетки от вирусной инфекции. NSP1 вынуждает протеасомы к лизису ключевых сигнальных компонентов, необходимых для стимуляции продукции интерферона в заражённой клетке и для реакции на интерферон, секретируемый соседними клетками. Целями для протеолитической деградации становятся несколько регуляторных факторов интерферона.[25]
NSP2 — РНК-связывающий белок, аккумулируется в цитоплазматических включениях (вироплазмах) и участвует в репликации генома[26][27].
NSP3 связывается с вирусной мРНК в заражённых клетках и отвечает за выключение синтеза клеточных белков[28]. NSP3 инактивирует два фактора инициации трансляции, необходимые для синтеза белков из мРНК клетки-хозяина. Во-первых, NSP3 выталкивает поли(а)-связывающий белок (PABP) из фактора инициации трансляции eIF4F. PABP необходим для эффективной трансляции транскриптов с 3'-хвостами, которые обнаруживаются у большинства транскриптов клетки-хозяина. Во-вторых, NSP3 инактивирует eIF2, стимулируя его фосфорилирование. В то же время эффективная трансляция вирусной мРНК не требует двух этих факторов, поскольку эта РНК не содержит 3'-концов.[29]
NSP4 — вирусный энтеротоксин, вызывающий диарею. Является первым обнаруженным вирусным энтеротоксином[30].
NSP5 кодируется сегментом 11 генома ротавируса А и в инфицированных вирусом клетках накапливается в вироплазмах[31].
NSP6 является белком, связывающим нуклеиновые кислоты[32], кодируется геном 11 по внефазовой открытой рамке считывания[33].
| Сегмент РНК (Ген) | Размер, спаренных оснований | Белок | Молекулярная масса, kDa | Положение | Копий на частицу | Функция |
|---|---|---|---|---|---|---|
| 1 | 3302 | VP1 | 125 | В вершинах ядра | <25 | РНК-зависимая РНК-полимераза |
| 2 | 2690 | VP2 | 102 | Формирует внутреннюю оболочку ядра | 120 | Стимулирует вирусную РНК-репликазу |
| 3 | 2591 | VP3 | 88 | В вершинах ядра | <25 | метилтрансфераза, мРНК-кэпирующий фермент |
| 4 | 2362 | VP4 | 87 | Шипы на поверхности капсида | 120 | Прикрепление к клетке, вирулентность |
| 5 | 1611 | NSP1 | 59 | Неструктурный белок | 0 | Связывание 5’РНК, блокирование продукции интерферона |
| 6 | 1356 | VP6 | 45 | Толща капсида (средний слой) | 780 | Структурная функция; видоспецифичный антиген |
| 7 | 1104 | NSP3 | 37 | Неструктурный белок | 0 | Усиливает активность вирусной мРНК, выключает синтез клеточных белков |
| 8 | 1059 | NSP2 | 35 | Неструктурный белок | 0 | НТФаза, участвует в упаковке РНК |
| 9 | 1062 | VP71 VP72 | 38 и 34 | На поверхности | 780 | Структурный белок; нейтрализирующий антиген |
| 10 | 751 | NSP4 | 20 | Неструктурный белок | 0 | Энтеротоксин |
| 11 | 667 | NSP5 NSP6 | 22 | Неструктурный белок | 0 | оцРНК- и дцРНК-связывающий модулятор активности NSP2, фосфопротеин |
Таблица составлена на основе штамма обезьяньего ротавируса SA11[34][35][36]. Размеры генов у некоторых других штаммов могут отличаться.
Репликация
[править | править код]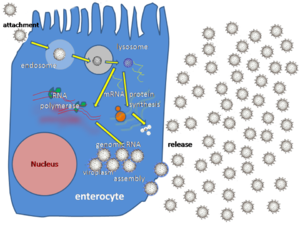
Ротавирусы реплицируются главным образом в кишечнике[37] и заражают энтероциты ворсинок тонкого кишечника, что приводит к структурным и функциональным изменениям эпителия[38]. Тройная белковая оболочка делает их устойчивыми к кислой среде желудка и пищеварительным ферментам в кишечнике.
Существует два возможных пути проникновения вируса в клетку: прямая пенетрация через клеточную мембрану и эндоцитоз. Предполагается, что трансмембранное проникновение опосредовано гидрофобной областью VP5, являющегося продуктом расщепления VP4. Эта область закрыта у нерасщеплённого VP4, поэтому вирионы с белковыми шипами, не подвергшимися расщеплению, не способны проникнуть в клетку этим способом. Второй путь проникновения — эндоцитоз. Вирус проникает в клетку путём опосредованного рецепторами эндоцитоза и образует везикулы, известные как эндосомы. Белки в третьем слое (VP7 и шип VP4) нарушают мембрану эндосомы, создав разницу в концентрации кальция. Это вызывает распад VP7-тримеров на одиночные белковые субъединицы, при этом оставшиеся вокруг вирусной двунитевой РНК белки VP2 и VP6 образуют двухслойную частицу (DLP)[39].
Одиннадцать дцРНК-нитей остаются под защитой двух белковых оболочек, где вирусная РНК-зависимая РНК-полимераза создаёт транскрипты мРНК вирусного генома. Оставаясь в ядре вириона, вирусная РНК избегает врождённого иммунного ответа, называемого РНК-интерференцией и вызываемого присутствием двухцепочечной РНК.
Во время инфекции ротавирус производит мРНК для биосинтеза белка и репликации генов. Большинство ротавирусных белков накапливается в вироплазмах, где реплицируется РНК и собираются DLP. Вироплазмы формируются вокруг ядра клетки уже через два часа после начала вирусной инфекции и состоят из вирусных фабрик, создаваемых, как предполагается, двумя вирусными неструктурными белками: NSP5 и NSP2. Ингибирование NSP5 РНК-интерференцией приводит к резкому снижению репликации ротавирусов. DLP мигрируют в эндоплазматический ретикулум, где они получают свой третий, внешний, слой (образованный VP7 и VP4). Потомство вируса высвобождается из клетки путём лизиса[21][40][41].
Передача
[править | править код]
Ротавирусы передаются фекально-оральным путем, при контакте с загрязненными руками, поверхностями и предметами[42] и, возможно, дыхательным путем[43]. Вирусная диарея очень заразна. Фекалии инфицированного человека могут содержать более 10 триллионов инфекционных частиц на грамм[44]; для передачи инфекции другому человеку требуется менее 100 из них[45].
Ротавирусы стабильны в окружающей среде и были обнаружены в образцах устья реки в количестве до 1-5 инфекционных частиц на галлон США. Вирусы выживают от 9 до 19 дней[46]. Санитарные меры, достаточные для уничтожения бактерий и паразитов, по-видимому, неэффективны в борьбе с ротавирусом, поскольку заболеваемость ротавирусной инфекцией в странах с высокими и низкими стандартами здравоохранения одинакова[43].
Признаки и симптомы
[править | править код]Ротавирусный энтерит — это заболевание от легкой до тяжелой степени, характеризующееся тошнотой, рвотой, водянистой диареей и субфебрильной температурой. Как только ребёнок заражается вирусом, проходит инкубационный период продолжительностью около двух дней, прежде чем появляются симптомы. Период болезни острый. Симптомы часто начинаются с рвоты, за которой следует от четырёх до восьми дней обильной диареи. Обезвоживание чаще встречается при ротавирусной инфекции, чем при большинстве случаев, вызванных бактериальными патогенами, и является наиболее частой причиной смерти, связанной с ротавирусной инфекцией[47].
Ротавирусные инфекции А могут возникать на протяжении всей жизни: первая обычно вызывает симптомы, но последующие инфекции обычно протекают в легкой или бессимптомной форме[48][44], поскольку иммунная система обеспечивает некоторую защиту[49]. Следовательно, частота симптоматических инфекций наиболее высока у детей в возрасте до двух лет и постепенно снижается к 45 годам[50]. Наиболее тяжелые симптомы, как правило, возникают у детей в возрасте от шести месяцев до двух лет, пожилых людей и людей с иммунодефицитом. Из-за иммунитета, приобретенного в детстве, большинство взрослых не восприимчивы к ротавирусу; у гастроэнтерита у взрослых обычно есть причина, отличная от ротавирусной, но бессимптомные инфекции у взрослых могут поддерживать передачу инфекции в сообществе[51]. Есть некоторые свидетельства того, что группа крови может влиять на восприимчивость к заражению ротавирусами[52].
Механизмы заболевания
[править | править код]
Ротавирусы размножаются в основном в кишечнике[53] и поражают энтероциты ворсинок тонкой кишки, что приводит к структурным и функциональным изменениям эпителия[54]. У людей, и особенно на животных моделях, имеются доказательства внекишечного распространения инфекционного вируса в другие органы и макрофаги[55].
Диарея вызывается множественной активностью вируса[56]. Нарушение всасывания происходит из-за разрушения клеток кишечника, называемых энтероцитами. Токсичный белок ротавируса NSP4 индуцирует возрастную и кальций-зависимую секрецию хлорида, нарушает реабсорбцию воды, опосредованную транспортером SGLT1 (котранспортер натрия / глюкозы 2), по-видимому, снижает активность дисахаридаз мембран щеточной каймы и активирует кальций-зависимые секреторные рефлексы кишечной нервной системы[57]. Повышенная концентрация ионов кальция в цитозоле (которые необходимы для сборки вирусов-потомков) достигается за счет NSP4, действующего как виропорин. Это увеличение ионов кальция приводит к аутофагии (саморазрушению) инфицированных энтероцитов[58].
NSP4 также секретируется. Эта внеклеточная форма, которая модифицируется ферментами протеазы в кишечнике, представляет собой энтеротоксин, который действует на неинфицированные клетки через рецепторы интегрина, что, в свою очередь, вызывает и увеличивает внутриклеточную концентрацию ионов кальция, секреторную диарею и аутофагию[59].
Рвота, характерная для ротавирусного энтерита, вызывается вирусом, поражающим энтерохромаффинные клетки на слизистой оболочке пищеварительного тракта. Инфекция стимулирует выработку 5'-гидрокситриптамина (серотонина). Это активирует блуждающие афферентные нервы, которые, в свою очередь, активируют клетки ствола головного мозга, которые контролируют рвотный рефлекс[60].
Здоровые энтероциты выделяют лактазу в тонкий кишечник; непереносимость молока из-за дефицита лактазы является симптомом ротавирусной инфекции[61], которая может сохраняться неделями[62]. Повторение легкой диареи часто следует за повторным введением молока в рацион ребёнка из-за бактериальной ферментации дисахарида лактозы в кишечнике[63].
Иммунные реакции
[править | править код]Конкретные ответы
[править | править код]Ротавирусы вызывают как В-, так и Т-клеточный иммунный ответ. Антитела к белкам ротавируса VP4 и VP7 нейтрализуют вирусную инфекционность in vitro и in vivo[64]. Вырабатываются специфические антитела классов IgM, IgA и IgG, которые, как было показано, защищают от ротавирусной инфекции путем пассивного переноса антител у других животных[65]. Материнский трансплацентарный IgG может играть определённую роль в защите новорожденных от ротавирусных инфекций, но, с другой стороны, может снизить эффективность вакцины[66].
Врожденные реакции
[править | править код]После заражения ротавирусами возникает быстрый врожденный иммунный ответ, включающий интерфероны I и III типов и другие цитокины (в частности, Th1 и Th2[67]), которые ингибируют репликацию вируса и рекрутируют макрофаги и естественные клетки-киллеры в инфицированные ротавирусом клетки[68]. Ротавирусная дцРНК активирует рецепторы распознавания образов, такие как toll-подобные рецепторы[69], которые стимулируют выработку интерферонов. Ротавирусный белок NSP1 противодействует эффектам интерферонов 1-го типа, подавляя активность регуляторных белков интерферона IRF3, IRF5 и IRF7[69].
Маркеры защиты
[править | править код]Уровни IgG и IgA в крови и IgA в кишечнике коррелируют с защитой от инфекции[70]. Было заявлено, что специфичные для ротавируса сывороточные IgG и IgA в высоких титрах (например, > 1: 200) являются защитными, и существует значительная корреляция между титрами IgA и эффективностью ротавирусной вакцины[71].
Диагностика и выявление
[править | править код]Диагноз заражения ротавирусом обычно следует за диагнозом гастроэнтерита как причины тяжелой диареи. Большинство детей, поступивших в больницу с гастроэнтеритом, проходят тестирование на ротавирус А[72][73]. Специфический диагноз заражения ротавирусом А ставится путем обнаружения вируса в кале ребёнка методом иммуноферментного анализа. На рынке имеется несколько лицензированных тест-наборов, которые являются чувствительными, специфичными и обнаруживают все серотипы ротавируса A[74]. Другие методы, такие как электронная микроскопия и ПЦР (полимеразная цепная реакция), используются в исследовательских лабораториях[75]. Полимеразная цепная реакция с обратной транскрипцией (ОТ-ПЦР) позволяет обнаруживать и идентифицировать все виды и серотипы ротавирусов человека[76].
Лечение и прогноз
[править | править код]Лечение острой ротавирусной инфекции неспецифично и включает в себя лечение симптомов и, что наиболее важно, лечение обезвоживания[77]. При отсутствии лечения дети могут умереть от возникающего в результате сильного обезвоживания[78]. В зависимости от тяжести диареи лечение состоит из пероральной регидратационной терапии, во время которой ребёнку дают дополнительную воду для питья, содержащую определённое количество соли и сахара[79]. В 2004 году Всемирная организация здравоохранения (ВОЗ) и ЮНИСЕФ рекомендовали использовать раствор для пероральной регидратации с низкой осмолярностью и прием добавок цинка как двустороннее средство лечения острой диареи[80]. Некоторые инфекции достаточно серьёзны, чтобы потребовать госпитализации, при которой вводятся жидкости путем внутривенной терапии или назогастральной интубации, а также контролируется уровень электролитов и сахара в крови ребёнка[72]. Ротавирусные инфекции редко вызывают другие осложнения, и для хорошо обследованного ребёнка прогноз отличный[81]. Было показано, что пробиотики сокращают продолжительность ротавирусной диареи[82], и согласно Европейскому обществу детской гастроэнтерологии «эффективные вмешательства включают введение специфических пробиотиков, таких как Lactobacillus rhamnosus или Saccharomyces boulardii, диосмектит или рацекадотрил»[83].
См. также
[править | править код]Примечания
[править | править код]- ↑ Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV).
- ↑ 1 2 Таксономия вирусов (англ.) на сайте Международного комитета по таксономии вирусов (ICTV). (Дата обращения: 19 ноября 2021).
- ↑ Атлас по медицинской микробиологии, вирусологии и иммунологии : Учебное пособие для студентов медицинских вузов / Под ред. А. А. Воробьева, А. С. Быкова. — М. : Медицинское информационное агентство, 2003. — С. 117. — ISBN 5-89481-136-8.
- ↑ Грачева Н. М., Аваков А. А., Блохина Т. А., Щербаков И. Т. Клинические аспекты ротавирусной инфекции // Лечащий врач. — 1998. — № 3. — ISSN 1560-5175. Архивировано 28 декабря 2014 года.
- ↑ 1 2 Kirkwood C. D. Genetic and antigenic diversity of human rotaviruses: potential impact on vaccination programs (англ.) // The Journal of Infectious Diseases : journal. — 2010. — September (vol. 202, no. Suppl). — P. S43—S48. — doi:10.1086/653548. — PMID 20684716.
- ↑ O'Ryan M. The ever-changing landscape of rotavirus serotypes (неопр.) // The Pediatric Infectious Disease Journal. — 2009. — March (т. 28, № 3 Suppl). — С. S60—S62. — doi:10.1097/INF.0b013e3181967c29. — PMID 19252426.
- ↑ Patton J.T. Rotavirus diversity and evolution in the post-vaccine world (англ.) // Discovery Medicine : journal. — 2012. — January (vol. 13, no. 68). — P. 85—97. — PMID 22284787. Архивировано 23 сентября 2015 года.
- ↑ Desselberger U., Wolleswinkel-van den Bosch J., Mrukowicz J., Rodrigo C., Giaquinto C., Vesikari T. Rotavirus types in Europe and their significance for vaccination (англ.) // Pediatr. Infect. Dis. J. : journal. — 2006. — Vol. 25, no. 1 Suppl.. — P. S30—S41. — doi:10.1097/01.inf.0000197707.70835.f3. — PMID 16397427. Архивировано 11 мая 2013 года.
- ↑ Desselberger, U.; Gray, James. Rotaviruses: methods and protocols (англ.) / Desselberger, U.; Gray, James. — Totowa, N. J.: Humana Press[англ.], 2000. — P. 2. — ISBN 0-89603-736-3.
- ↑ Carter J., & Saunders V. A. (2007). Virology: principles and applications. John Wiley & Sons; 148—151.
- ↑ 1 2 Pesavento J. B., Crawford S. E., Estes M. K., Prasad B. V. Rotavirus proteins: structure and assembly (англ.) // Curr. Top. Microbiol. Immunol.. — 2006. — Vol. 309. — P. 189—219. — doi:10.1007/3-540-30773-7_7. — PMID 16913048.
- ↑ Prasad B. V., Chiu W. Structure of rotavirus (англ.) // Curr. Top. Microbiol. Immunol.. — 1994. — Vol. 185. — P. 9—29. — PMID 8050286.
- ↑ Patton J. T. Structure and function of the rotavirus RNA-binding proteins (англ.) // Journal of General Virology[англ.] : journal. — Microbiology Society[англ.], 1995. — Vol. 76, no. 11. — P. 2633—2644. — doi:10.1099/0022-1317-76-11-2633. — PMID 7595370. Архивировано 9 декабря 2012 года.
- ↑ Patton J. T. Rotavirus RNA replication and gene expression (неопр.) // Novartis Found. Symp.. — 2001. — Т. 238. — С. 64—77; discussion 77—81. — doi:10.1002/0470846534.ch5. — PMID 11444036.
- ↑ Vásquez-del Carpió R., Morales J. L., Barro M., Ricardo A., Spencer E. Bioinformatic prediction of polymerase elements in the rotavirus VP1 protein (англ.) // Biol. Res. : journal. — 2006. — Vol. 39, no. 4. — P. 649—659. — doi:10.4067/S0716-97602006000500008. — PMID 17657346. Архивировано 1 декабря 2017 года.
- ↑ Arnoldi F., Campagna M., Eichwald C., Desselberger U., Burrone O. R. Interaction of rotavirus polymerase VP1 with nonstructural protein NSP5 is stronger than that with NSP2 (англ.) // J. Virol. : journal. — 2007. — Vol. 81, no. 5. — P. 2128—2137. — doi:10.1128/JVI.01494-06. — PMID 17182692. — PMC 1865955. Архивировано 28 сентября 2011 года.
- ↑ Angel J., Franco M. A., Greenberg H. B. Desk Encyclopedia of Human and Medical Virology (англ.) / Mahy B. W. J., Van Regenmortel M. H. V.. — Boston: Academic Press, 2009. — P. 277. — ISBN 0-12-375147-0.
- ↑ Cowling V. H. Regulation of mRNA cap methylation (англ.) // Biochem. J.. — 2010. — January (vol. 425, no. 2). — P. 295—302. — doi:10.1042/BJ20091352. — PMID 20025612. — PMC 2825737.
- ↑ Gardet A., Breton M., Fontanges P., Trugnan G., Chwetzoff S. Rotavirus spike protein VP4 binds to and remodels actin bundles of the epithelial brush border into actin bodies (англ.) // J. Virol. : journal. — 2006. — Vol. 80, no. 8. — P. 3947—3956. — doi:10.1128/JVI.80.8.3947-3956.2006. — PMID 16571811. — PMC 1440440. Архивировано 28 сентября 2011 года.
- ↑ Arias C. F., Isa P., Guerrero C. A., Méndez E., Zárate S., López T., Espinosa R., Romero P., López S. Molecular biology of rotavirus cell entry (неопр.) // Arch. Med. Res.. — 2002. — Т. 33, № 4. — С. 356—361. — doi:10.1016/S0188-4409(02)00374-0. — PMID 12234525.
- ↑ 1 2 Jayaram H., Estes M. K., Prasad B. V. Emerging themes in rotavirus cell entry, genome organization, transcription and replication (англ.) // Virus Research : journal. — 2004. — April (vol. 101, no. 1). — P. 67—81. — doi:10.1016/j.virusres.2003.12.007. — PMID 15010218.
- ↑ Bishop R. F. Natural history of human rotavirus infection (англ.) // Arch. Virol. Suppl.. — 1996. — Vol. 12. — P. 119—128. — PMID 9015109.
- ↑ Beards G. M., Campbell A. D., Cottrell N. R., Peiris J. S., Rees N., Sanders R. C., Shirley J. A., Wood H. C., Flewett T. H. Enzyme-linked immunosorbent assays based on polyclonal and monoclonal antibodies for rotavirus detection (англ.) // J. Clin. Microbiol. : journal. — 1984. — 1 February (vol. 19, no. 2). — P. 248—254. — PMID 6321549. — PMC 271031. Архивировано 27 сентября 2011 года.
- ↑ Hua J., Mansell E. A., Patton J. T. Comparative analysis of the rotavirus NS53 gene: conservation of basic and cysteine-rich regions in the protein and possible stem-loop structures in the RNA (англ.) // Virology : journal. — 1993. — Vol. 196, no. 1. — P. 372—378. — doi:10.1006/viro.1993.1492. — PMID 8395125.
- ↑ Arnold M.M. The Rotavirus Interferon Antagonist NSP1: Many Targets, Many Questions (англ.) // Journal of Virology : journal. — 2016. — Vol. 90, no. 11. — P. 5212—5215. — doi:10.1128/JVI.03068-15. — PMID 27009959.
- ↑ Kattoura M. D., Chen X., Patton J. T. The rotavirus RNA-binding protein NS35 (NSP2) forms 10S multimers and interacts with the viral RNA polymerase (англ.) // Virology : journal. — 1994. — Vol. 202, no. 2. — P. 803—813. — doi:10.1006/viro.1994.1402. — PMID 8030243.
- ↑ Taraporewala Z. F., Patton J. T. Nonstructural proteins involved in genome packaging and replication of rotaviruses and other members of the Reoviridae (англ.) // Virus Res. : journal. — 2004. — Vol. 101, no. 1. — P. 57—66. — doi:10.1016/j.virusres.2003.12.006. — PMID 15010217.
- ↑ Poncet D., Aponte C., Cohen J. Rotavirus protein NSP3 (NS34) is bound to the 3' end consensus sequence of viral mRNAs in infected cells (англ.) // J. Virol. : journal. — 1993. — 1 June (vol. 67, no. 6). — P. 3159—3165. — PMID 8388495. — PMC 237654. Архивировано 28 сентября 2011 года.
- ↑ López, S; Arias, C.F. Rotavirus-host cell interactions: an arms race (англ.) // Current Opinion in Virology. — Elsevier, 2012. — August (vol. 2, no. 4). — P. 389—398. — doi:10.1016/j.coviro.2012.05.001. — PMID 22658208.
- ↑ Hyser J. M., Estes M. K. Rotavirus vaccines and pathogenesis: 2008 (англ.) // Current Opinion in Gastroenterology. — Lippincott Williams & Wilkins[англ.], 2009. — January (vol. 25, no. 1). — P. 36—43. — doi:10.1097/MOG.0b013e328317c897. — PMID 19114772. — PMC 2673536. Архивировано 11 мая 2013 года.
- ↑ Afrikanova I., Miozzo M. C., Giambiagi S., Burrone O. Phosphorylation generates different forms of rotavirus NSP5 (англ.) // Journal of General Virology[англ.] : journal. — Microbiology Society[англ.], 1996. — Vol. 77, no. 9. — P. 2059—2065. — doi:10.1099/0022-1317-77-9-2059. — PMID 8811003. Архивировано 26 мая 2012 года.
- ↑ Rainsford E. W., McCrae M. A. Characterization of the NSP6 protein product of rotavirus gene 11 (англ.) // Virus Res. : journal. — 2007. — Vol. 130, no. 1—2. — P. 193—201. — doi:10.1016/j.virusres.2007.06.011. — PMID 17658646.
- ↑ Mohan K. V., Atreya C. D. Nucleotide sequence analysis of rotavirus gene 11 from two tissue culture-adapted ATCC strains, RRV and Wa (англ.) // Virus Genes : journal. — 2001. — Vol. 23, no. 3. — P. 321—329. — doi:10.1023/A:1012577407824. — PMID 11778700.
- ↑ Desselberger U. Rotavirus: basic facts. In Rotaviruses Methods and Protocols. Ed. Gray, J. and Desselberger U. Humana Press, 2000, pp. 1—8. ISBN 0-89603-736-3
- ↑ Patton J. T. Rotavirus RNA replication and gene expression. In Novartis Foundation. Gastroenteritis Viruses, Humana Press, 2001, pp. 64—81. ISBN 0-471-49663-4
- ↑ Claude M. Fauquet; J. Maniloff; Desselberger, U. Virus taxonomy: classification and nomenclature of viruses: 8th report of the International Committee on Taxonomy of Viruses (англ.). — Amsterdam: Elsevier/Academic Press, 2005. — P. 489. — ISBN 0-12-249951-4.
- ↑ Greenberg H. B., Estes M. K. Rotaviruses: from pathogenesis to vaccination (неопр.) // Gastroenterology. — 2009. — May (т. 136, № 6). — С. 1939—1951. — doi:10.1053/j.gastro.2009.02.076. — PMID 19457420.
- ↑ Greenberg H. B., Clark H. F., Offit P. A. Rotavirus pathology and pathophysiology (англ.) // Curr. Top. Microbiol. Immunol.. — 1994. — Vol. 185. — P. 255—283. — PMID 8050281.
- ↑ Baker M., Prasad B. V. Rotavirus cell entry (англ.) // Current Topics in Microbiology and Immunology. — 2010. — Vol. 343. — P. 121—148. — doi:10.1007/82_2010_34. — PMID 20397068.
- ↑ Patton J. T., Vasquez-Del Carpio R., Spencer E. Replication and transcription of the rotavirus genome (англ.) // Curr. Pharm. Des. : journal. — 2004. — Vol. 10, no. 30. — P. 3769—3777. — doi:10.2174/1381612043382620. — PMID 15579070.
- ↑ Ruiz M. C., Leon T., Diaz Y., Michelangeli F. Molecular biology of rotavirus entry and replication (англ.) // TheScientificWorldJournal : journal. — 2009. — Vol. 9. — P. 1476—1497. — doi:10.1100/tsw.2009.158. — PMID 20024520.
- ↑ Butz AM, Fosarelli P, Dick J, Cusack T, Yolken R (1993). "Prevalence of rotavirus on high-risk fomites in day-care facilities". Pediatrics. 92 (2): 202—205. doi:10.1542/peds.92.2.202. PMID 8393172. S2CID 20327842.
- ↑ 1 2 Dennehy PH (2000). "Transmission of rotavirus and other enteric pathogens in the home". Pediatric Infectious Disease Journal. 19 (Suppl 10): S103—105. doi:10.1097/00006454-200010001-00003. PMID 11052397. S2CID 28625697.
- ↑ 1 2 Bishop RF. Natural history of human rotavirus infection // Viral Gastroenteritis. — 1996. — Vol. 12. — P. 119–128. — ISBN 978-3-211-82875-5. — doi:10.1007/978-3-7091-6553-9_14.
- ↑ Grimwood K, Lambert SB (2009). "Rotavirus vaccines: opportunities and challenges". Human Vaccines. 5 (2): 57—69. doi:10.4161/hv.5.2.6924. PMID 18838873. S2CID 31164630. Архивировано 15 сентября 2019. Дата обращения: 17 сентября 2022.
- ↑ Rao VC, Seidel KM, Goyal SM, Metcalf TG, Melnick JL (1984). "Isolation of enteroviruses from water, suspended solids, and sediments from Galveston Bay: survival of poliovirus and rotavirus adsorbed to sediments" (PDF). Applied and Environmental Microbiology. 48 (2): 404—409. Bibcode:1984ApEnM..48..404R. doi:10.1128/AEM.48.2.404-409.1984. PMC 241526. PMID 6091548. Архивировано 27 сентября 2011. Дата обращения: 17 сентября 2022.
- ↑ Maldonado YA, Yolken RH (1990). "Rotavirus". Baillière's Clinical Gastroenterology. 4 (3): 609—625. doi:10.1016/0950-3528(90)90052-I. PMID 1962726.
- ↑ Glass RI, Parashar UD, Bresee JS, Turcios R, Fischer TK, Widdowson MA, Jiang B, Gentsch JR (2006). "Rotavirus vaccines: current prospects and future challenges". The Lancet. 368 (9532): 323—332. doi:10.1016/S0140-6736(06)68815-6. PMID 16860702. S2CID 34569166.
- ↑ Offit PA. Gastroenteritis viruses. — New York : Wiley, 2001. — P. 106–124. — ISBN 978-0-471-49663-2.
- ↑ Epidemiology of Group A Rotaviruses: Surveillance and Burden of Disease Studies // Rotaviruses: Methods and Protocols. — Totowa, NJ : Humana Press, 2000. — Vol. 34. — P. 217–238. — ISBN 978-0-89603-736-6. — doi:10.1385/1-59259-078-0:217.
- ↑ Anderson EJ, Weber SG (2004). "Rotavirus infection in adults". The Lancet Infectious Diseases. 4 (2): 91—99. doi:10.1016/S1473-3099(04)00928-4. PMC 7106507. PMID 14871633.
- ↑ Elhabyan A, Elyaacoub S, Sanad E, Abukhadra A, Elhabyan A, Dinu V (November 2020). "The role of host genetics in susceptibility to severe viral infections in humans and insights into host genetics of severe COVID-19: A systematic review". Virus Research. 289: 198163. doi:10.1016/j.virusres.2020.198163. PMC 7480444. PMID 32918943.
- ↑ Greenberg HB, Estes MK (2009). "Rotaviruses: from pathogenesis to vaccination". Gastroenterology. 136 (6): 1939—1951. doi:10.1053/j.gastro.2009.02.076. PMC 3690811. PMID 19457420.
- ↑ Rotavirus pathology and pathophysiology // Rotaviruses. — New York : Springer, 1994. — Vol. 185. — P. 255–283. — ISBN 9783540567615. — doi:10.1007/978-3-642-78256-5_9.
- ↑ Crawford SE, Patel DG, Cheng E, Berkova Z, Hyser JM, Ciarlet M, Finegold MJ, Conner ME, Estes MK (2006). "Rotavirus viremia and extraintestinal viral infection in the neonatal rat model". Journal of Virology. 80 (10): 4820—4832. doi:10.1128/JVI.80.10.4820-4832.2006. PMC 1472071. PMID 16641274.
- ↑ Ramig RF (2004). "Pathogenesis of intestinal and systemic rotavirus infection". Journal of Virology. 78 (19): 10213—10220. doi:10.1128/JVI.78.19.10213-10220.2004. PMC 516399. PMID 15367586.
- ↑ Hyser JM, Estes MK (2009). "Rotavirus vaccines and pathogenesis: 2008". Current Opinion in Gastroenterology. 25 (1): 36—43. doi:10.1097/MOG.0b013e328317c897. PMC 2673536. PMID 19114772.
- ↑ Hyser JM, Collinson-Pautz MR, Utama B, Estes MK (2010). "Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity". mBio. 1 (5). doi:10.1128/mBio.00265-10. PMC 2999940. PMID 21151776.
- ↑ Berkova Z, Crawford SE, Trugnan G, Yoshimori T, Morris AP, Estes MK (2006). "Rotavirus NSP4 induces a novel vesicular compartment regulated by calcium and associated with viroplasms". Journal of Virology. 80 (12): 6061—6071. doi:10.1128/JVI.02167-05. PMC 1472611. PMID 16731945.
- ↑ Hagbom M, Sharma S, Lundgren O, Svensson L (2012). "Towards a human rotavirus disease model". Current Opinion in Virology. 2 (4): 408—418. doi:10.1016/j.coviro.2012.05.006. PMID 22722079.
- ↑ Farnworth ER (2008). "The evidence to support health claims for probiotics". The Journal of Nutrition. 138 (6): 1250S—1254S. doi:10.1093/jn/138.6.1250S. PMID 18492865.
- ↑ Ouwehand A, Vesterlund S (2003). "Health aspects of probiotics". IDrugs: The Investigational Drugs Journal. 6 (6): 573—580. PMID 12811680.
- ↑ Arya SC (1984). "Rotaviral infection and intestinal lactase level". Journal of Infectious Diseases. 150 (5): 791. doi:10.1093/infdis/150.5.791. PMID 6436397.
- ↑ Ward R (2009). "Mechanisms of protection against rotavirus infection and disease". The Pediatric Infectious Disease Journal. 28 (Suppl 3): S57—S59. doi:10.1097/INF.0b013e3181967c16. PMID 19252425.
- ↑ Vega CG, Bok M, Vlasova AN, Chattha KS, Fernández FM, Wigdorovitz A, Parreño VG, Saif LJ (2012). "IgY antibodies protect against human Rotavirus induced diarrhea in the neonatal gnotobiotic piglet disease model". PLOS ONE. 7 (8): e42788. Bibcode:2012PLoSO...742788V. doi:10.1371/journal.pone.0042788. PMC 3411843. PMID 22880110.
- ↑ Mwila K, Chilengi R, Simuyandi M, Permar SR, Becker-Dreps S (2017). "Contribution of Maternal Immunity to Decreased Rotavirus Vaccine Performance in Low- and Middle-Income Countries". Clinical and Vaccine Immunology. 24 (1). doi:10.1128/CVI.00405-16. PMC 5216432. PMID 27847365.
- ↑ Gandhi GR, Santos VS, Denadai M, da Silva Calisto VK, de Souza Siqueira Quintans J, de Oliveira e Silva AM, de Souza Araújo AA, Narain N, Cuevas LE, Júnior LJ, Gurgel RQ (2017). "Cytokines in the management of rotavirus infection: A systematic review of in vivo studies". Cytokine. 96: 152—160. doi:10.1016/j.cyto.2017.04.013. PMID 28414969. S2CID 3568330. Архивировано 6 августа 2020. Дата обращения: 17 сентября 2022.
- ↑ Holloway G, Coulson BS (2013). "Innate cellular responses to rotavirus infection". The Journal of General Virology. 94 (6): 1151—1160. doi:10.1099/vir.0.051276-0. PMID 23486667.
- ↑ 1 2 Villena J, Vizoso-Pinto MG, Kitazawa H (2016). "Intestinal Innate Antiviral Immunity and Immunobiotics: Beneficial Effects against Rotavirus Infection". Frontiers in Immunology. 7: 563. doi:10.3389/fimmu.2016.00563. PMC 5136547. PMID 27994593.
- ↑ Rotaviruses: immunological determinants of protection against infection and disease // Advances in Virus Research Volume 44. — 1994. — Vol. 44. — P. 161–202. — ISBN 9780120398447. — doi:10.1016/S0065-3527(08)60329-2.
- ↑ Patel M, Glass RI, Jiang B, Santosham M, Lopman B, Parashar U (2013). "A systematic review of anti-rotavirus serum IgA antibody titer as a potential correlate of rotavirus vaccine efficacy". The Journal of Infectious Diseases. 208 (2): 284—294. doi:10.1093/infdis/jit166. PMID 23596320.
- ↑ 1 2 Patel MM, Tate JE, Selvarangan R, Daskalaki I, Jackson MA, Curns AT, Coffin S, Watson B, Hodinka R, Glass RI, Parashar UD (2007). "Routine laboratory testing data for surveillance of rotavirus hospitalizations to evaluate the impact of vaccination". The Pediatric Infectious Disease Journal. 26 (10): 914—919. doi:10.1097/INF.0b013e31812e52fd. PMID 17901797. S2CID 10992309.
- ↑ The Pediatric ROTavirus European CommitTee (PROTECT) (2006). "The paediatric burden of rotavirus disease in Europe". Epidemiology and Infection. 134 (5): 908—916. doi:10.1017/S0950268806006091. PMC 2870494. PMID 16650331.
- ↑ Desk Encyclopedia of Human and Medical Virology. — Boston : Academic Press, 2009. — P. 278. — ISBN 978-0-12-375147-8.
- ↑ Gastroenteritis viruses. — New York : Wiley, 2001. — P. 14. — ISBN 978-0-471-49663-2.
- ↑ Fischer TK, Gentsch JR (2004). "Rotavirus typing methods and algorithms". Reviews in Medical Virology. 14 (2): 71—82. doi:10.1002/rmv.411. PMC 7169166. PMID 15027000. Архивировано 21 апреля 2022. Дата обращения: 17 сентября 2022.
- ↑ Diggle L (2007). "Rotavirus diarrhea and future prospects for prevention". British Journal of Nursing. 16 (16): 970—974. doi:10.12968/bjon.2007.16.16.27074. PMID 18026034.
- ↑ Alam NH, Ashraf H (2003). "Treatment of infectious diarrhea in children". Paediatric Drugs. 5 (3): 151—165. doi:10.2165/00128072-200305030-00002. PMID 12608880. S2CID 26076784.
- ↑ Sachdev HP (1996). "Oral rehydration therapy". Journal of the Indian Medical Association. 94 (8): 298—305. PMID 8855579.
- ↑ World Health Organization, UNICEF. Joint Statement: Clinical Management of Acute Diarrhoea. Дата обращения: 3 мая 2012. Архивировано 25 апреля 2012 года.
- ↑ Ramig RF (2007). "Systemic rotavirus infection". Expert Review of Anti-infective Therapy. 5 (4): 591—612. doi:10.1586/14787210.5.4.591. PMID 17678424. S2CID 27763488.
- ↑ Ahmadi E, Alizadeh-Navaei R, Rezai MS (2015). "Efficacy of probiotic use in acute rotavirus diarrhea in children: A systematic review and meta-analysis". Caspian Journal of Internal Medicine. 6 (4): 187—195. PMC 4649266. PMID 26644891.
- ↑ Guarino A, Ashkenazi S, Gendrel D, Lo Vecchio A, Shamir R, Szajewska H (2014). "European Society for Pediatric Gastroenterology, Hepatology, and Nutrition/European Society for Pediatric Infectious Diseases evidence-based guidelines for the management of acute gastroenteritis in children in Europe: update 2014". Journal of Pediatric Gastroenterology and Nutrition. 59 (1): 132—152. doi:10.1097/MPG.0000000000000375. PMID 24739189. S2CID 4845135.