Альтернативный сплайсинг: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
Sirozha (обсуждение | вклад) Отклонено последнее 1 изменение (Iaa.aka) |
Minina (обсуждение | вклад) Нет описания правки |
||
| Строка 1: | Строка 1: | ||
[[Файл:DNA alternative splicing.gif|450px|right|thumb|Благодаря альтернативному сплайсингу образуются три разные [[Изоформа белка|изоформы]] одного и того же белка]] |
|||
[[Файл:DNA alternative splicing.gif|thumb|400px|Альтернативный сплайсинг мРНК]] |
|||
'''Альтернати́вный спла́йсинг''' — процесс в [[Экспрессия генов|экспрессии генов]], приводящий к образованию нескольких [[Белок|белков]] с одного [[ген]]а. В ходе этого процесса отдельные [[экзон]]ы могут быть включены в зрелую [[мРНК]] или же отброшены<ref name=Black/>. В результате белки, считанные с мРНК одного гена, подвергшихся альтернативному [[сплайсинг]]у, будут иметь разные [[Аминокислоты|аминокислотные]] последовательности и нередко и разные функции. Благодаря альтернативному сплайсингу в человеческом [[геном]]е закодировано гораздо больше белков, чем можно было бы ожидать с его 20 тысяч белоккодирующих генов. |
|||
Альтернативный сплайсинг широко распространен у [[Эукариоты|эукариот]], значительно увеличивая разнообразие белков, закодированных в их геномах. У [[человек]]а около 95 % многоэкзонных генов подвергаются альтернативному сплайсингу<ref name=Pan2008/>. Существует много разновидностей альтернативного сплайсинга, из которых наиболее часто встречается пропуск экзонов. При пропуске экзонов отдельные экзоны в некоторых тканях или при определённых условиях могут быть включены в зрелую мРНК или пропущены<ref name=Black/>. |
|||
'''Альтернативный сплайсинг''' — процесс, позволяющий одному гену производить несколько мРНК и, соответственно, белков. Большинство генов в эукариотических геномах содержат [[экзон]]ы и [[интрон]]ы. После транскрипции в процессе [[сплайсинг]]а [[интрон]]ы удаляются из пре-мРНК. А вот [[экзон]] может включаться (или нет) в состав конечного транскрипта. Таким образом, с помощью альтернативного сплайсинга можно получить множество транскриптов, а, следовательно, и белков. Объединение различных сайтов [[сплайсинг]]а позволяет индивидуальным генам экспрессировать множество [[мРНК]], которые кодируют белки, порой, с антагонистическими функциями. [[Экзон]] одного варианта сплайсинга может оказаться [[интрон]]ом в альтернативном пути. Разные варианты сплайсинга могут приводить к образованию разных изоформ одного и того же белка. Например, ген [[тропонин]]а состоит из 18 [[экзон]]ов и кодирует многочисленные изоформы этого мышечного белка. Разные изоформы [[тропонин]]а образуются в разных тканях и на определенных стадиях их развития. |
|||
Образование альтернативно сплайсированных мРНК находится под контролем системы [[Трансрегуляторные элементы|''транс'']]-действующих белков, которые связываются с [[Цис-регуляторные элементы|''цис''-сайтами]] самого первичного транскрипта. К этим белкам относятся активаторы сплайсинга, которые способствуют использованию отдельных сайтов сплайсинга, а также репрессоры сплайсинга, которые выключают использование некоторых сайтов сплайсинга. Механизмы альтернативного сплайсинга очень разнообразны, и постоянно открываются новые примеры, в частности, с помощью высокопроизводительных методов. Учёные работают над тем, чтобы полностью изучить все механизмы сплайсинга, чтобы с помощью «кода сплайсинга» можно было предсказывать продукты альтернативного сплайсинга для данного гена в определённых условиях<ref name=Matlin/><ref name=David/>. |
|||
Предположено, что у эукариот альтернативный [[сплайсинг]] может быть важным эволюционным достижением: повысилась эффективность хранения информации.<ref name=ReferenceC>[http://www.bioscience-explained.org/ENvol4_1/pdf/spliceeng.pdf Alternative splicing]</ref> Недавно было показано, что у примерно 95 % мультиэкзонных генов человека наблюдается альтернативный сплайсинг.<ref name=ReferenceB>[http://www.nature.com/ng/journal/v40/n12/full/ng.259.html Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing]</ref> Геном круглого червя ''[[Caenorhabditis elegans]]'' по количеству генов практически не отличается от генома человека, однако альтернативному сплайсингу подвергаются пре-мРНК только 15 % генов. Таким образом, альтернативный сплайсинг позволяет увеличить разнообразие белковых продуктов [[ген]]ов, сохраняя при этом относительно небольшое количество различных [[ген]]ов в [[геном]]е и не создавая избыточных копий генов. |
|||
Ненормальные случаи альтернативного сплайсинга нередко приводят к болезням; значительное количество [[Генетические заболевания|генетических заболеваний]] человека возникает из-за альтернативного сплайсинга<ref name=Matlin/>. Нарушенный сплайсинг может приводить к развитию [[Рак (заболевание)|рака]], и в различных видах рака гены факторов сплайсинга нередко [[Мутация|мутируют]]<ref name="Skotheim and Nees 2007">{{Cite doi|10.1016/j.biocel.2007.02.016}}</ref><ref name=He2009/><ref name=Fackenthal/><ref name = "Sveen 2015">{{cite pmid|26300000}}</ref><ref name="Sveen 2015"/>. |
|||
Разные варианты альтернативного сплайсинга одной пре-мРНК могут осуществляться в разные периоды развития организма и/или в разных тканях, а также у разных особей одного вида.<ref>[http://www.nature.com/nature/journal/v456/n7221/full/nature07509.html Alternative isoform regulation in human tissue transcriptomes : Article : Nature]</ref> |
|||
== История изучения == |
|||
Один из наиболее впечатляющих примеров альтернативного сплайсинга — ген ''dscam'' в ''Drosophila melanogaster'', содержащий 116 экзонов, из которых 17 всегда попадают в конечную мРНК. Теоретически, данная система может продуцировать 38016 различных белков. В действительности, обнаружено более 18000 в ''Drosophila melanogaster''. Белки Dscam отвечают за правильное местонахождение нейронов и также, вероятно, участвуют в распознавании и фагоцитозе чужеродных бактерий.<ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1461822/pdf/11606537.pdf Alternative splicing of the Drosophila Dscam pre-mRNA is both temporally and spatially regulated]</ref> |
|||
Впервые альтернативный сплайсинг был описан в 1977 году<ref>{{cite pmid|902310}}</ref><ref>{{cite pmid|269380}}</ref>. [[Аденовирус]] образует пять различных [[Транскрипция (биология)|транскриптов]] в ранней стадии [[Инфекция|инфекционного]] цикла, до [[Репликация ДНК|репликации]] [[вирус]]ной [[ДНК]], и ещё один после начала репликации ДНК. После начала репликации ДНК продолжают образовываться ранние первичные транскрипты. Дополнительный одиночный транскрипт, образуемый на поздних стадиях инфекционного цикла, очень крупный и считывается с 5/6 аденовирусного генома размером 32 килобазы. Он гораздо крупнее каждого из вирусных транскриптов, образованных в заражённой клетке на ранних стадиях инфекционного цикла. Учёные показали, что первичный транскрипт, образуемый аденовирусом типа 2 на поздних стадиях инфекции, подвергается сплайсингу разными способами, в результате чего образуются различные мРНК, кодирующие разные вирусные белки. Кроме того, первичный транскрипт содержит множество сайтов [[Полиаденилирование|полиаденилирования]], в результате чего у разных мРНК могут получаться разные 3'-концы<ref name=Leff86>{{cite pmid|3017190}}</ref><ref name="pmid719751">{{cite pmid|719751}}</ref><ref name="pmid729004">{{cite pmid|729004}}</ref>. |
|||
В 1981 году альтернативный сплайсинг был описан у обычного [[Клетка (биология)|клеточного]] гена. Было показано, что в клетках [[Млекопитающие|млекопитающих]] [[гормон]] [[кальцитонин]] образуется в результате альтернативного сплайсинга. Первичный транскрипт гена кальцитонина содержит 6 экзонов; в зрелую мРНК, кодирующую кальцитонин, входят экзоны 1—4, и сигнал полиаденилирования находится в экзоне 4. У другой мРНК, образуемой из того же первичного транскрипта, при сплайсинге экзон 4 пропускается, и зрелая мРНК содержит экзоны 1—3, 5 и 6. Она кодирует белок, известный как {{нп5|CGRP||en|Calcitonin gene-related peptide}} ({{lang-en|calcitonin gene related peptide}})<ref name="pmid7207587">{{cite pmid|7207587}}</ref><ref name="pmid6952224">{{cite pmid|6952224}}</ref>. В начале 1980-х также был открыт альтернативный сплайсинг в генах [[иммуноглобулин]]ов млекопитающих<ref name=Leff86/><ref name="pmid6786756">{{cite pmid|6786756}}</ref>. |
|||
== Классификация == |
|||
[[Файл:Alt splicing bestiary.jpg|right|thumb|Alt splicing bestiary]] |
|||
С тех пор было показано, что альтернативный сплайсинг распространён среди всех эукариот<ref name=Black/>. Рекорд по количеству белков, образуемых с одного и того же гена, принадлежит гену плодовой мушки ''[[Drosophila melanogaster]]'', известному как {{нп5|Dscam||en|DSCAM}}. Он имеет 38016 разных вариантов сплайсинга<ref name=Schmucker>{{cite pmid|10892653}}</ref>. |
|||
Существует несколько механизмов альтернативного сплайсинга: |
|||
# '''Пропуск экзона или экзонной кассеты''': в этом случае экзон может вырезаться из первичного транскрипта или сохраняться в нём. Это наиболее часто используемый механизм у млекопитающих. |
|||
# '''Взаимоисключающие экзоны''': из двух экзонов в конечном транскрипте сохраняется только один. |
|||
# '''Использование альтернативного донорного сайта''': есть несколько альтернативных 5'-участков [[сплайсинг]]а (донорных сайтов), что изменяет 3'-границу вышележащего (upstream) экзона. |
|||
# '''Использование альтернативного акцепторного сайта''': используются разные 3'-участки [[сплайсинг]]а (акцепторные сайты), что меняет 5'-границы нижележащего (downstream) экзона. |
|||
# '''Удержание интрона''': интрон сохраняется в последовательности транскрипта. Если интрон находится в кодирующей последовательности, то он может кодировать стоп-кодон или же сдвигать рамку считывания. А это может привести к потере функциональности белка, поэтому данный механизм альтернативного сплайсинга используется крайне редко у млекопитающих. |
|||
== Модели == |
|||
Помимо описанных основных механизмов альтернативного сплайсинга существует ещё 2 других механизма, благодаря которым разные мРНК могут получаться из одного гена: '''множественные промоторы''' и '''множественные сайты полиаденилирования'''. Транскрипция может начинаться с разных точек. В результате получаются транскрипты с разными экзонами на 5’-конце (множественные промоторы). Множественные сайты полиаденилирования обеспечивают разные 3’-концы для транскрипта. Оба механизма найдены в комбинации с альтернативным сплайсингом и добавляют разнообразия в типы мРНК, полученных от одного гена.<ref name=ReferenceB /><ref>[http://www.nature.com/nrg/journal/v11/n5/full/nrg2776.html Alternative splicing and evolution: diversification, exon definition and function]</ref> |
|||
[[Файл:Alt splicing bestiary2.jpg|left|thumb|Традиционная классификация базовых типов альтернативного сплайсинга]] |
|||
[[Файл:Splice types frequency.jpg|lright|thumb|Относительная частота моделей альтернативного сплайсинга различается у человека и плодовой мушки<ref name=Sammeth/>]] |
|||
Существует пять моделей протекания альтернативного сплайсинга<ref name=Black/><ref name=Pan2008>{{cite pmid|18978789}}</ref><ref name=Matlin>{{cite pmid|15956978}}</ref><ref name=Sammeth>{{cite pmid|18688268}}</ref>: |
|||
* ''Пропуск экзона'' или ''кассетный экзон''. В этом случае экзон может быть включен в состав зрелой мРНК или вырезан. Это наиболее распространённая модель протекания альтернативного сплайсинга у пре-мРНК млекопитающих<ref name=Sammeth/>. |
|||
* ''Взаимоисключающие экзоны''. Из двух экзонов в зрелой мРНК находится только один, но не оба. |
|||
* ''Альтернативный донорный сайт''. Используется альтернативный 5'-конец [[интрон]]а (донорный сайт), так что меняется 3'-конец вышестоящего экзона. |
|||
* ''Альтернативный акцепторный сайт''. Используется альтернативный 3'-конец интрона (акцепторный сайт), так что меняется 5'-конец нижестоящего экзона. |
|||
* ''Сохранение интрона''. Последовательность может быть вырезана как интрон или оставлена в зрелой мРНК. Этот способ отличается от пропуска экзона, поскольку сохраняемая последовательность не фланкирована интронами. Если оставленный интрон попадает в {{нп5|Кодирующая область|кодирующую область|en|Coding region}}, то он должен кодировать аминокислотную последовательность с такой же [[Открытая рамка считывания|рамкой считывания]], как и соседние экзоны. Если он будет содержать [[стоп-кодон]] или будет иметь другую рамку считывания, нежели окружающие экзоны, то произойдёт сдвиг рамки считывания и белок будет нефункциональным. Это самый редкий путь альтернативного сплайсинга у млекопитающих<ref name=Sammeth/>. |
|||
Кроме этих пяти основных моделей протекания альтернативного сплайсинга, существуют два способа получения нескольких белков с одного гена: использования множественных [[промотор]]ов и множественных сайтов полиаденилирования. Использование множественных промоторов относится скорее к регуляции транскрипции, чем к альтернативному сплайсингу. Начиная транскрипцию с разных точек, можно получить транскрипты с различающимися 5'-концевыми экзонами. С другой стороны, использование множественных сайтов полиаденилирования приведёт к образованию разных 3'-концов у созревающих транскриптов. Оба этих механизма в сочетании с пятью моделями сплайсинга обеспечивают разнообразие мРНК, считываемых с одного и того же гена<ref name=Black/><ref name=Matlin/>. |
|||
[[Файл:Hyaluronidase3.gif|right|thumb|Схематическое представление мРНК гена мышиной гуалуронидазы. Экзоны и интроны изображены не в масштабе]] |
|||
Эти модели хорошо описывают базовые механизмы сплайсинга, однако они могут не подходить для сложных случаев. Например, на рисунке справа представлены три формы [[Мыши|мышиного]] гена [[Гиалуронидаза|гиалуронидазы]] 3, полученные в ходе аналогичного сплайсинга. Сравнение экзонов первой формы (зелёная) и второй (жёлтая) указывает на то, что имело место сохранение интрона, а сравнение второй формы с третьей (синяя) показывает, что произошёл пропуск экзона<ref name=Sammeth/>. |
|||
== Механизм == |
== Механизм == |
||
=== Общая схема сплайсинга === |
|||
{{main|Сплайсинг}} |
|||
[[Сплайсинг]] осуществляется сплайсосомой — массивной структурой, в состав которой входит 5 малых ядерных нуклеопротеидных частиц (snRNP) — U1, U2, U4, U5 и U6 (U3 не участвует в [[сплайсинг]]е) — и большого количества вспомогательных белков. Вместе они способны точно распознать сайты [[сплайсинг]]а и катализировать 2 шага реакции сплайсинга. |
|||
[[Файл:A complex.jpg|thumb|Сплайсосомный комплекс А определяет 5'- и 3'-концы интронов<ref name=Matlin/>]] |
|||
Когда пре-мРНК транскрибируется с ДНК, она содержит несколько интронов и экзонов. Так, у нематод обычно имеется 4—5 интронов и экзонов, а у дрозофилы может быть более 100 интронов и экзонов в одной пре-мРНК. В ходе сплайсинга экзоны должны быть оставлены в транскрипте, а интроны удалены. Регуляция и выбор сайтов сплайсинга осуществляется ''транс''-действующими белками-активаторами и репрессорами сплайсинга, а также ''цис''-действующими элементами в самой пре-мРНК, такими как энхансеры и сайленсеры сплайсинга. Типичные эукариотические интроны имеют {{нп5|Консенсусная последовательность|консенсусные последовательности|en|Consensus sequence}} в определённых местах; так, на 5'-конце каждого интрона располагается ди[[нуклеотид]] GU. Рядом с 3'-концом находится точка ветвления, и в точке ветвления всегда находится нуклеотид [[Аденозин|А]], а вокруг него последовательности несколько варьируют. У человека консенсусная последовательность вокруг точки ветвления выглядит как yUnAy<ref name=Gao>{{cite pmid|18285363}}</ref>. После точки ветвления располагается ряд [[Пиримидины|пиримидинов]] ({{нп5|полипиримидиновый тракт||en|Polypyrimidine tract}}), 3'-конец которого выглядит как AG<ref name=Matlin/>. |
|||
Сплайсинг пре-мРНК осуществляет [[РНК]]-белковый комплекс, известный как [[сплайсосома]]. В состав сплайсосомы входят {{нп5|малые ядерные рибонуклеопротеиды||en|snRNP}} (snRNP), обозначаемые {{нп5|U1 (РНК)|U1|en|U1 spliceosomal RNA}}, {{нп5|U2 (РНК)|U2|en|U2 spliceosomal RNA}}, {{нп5|U4 (РНК)|U4|en|U4 spliceosomal RNA}}, {{нп5|U5 (РНК)|U5|en|U5 spliceosomal RNA}} и {{нп5|U6 (РНК)|U6|en|U6 spliceosomal RNA}} (U3 не участвует в сплайсинге мРНК)<ref name=Clark>{{cite book |author=Clark, David |title=Molecular biology |publisher=Elsevier Academic Press |location=Amsterdam |year=2005 |isbn=0-12-175551-7}}</ref>. U1 связывается с 5'-концевым динуклеотидом GU, а U2, при участии белковых факторов U2AF, связывается с точкой ветвления. На этой стадии комплекс известен как сплайсосомный А-комплекс. Во время формирования А-комплекса определяются 5'- и 3'-границы удаляемого интрона, а также концы экзонов, которые должны быть оставлены<ref name=Matlin/> . |
|||
Далее с А-комплексом связывается комплекс U4, U5, U6. После этого U6 замещает U1, а U1 и U4 покидает комплекс. Оставшийся комплекс осуществляет две реакции [[Переэтерификация|пересэтерификации]]. В ходе первой реакции 5'-конец интрона отрезается от вышележащего экзона и присоединяется к нуклеотиду А в точке ветвления с помощью 2',5'-[[Фосфодиэфирная связь|фосфодиэфирной связи]]. В ходе второй реакции отрезается 3'-конец интрона, и два экзона соединяются фосфодиэфирной связью. Интрон, принимающий форму лассо, высвобождается и далее деградирует<ref name=Black>{{cite pmid|12626338}}</ref>. |
|||
=== Регуляторные элементы и белки === |
|||
[[Файл:Splicing repression.jpg|thumb|left|Репрессия сплайсинга]] |
|||
Сплайсинг регулируется ''транс''-действующими белками (активаторами и репрессорами) и соответствующими ''цис''-регуляторными элементами (сайленсерами и энхансерами) на пре-мРНК. Впрочем, имеются данные, что во многих случаях действие фактора сплайсинга зависит от положения. Когда фактор сплайсинга связан с интронным энхансерным элементом, он действует как активатор сплайсинга, а когда связывается с регуляторным сайтом в экзоне, действует как репрессор, и наоборот<ref name="Lim">{{cite pmid|21685335}}</ref>. В регуляции сплайсинга также принимает участие [[вторичная структура]] пре-мРНК, сближающая друг с другом два регуляторных элемента или маскируя последовательности, которые могли бы служить местами связывания факторов сплайсинга<ref>{{cite pmid|19959365}}</ref><ref name="ncbi.nlm.nih.gov">{{cite pmid|198614269}}</ref>. Вместе все эти элементы образуют «код сплайсинга», который определяет, как будет проходить сплайсинг в данных клеточных условиях<ref name=Wang>{{Cite pmid|18369186}}</ref><ref name=Barash>{{Cite pmid|20445623}}</ref>. |
|||
Имеется два типа ''цис''-активирующих элементов в пре-мРНК, и им соответствуют ''транс''-активирующие РНК-связывающие белки. Сайленсеры сплайсинга — это элементы, с которыми связываются белки-репрессоры сайленсинга, при этом снижая вероятность того, что по соседству будет находиться сайт сплайсинга. Сайленсеры сплайсинга могут находиться в интронах (интронные сайленсеры сплайсинга, ISS) или в экзонах (экзонные сайленсеры сплайсинга, ESS). Их последовательности, как и связывающиеся белки, сильно разнятся. Большинство репрессоров сплайсинга являются {{нп5|Гетерогенные ядерные рибонуклеопротеиды|гетерогенными ядерными рибонуклеопротеидами|en|Heterogeneous ribonucleoprotein particle}} (hnRNP), такие как {{нп5|hnRNPA1||en|Heterogeneous nuclear ribonucleoprotein A1}} и белок, связывающий полипиримидиновый тракт (PTB). С энхансерами сплайсинга связываются белки-активаторы сплайсинга, увеличивая вероятность того, что рядом будет находиться сайт сплайсинга. Они тоже могут находиться в интронах (интронные энхансеры сплайсинга, ISE) или в экзонах (экзонные энхансеры сплайсинга, ESE). Большая часть белков, связывающихся с ISE и ESE, относится к [[Белковое семейство|семейству белков]] SR. Эти белки содержат [[Мотив (молекулярная биология)|мотивы]], распознающие РНК, а также [[Домен белка|домены]], обогащённые [[аргинин]]ом и [[серин]]ом<ref name=Matlin/><ref name=Wang/. |
|||
[[Файл:Splicing activation.jpg|thumb|left|Активация сплайсинга]] |
|||
Таким образом, факторы сплайсинга работают взаимозависимо, а также зависят от окружающих условий<ref name=Barash/>. Наличие определённых ''цис''-регуляторных последовательностей РНК увеличивают вероятность того, что рядом будет находиться сайт сплайсинга, а в других уменьшают эту вероятность в зависимости от контекста. Например, некоторые такие элементы влияют на сплайсинг только тогда, когда рядом располагается ряд определённых элементов. Кроме того, ''цис''-регуляторные элементы могут давать разный эффект при экспрессии в клетке определённых белков. Адаптивное значение энхансеров и сайленсеров сплайсинга подтверждается работами, показывающими, что в человеческих генах на мутации, приводящие к образованию новых сайленсеров или разрушению старых энхансеров, действует строгий [[Естественный отбор|отбор]]<ref name="Ke">{{cite pmid|18204002}}</ref><ref>{{cite pmid|15340491}}</ref>. |
|||
=== Примеры === |
|||
==== Пропуск экзона: ген ''dsx'' дрозофилы ==== |
|||
[[Файл:Dsx splicing.jpg|thumb|Альтернативный сплайсинг пре-мРНК ''dsx'']] |
|||
Пре-мРНК гена ''{{нп5|dsx||en|Doublesex}}'' ''D. melanogaster'' содержит 6 экзонов. У самцов в зрелую мРНК входят экзоны 1, 2, 3, 5, 6, и они кодируют белок, который функционирует как [[Транскрипционный фактор|регулятор транскрипции]] в развитии по мужскому типу. У самок в зрелую мРНК входят экзоны 1, 2, 3 и 4, и экзон 4 содержит сигнал полиаденилирования, по которому мРНК разрезается. Получившийся белок функционирует как регулятор транскрипции в развитии по женскому типу<ref name=Lynch>{{cite pmid|8769651}}</ref>. |
|||
В описанном примере имеет место альтернативный сплайсинг по типу пропуска экзона. Интрон, лежащий выше экзона 4, содержит полипиримидиновый тракт, который не вполне удовлетворяет консенсусной последовательности сплайсинга, поэтому белки U2AF связываются с ним плохо в отсутствие активаторов сплайсинга. По этой причине у самцов этот 3'-акцепторный сайт сплайсинга не используется. У самок, однако, имеется активатор сплайсинга Transformer (Tra). SR-белок Tra2 образуется у обоих полов и связывается с ESE в экзоне 4; если имеется Tra, то он связывается с Tra2 и вместе с ещё одним SR-белком формирует комплекс, который содействует связыванию белков U2AF со слабым пиримидиновым трактом. U2 рекрутируется к соответствующей точке ветвления, и в результате экзон 4 включается в состав зрелой мРНК<ref name=Lynch/><ref name=Graveley>{{cite pmid|11421359}}</ref>. |
|||
==== Альтернативные акцепторные сайты: ''Transformer'' дрозофилы ==== |
|||
[[Файл:transformer splicing.gif|left|thumb|Альтернативный сплайсинг пре-мРНК гена ''Transformer'' дрозофилы]] |
|||
Пре-мРНК гена ''Transformer'' (Tra) ''D. melanogaster'' подвергаются альтернативному сплайсингу по модели альтенативных акцепторных сайтов. Ген Tra кодирует белок, экспрессирующийся только у самок. Первичный транскрипт этого гена содержит интрон с двумя возможными акцепторными сайтами. У самцов используется вышележащий акцепторный сайт. В итоге в мРНК включается удлинённый вариант экзона 2, содержащий преждевременный стоп-кодон. Поэтому у самцов образуется укороченный неактивный белок. У самок образуется белок, играющий ключевую роль в определении пола и известный как Sex lethal (Sxl). Белок Sxl является репрессором сплайсинга, который связывается с ISS в РНК-транскрипте Tra рядом с вышележащим акцепторным сайтом, предотвращая связывание белка U2AF с полипиримидиновым трактом. В результате сплайсосома связывается с нижележащим акцепторным сайтом. При использовании этого сайта преждевременный стоп-кодон удаляется в составе интрона. Получившаяся мРНК кодирует белок Tra, который сам выступает регулятором альтернативного сплайсинга других связанных с полом генов (см. выше пример гена ''dsx'')<ref name=Black/>. |
|||
==== Альтернативный сплайсинг рецептора Fas ==== |
|||
Сборка сплайсосомы начинается с распознавания U1 5’-сайта [[сплайсинг]]а. После этого фактор [[сплайсинг]]а 1 (SF1) связывается в точке разветвления (branch point, обычно это аденозин). Такой комплекс носит название E’. Затем большая субъединица (65 kDa) гетеродимерного вспомогательного фактора U2 (U2AF) образует комплекс с полипиримидиновым участком (polypyrimidine tract), а малая субъединица (35 kDa) U2AF связывается с 3’-терминальным AG на стыке интрона и экзона. Весь этот образовавшийся комплекс является АТФ-независимым и уже называется комплексом E. Позже, после замены SF1 на U2 в точке разветвления, комплекс Е становится АТФ-зависимым комплексом А. |
|||
[[Файл:Fas alternative splicing.jpg|thumb|right|Альтернативный сплайсинг пре-мРНК рецептора Fas]] |
|||
Множество [[Изоформа белка|изоформ]] [[Клеточный рецептор|рецептора]] {{нп5|Fas||en|Fas receptor}} образуются в результате альтернативного сплайсинга. Две нормальные изоформы у человека образуются по механизму пропуска экзона. мРНК, содержащая 6 экзонов, кодирует [[Клеточная мембрана|мембраносвязанную]] форму рецептора Fas, которая стимулирует [[апоптоз]]. Повышенное образование рецептора Fas в клетках, постоянно подвергающихся воздействию солнечного света, и отсутствие этого рецептора в клетках [[Рак кожи|рака кожи]] свидетельствует о том, что этот механизм играет важную роль в элиминации клеток, которые встали на путь превращения в раковые<ref name=Filipowicz>{{cite pmid|11857317}}</ref>. Если экзон 6 пропускается, то образуется водорастворимая изоформа белка Fas, которая неспособна стимулировать апоптоз. Вставка или пропуск экзона зависят от действия двух белков-антагонистов {{нп5|TIA-1||en|TIA1}} и PTB. |
|||
Донорный сайт, расположенный на 5'-конце интрона, следующим после экзона 6 в пре-мРНК, плохо согласуется с консенсусной последовательностью сплайсинга, и не всегда связывается с snRNP U1<ref name=Matlin/>. Если U1 не связывается, экзон пропускается (картинка а на рисунке справа). Связывание белка TIA-1 с интронным энхансером сплайсинга стабилизирует связывание U1. Образующийся на 5'-конце интрона донорный сайт помогает связываться фактору сплайсинга U2AF с 3'-сайтом сплайсинга, расположенным выше экзона, хотя механизм этого ещё не понятен (картинка b на рисунке справа)<ref name=Izquierdo2005>{{cite pmid|16109372}} |
|||
Дальнейшее образование тройного U4/U6-U5 комплекса приводит к формированию комплекса В, который после экстенсивных конформационных изменений и перестроек превращается в каталитически активный комплекс С.<ref name=ReferenceA>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2958924/ Mechanisms of alternative splicing regulation: insights from molecular and genomics approaches]</ref> |
|||
</ref>. Экзон 6 содержит обогащённый пиримидинами сайленсер сплайсинга (''ure6''), с которым может связываться PTB. Если связывание PTB происходит, то донорный сайт на 5'-конце интрона не способствует связыванию фактора U2AF, и экзон пропускается (картинка с на рисунке справа). |
|||
U6 занимает позицию U1, а U1 и U4 уходят. Оставшийся комплекс претерпевает 2 реакции переэтерификации. В ходе первой реакции, 5’-конец интрона отщепляется от экзона, что перед ним, и присоединяется к сайту разветвления А через 2’,5’-фосфодиэфирную связь. Во второй реакции переэтерификации, 3’-конец интрона отщепляется от экзона, что расположен далее по последовательности, и два экзона объединяются. Образовавшийся интрон высвобождается в форме лассо и позднее деградирует.<ref>[http://www.annualreviews.org/doi/abs/10.1146/annurev.biochem.72.121801.161720 Mechanisms of alternative pre-messenger RNA splicing]</ref> |
|||
Описанный выше механизм является примером определения экзона при сплайсинге. Сплайсосома собирается в области интрона, и snRNP так укладывают РНК, что 5'- и 3'-концы интрона соединяются. Однако в описанном выше случае происходит также взаимодействие концов экзона. В этом случае взаимодействия, определяющие границы экзона, необходимы для связывания коровых факторов сплайсинга до сборки сплайсосомы на границах фланкирующих интронов<ref name=Izquierdo2005/>. |
|||
== Регуляция == |
|||
Существуют различные способы регуляции альтернативного сплайсинга: РНК-белковые взаимодействия, РНК-РНК взаимодействия (при взаимоисключающих экзонах), взаимодействие пре-мРНК с некодирующими РНК, в том числе малыми ядрышковыми РНК и др. Несмотря на столь потенциальное разнообразие регуляторных механизмов, РНК-белковые взаимодействия считаются основными способами регуляции [[сплайсинг]]а.<ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3165201/ Understanding splicing regulation through RNA splicing maps]</ref> |
|||
==== Конкуренция репрессора и активатора: экзон 2 гена ''tat'' ВИЧ-1 ==== |
|||
=== Регуляторные элементы === |
|||
[[Файл:Tat exon2 splicing.jpg|thumb|left|Альтернативный сплайсинг экзона 2 ''tat'' ВИЧ-1]] |
|||
Участки последовательности РНК и регуляторы белков определяют, какой экзон удалить, а какой оставить. Существует 4 категории регуляторов: |
|||
[[ВИЧ]] — [[Ретровирусы|ретровирус]], являющийся причиной развития [[СПИД]]а — образует единственную пре-мРНК, из которой далее посредством альтернативного сплайсинга образуется более 40 различных мРНК<ref name=Zahler2004>{{cite pmid|14703516}}</ref>. Равновесие между по-разному сплайсированными транскриптами обеспечивает образование мРНК, кодирующих все белки, необходимые для репликации вируса<ref name=Jaquenet2001>{{cite pmid|11526107}}</ref>. Один из по-разному сплайсированных транскриптов содержит транскрипт гена ''{{нп5|tat (ВИЧ)|tat|en|tat (HIV)}}'', у которого экзон 2 является кассетным, то есть может быть включён в итоговый транскрипт или не включён. Включение этого экзона регулируется репрессором сплайсинга hnRNP A1 и {{нп5|SR-белки|SR-белком|en|SR protein}} SC35. В экзоне 2 сайленсерная последовательность (ESS) и энхансерная последовательность (ESE) перекрываются. Если с ESS связывается репрессор А1, то он запускает [[Кооперативность|кооперативное]] связывание [[Молекула|молекул]] А1, закрывая 5'-концевой донорный сайт выше экзона 2 и препятствуя связыванию U2AF35 с полипиримидиновым трактом. Если SC35 связывается с ESE, то он препятствует связыванию А1, и 5'-донорный сайт остаётся доступным для сплайсосомы. Конкуренция между репрессором и активатором приводит к образованию РНК, содержащей или не содержащей экзон 2<ref name=Zahler2004/>. |
|||
* энхансеры сплайсинга экзонов (exonic splicing enhancers, ESEs) |
|||
* сайленсеры сплайсинга экзонов (exonic splicing silencers, ESSs) |
|||
* энхансеры сплайсинга интронов (intronic splicing enhancers, ISEs) |
|||
* сайленсеры сплайсинга интронов (intronic splicing silencers, ISSs) |
|||
== Адаптивное значение == |
|||
=== Регуляторы, помогающие распознать сайт сплайсинга === |
|||
Альтернативный сплайсинг — это одно из исключений из правила, что одному гену соответствует один белок (гипотеза «один ген — один [[фермент]]»)<ref name=HHMI> |
|||
Белки SR (серин(S)/аргинин(R)-богатые белки) играют важную роль в распознавании сайта [[сплайсинг]]а. Например, когда они взаимодействуют с энхансерами экзонов (ESE), они помогают связываться U1 с 5’-сайтом [[сплайсинг]]а, а также U2AF и U2 с 3’-сайтом [[сплайсинг]]а. Эти взаимодействия (SR c энхансерами) происходят благодаря их RS доменам (содержат Arg-Ser повторы). Белки SR могут работать вместе с другими положительными регуляторами.<ref name=ReferenceA /> |
|||
{{cite web |
|||
|url=http://www.hhmi.org/bulletin/sept2005/features/splicing.html |
|||
|title=HHMI Bulletin September 2005: Alternative Splicing |
|||
|publisher=www.hhmi.org |
|||
|accessdate=2009-05-26| archiveurl= https://web.archive.org/web/20090622085747/http://www.hhmi.org/bulletin/sept2005/features/splicing.html| archivedate= 22 June 2009 <!--DASHBot-->| deadurl= no}} |
|||
</ref>. Корректнее было бы сказать, «один ген — много [[полипептид]]ов». Внешняя информация нужна для того, чтобы решить, какой именно полипептид образовать с данной мРНК. Поскольку способы регуляции наследуются, то это даёт мутациям новый путь к изменению экспрессии генов<ref name=Fackenthal/>. |
|||
Предполагается, что для эукариот альтернативный сплайсинг — очень важный шаг на пути к повышению эффективности, поскольку он даёт возможность хранить информацию более экономно. Один ген может давать начало нескольким белкам, а не один ген даёт начало только одному белку, поэтому одно и то же разнообразие [[протеом]]а можно получить с генома существенно меньшего размера<ref name=Black/>. Это также обеспечивает [[Эволюция (биологическая)|эволюционную]] гибкость. Из-за единственной точечной мутации может привести к включению или исключению экзона из транскрипта, благодаря чему может быть получена новая изоформа белка без потери его основной формы<ref name=Black/>. Обнаружены действительно неупорядоченные регионы, которые содержат много неконститутивных экзонов, поэтому изоформы белка может выполнять новые функции, изменяя функциональные модули в этих местах<ref name=Romero>{{cite pmid|16717195}}</ref><ref>{{cite pmid|24951248}}</ref><ref name=Eksi>{{cite pmid|24244129}}</ref>. Сравнительные оценки показывают, что возникновение альтернативного сплайсинга в ходе эволюции предшествовало появлению многоклеточности, и предполагается, что альтернативный сплайсинг был одним из средств, обеспечивающих возникновение [[Многоклеточные организмы|многоклеточных организмов]]<ref name=Irimia>{{cite pmid|17916237}}</ref>. |
|||
SR белок SRp38 (также известный как TASR, NSSR, FUSIP1 и SRrp40) в дефосфорилированном действует как репрессор [[сплайсинг]]а.<ref>[http://www.nature.com/nature/journal/v427/n6974/full/nature02288.html Dephosphorylated SRp38 acts as a splicing repressor in response to heat shock]</ref> Однако, недавние исследования показали, что в фосфорилированном состоянии он также функционирует в качестве активатора, зависящего от сплайсируемой последовательности.<ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2668916/ Phosphorylation switches the general splicing repressor SRp38 to a sequence-specific activator]</ref> |
|||
Исследования в рамках [[Проект «Геном человека»|проекта «Геном человека»]], а также других проектов по [[Секвенирование|секвенированию]] геномов показали, что геном человека всего лишь на 30% больше генома [[Круглые черви|круглого червя]] ''[[Caenorhabditis elegans]]'', и всего лишь в два раза больше, чем у плодовой мушки ''Drosophila melanogaster''. Эти данные наводят на мысль, что сложность человека и [[позвоночные|позвоночных]] [[Животные|животных]] вообще может быть связана с более активным, по сравнению с [[Беспозвоночные|беспозвоночными]], использованию альтернативного сплайсинга<ref name=ewing>{{cite pmid|10835644}}</ref><ref name=Crollius>{{cite pmid|10835645}}</ref>. Однако дальнейшее изучение последовательностей человека, мыши, [[Крыса|крысы]], [[Корова|коровы]], ''D. melanogaster'', ''C. elegans'' и [[растения]] ''[[Arabidopsis thaliana]]'' показало, что между человеком и другими животными не наблюдается значительной разнице в использовании альтернативного сплайсинга<ref> |
|||
=== Ингибирование распознавания сайта сплайсинга === |
|||
{{cite pmid|11743582}}</ref>. Впрочем, имеются сведения, что полученные данные были артефактом. При сравнении частоты использования альтернативного сплайсинга случайного набора генов каждого организма оказалось, что позвоночные всё-таки прибегают к альтернативному сплайсингу чаще беспозвоночных<ref name=Kim>{{cite pmid|17158149}}</ref>. |
|||
Ингибировать распознавание сайта [[сплайсинг]]а можно разными способами. Первый, когда сайленсеры [[сплайсинг]]а располагаются близко к сайту [[сплайсинг]]а или к энхансерам [[сплайсинг]]а, и, соответственно, может происходить стерическое ингибирование, при котором блокируется доступ к snRNP или положительным регуляторным факторам. Например, полипиримидин-связывающий белок (PTB; также известный как PTB1 и hnRNP I), связывается с полипиримидиновым участком и, тем самым, блокирует связывание U2AF с экзонами. Тканеспецифичные факторы [[сплайсинг]]а FOX1 и FOX2, связываясь с интронной последовательностью, могут ингибировать формирование комплекса E', предотвращая связывание SF1 с точкой разветвления.<ref name=ReferenceA /> |
|||
== Клиническое значение == |
|||
Ингибиторы [[сплайсинг]]а могут стерически мешать связыванию активаторов с энхансерами. Семейство белков Hu/ELAV ингибирует связывание U1, конкурируя с белком TIA1, который взаимодействует с AU-богатой областью, расположенной далее по последовательности в 5’-сайте сплайсинга экзона 23а нейрофиброматозного типа 1.<ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2258745/ Regulation of Neuron-Specific Alternative Splicing of Neurofibromatosis Type 1 Pre-mRNA]</ref> FOX1 и FOX2 также ингибируют формирование комплекса Е, связываясь с экзонной последовательностью, близко расположенной от ESE, где связываются TRA2 и SRp55. Из-за этого U2AF не может закрепиться.<ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519742/ Repression of Prespliceosome Complex Formation at Two Distinct Steps by Fox-1/Fox-2 Proteins]</ref> |
|||
Изменения в машинерии процессинга РНК могут приводить к нарушениям сплайсинга многих транскриптов, а однонуклеотидные замены в сайтах сплайсинга или ''цис''-регуляторных сайтах сплайсинга приводят к различиям в сплайсинге одного и того же гена, как и при спайсинге транскрипта мутировавшего гена. В работе 2005 года было показано, что свыше 60 % мутаций, приводящих к развитию болезней, влияют не на саму кодирующую последовательность, а на сплайсинг<ref name="Lopez-Bigas">{{cite pmid|15792793}}</ref>. Показано также, что треть наследственных заболеваний связана со сплайсингом<ref name="Lim" />. |
|||
Ненормально сплайсированные клетки встречаются в заметной доле раковых клеток<ref name="Skotheim and Nees 2007" /><ref name="He2009">{{cite pmid|19266097}}</ref><ref name="Sveen 2015" />. Анализ [[Секвенирование РНК|RNA-Seq]] и протеомов показал поразительно дифференциальную экспрессию сплайсинговых изоформ белков, участвующих в [[Передача сигнала (биология)|сигнальных путях]], связанных с развитием рака<ref>{{cite pmid|24802673}}</ref>. Неизвестно, влияют ли нарушения сплайсинга на развития рака напрямую, или же они являются следствием поломки клеточных процессов в связи с переходом к раковому росту. Интересно, что при некоторых видах рака, такой как [[рак толстой кишки]] или [[рак простаты]], количество ошибок в сплайсинге значительно варьировало у разных пациентов; этот феномен назвали транскриптомной нестабильностью<ref name="Sveen 2011">{{cite pmid|25109687}}</ref><ref name="Sveen 2014">{{cite pmid|21619627}}</ref>. Далее было показано, что транскриптомная нестабильность связана с пониженной экспрессией генов факторов сплайсинга. Действительно, в общем случае в раковых клетках альтернативный сплайсинг используется меньше, чем у нормальных клеток, причём модели сплайсинга тоже различаются. Так, в раковых клетках сохранение интрона происходит чаще, чем в нормальных клетках, а пропуск экзона — реже. Особенности сплайсинга в раковых клетках могут быть связаны с высокой частотой соматических мутаций в генах факторов сплайсинга, а некоторые особенности могут быть обусловлены изменениями в [[Фосфорилирование|фосфорилировании]] ''транс''-регуляторных факторов сплайсинга<ref name="Kim2008">{{cite pmid|18054115}}</ref><ref name="Fackenthal">{{Cite pmid|19048051}}</ref>. Некоторые особенности могут быть связаны с изменениями относительного количества факторов сплайсинга; например, в клетках [[Рак груди|рака груди]] наблюдаются повышенные уровни фактора сплайсинга {{нп5|SF2/ASF||en|Serine/arginine-rich splicing factor 1}}<ref name="Ghigna2005">{{cite pmid|16364913}}</ref>. В одном исследовании было показано, что относительно небольшая доля (383 из 26000) вариантов альтернативного сплайсинга в раковых клетках встречалась сильно чаще, чем в нормальных, поэтому существует ограниченное количество генов, неправильный сплайсинг которых приводит к развитию опухоли<ref name="pmid15048092">{{cite pmid|15048092}}</ref>. Считается, однако, что губительное действие нарушенного сплайсинга сдерживается особым клеточным посттранскрипционным механизмом контроля — [[Нонсенс-опосредованный распад|нонсенс-опосредованным распадом]]<ref name="Danckwardt2002">{{cite pmid|11861299}}</ref>. |
|||
=== Регуляция сплайсинга в зависимости от позиции экзона === |
|||
Действие цис-регуляторных элементов иногда зависит от позиции регулируемого экзона. Несколько белков, например такие как NOVA1, NOVA2, FOX1, FOX2, hnRNP l, hnRNP l-подобный, hnRNP F и hnRNP H, могут действовать либо как репрессоры, либо как активаторы в зависимости от места сайта связывания.<ref>[http://www.nature.com/nature/journal/v444/n7119/full/nature05304.html An RNA map predicting Nova-dependent splicing regulation]</ref><ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2519734/ hnRNP H and hnRNP F Complex with Fox2 To Silence Fibroblast Growth Factor Receptor 2 Exon IIIc]</ref><ref>[http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1142566/ Nova autoregulation reveals dual functions in neuronal splicing]</ref> |
|||
Одним из человеческих генов, специфический вариант сплайсинга которого связан с развитием рака, является один из генов [[ДНК-метилтрансфераза|DNMT]]. Три гена DNMT кодируют ферменты, которые добавляют [[Метильная группа|метильные группы]] к ДНК, и эта модификация часто имеет регуляторные эффекты. Несколько неправильно сплайсированных мРНК гена {{нп5|DNMT3B||en|DNMT3B}} были найдены в опухолях и клетках раковых {{нп5|Линия клеток|линий|en|Immortalised cell line}}. Экспрессия двух из этих мРНК вызывала изменения в [[Метилирование|метилировании]] ДНК в этих клетках. Клетки с одной ненормальной мРНК росли вдвое быстрее, чем контрольные клетки, поэтому эти мРНК связаны с развитием рака<ref name="Fackenthal" />. |
|||
Позиция [[сплайсинг]]а может определять действие факторов [[сплайсинг]]а. Энхансеры могут располагаться таким образом, что, когда факторы [[сплайсинг]]а связываются с ними, другой экзон лучше расположен для действия сплайсосомы. Или наоборот, элементы сайленсинга соревнуются с компонентами сплайсосомы или как-то изменяют структуру мРНК, препятствуя узнаванию сайта [[сплайсинг]]а.<ref name=ReferenceA /> |
|||
Другим примером может служить [[протоонкоген]] Ron ({{нп5|MST1R||en|MST1R}}). Важным свойством раковых клеток является их способность переселяться в нормальные [[Ткань (биология)|ткани]] и нарушать их работу. Образование ненормально сплайсированной мРНК Ron было связано с повышенными уровнями SF2/ASF в клетках рака груди. Ненормальная изоформа Ron, образованная с этой мРНК, увеличивала подвижность клеток<ref name="Ghigna2005" />. |
|||
== Методы обнаружения альтернативного сплайсинга == |
|||
Основным методом определения мест альтернативного сплайсинга в данный момент является [[секвенирование]] библиотек [[кДНК]], полученных на матрице [[мРНК]]. Данный метод называется [[Секвенирование РНК|секвенированием РНК]]. Также возможно применение [[ДНК-микрочип]]ов. |
|||
Сверхэкспрессия укороченного варианта белка {{нп5|FOSB||en|FOSB}} — ΔFosB — в специфической популяции [[нейрон]]ов [[Прилежащее ядро|прилежащего ядра]] лежит в основе возникновения и поддержания привыкания к наркотикам и естественному вознаграждению ({{Lang-en|natural reward}})<ref name="Cellular basis">{{cite pmid|24459410}}</ref><ref name="What the ΔFosB?">{{cite pmid|25083822}}</ref><ref name="G9a reverses ΔFosB plasticity">{{cite pmid|23020045}}</ref><ref name="Natural and drug addictions">{{cite pmid|21459101}}</ref>. |
|||
== Альтернативный сплайсинг и заболевания == |
|||
Изменения в альтернативном сплайсинге могут вызывать разные заболевания: нейродегенеративные, сердечно-сосудистые, дыхательные заболевания, а также рак и [[болезнь Альцгеймера]]. В тканях, где происходит множество процессов (например головной мозг), требуется большое количество белков для выполнения различных функций. Любой дефект в альтернативном сплайсинге может резко изменить состав белков в клетке, вследствие чего и возникнут многие неврологические заболевания. Эти дефекты могут быть разделены на две основные группы: первичные и вторичные дефекты сплайсинга. |
|||
Недавние провокационные исследования показывают на роль структуры [[хроматин]]а и модификаций [[гистон]]ов в регуляции альтернативного сплайсинга. Поэтому [[Эпигенетика|эпигенетические]] факторы могут влиять не только на экспрессию генов, но и на их сплайсинг<ref name="Luco2011">{{cite pmid|21215366}}</ref>. |
|||
=== Первичные дефекты сплайсинга === |
|||
При первичных дефектах [[сплайсинг]]а происходит мутация в последовательности, которая важна для правильного прохождения альтернативного сплайсинга. Следовательно, сплайсинг не происходит должным образом. Многие мутации, приводящие к заболеваниям, вызваны нарушениями сплайсинга пре-мРНК. Примерами являются [[синдром Луи-Бара]] и [[нейрофиброматоз]]. Половина пациентов, страдающих этими заболеваниями, несет мутации, которые влияют на прохождение [[сплайсинг]]а пре-мРНК. Другой пример первичных нарушений сплайсинга был обнаружен у пациентов с лобно-височной деменцией (аутосомно-доминантное расстройство, связано с 17 хромосомой). Дефекты в сплайсинге приводят к изменению транскриптов тау-белков, ассоциированных с микротрубочками, - MAPT. Эти белки накапливаются в аксонах или растущих нейронах. Изменения в сплайсинге MAPT могут также привести к другим заболеваниям, таким как [[болезнь Альцгеймера]], мышечная дистрофия, дисфункция глии и дегенерация спинного мозга. Мышечная дистрофия Дюшенна (МДД) — это результат мутаций в гене [[дистрофин]]а. Мутации вызывают нарушения в [[сплайсинг]]е и продукцию неправильных мРНК. Разные мутации в пре-мРНК [[дистрофин]]а вызывают промежуточный вариант МДД, мышечную дистрофию Беккера (МДБ). |
|||
== Полногеномный анализ == |
|||
=== Вторичные дефекты сплайсинга === |
|||
Полногеномный анализ альтернативного сплайсинга — сложная задача. Обычно альтернативно сплайсированные транскрипты обнаруживают при сравнении {{нп5|EST (генетика)|экспрессируемых последовательностей-меток|en|Expressed sequence tag}} ({{lang-en|Expressed sequence tag, EST}}). Большинство {{нп5|Библиотека (биология)|библиотек EST|en|Library (biology)}} собраны из очень ограниченного числа тканей, поэтому тканеспецифичные транскрипты не были учтены. Однако появились высокопроизводительные методы для изучения сплайсинга, такие как [[ДНК-микрочип]]ы и {{нп5|Покрытие (генетика)|глубокое секвенирование|en|Coverage (genetics)}} ({{Lang-en|deep sequencing}}). Эти методы могут быть использованы для поиска полиморфизмов и мутаций, расположенных в или вокруг элементов сплайсинга, которые влияют на связывание белков. При сочетании с такими методами изучения сплайсинга, как ''[[in vitro]]'' анализ [[Репортёрный ген|репортерных генов]], могут быть исследованы функциональные эффекты полиморфизмов и мутаций на сплайсинг пре-мРНК<ref name=Lim/><ref name=Wang/><ref>{{cite pmid|12114529}}</ref>. |
|||
Под вторичными дефектами сплайсинга подразумеваются мутации в регуляторных факторах, которые очень важны для процесса сплайсинга. Действие различных активаторов и/или репрессоров может определять пути прохождения альтернативного сплайсинга. В зависимости от того, какой регулятор действует, пре-мРНК меняется. В некоторых случаях, это может привести к серьёзным нарушениям. Примером таких нарушений является [[Синдром Прадера-Вилли]], генетическая болезнь, характеризующаяся интеллектуальными и поведенческими отклонениями. |
|||
При анализе с помощью микрочипов были использованы фрагменты ДНК, являющиеся отдельными экзонами (такие как микрочип {{нп5|Affymetrix||en|Affymetrix}}) или границами между экзонами. Затем в микрочип добавляется меченая кДНК из интересующей ткани. Пробы кДНК [[Комплементарность (биология)|комплементарно]] связываются с ДНК, произошедшей от экзонов, которые присутствуют в мРНК из исследуемой ткани, или с ДНК от места соединения двух экзонов. Благодаря этому методу можно определить присутствие определённых альтернативно сплайсированных мРНК<ref name=Pan2004>{{cite pmid|15610736}}</ref>. |
|||
Фермент ацетилхолинэстераза (AChE) собирается из серии белков. В ходе альтернативного сплайсинга может получаться три изоформы белка, которые вызывают такие нейродегенеративные заболевания, как [[болезнь Альцгеймера]] и [[болезнь Паркинсона]]. Разбалансировка регуляторов сплайсинга может приводить к избытку AChE-R, изоформе AChE, найденной у пациентов, страдающих болезнью Альцгеймера.<ref name=ReferenceC /> |
|||
Метод CLIP ({{lang-en|Cross-linking and immunoprecipitation}} — образование поперечных сшивок и [[иммунопреципитация]]) использует [[УФ-излучение]] для образования сшивок между белками и РНК, подвергающихся сплайсингу. Затем ''транс''-действующие регуляторные белки сплайсинга осаждаются при помощи специальных [[Антитела|антител]]. Когда РНК, связанная с белком, изолируется и клонируется, то определяется последовательность РНК, связанная с регуляторным белком<ref name=David/>. Использование репортерных генов позволяет определить белки сплайсинга, участвующие в специфичных случаях альтернативного спайсинга: в зависимости от того, каким образом прошёл сплайсинг, репортерный ген будет давать начало двум разным [[Флуоресцентный белок|флуоресцентным белкам]]. Этот метод был использован для изоляции [[мутант]]ов с нарушенным сплайсингом, а значит, и определения регуляторных белков сплайсинга, инактивированных у этих мутантов<ref name=David>{{cite pmid|18245441}}</ref>. |
|||
== См. также == |
|||
* [[Секвенирование РНК]] |
|||
== Примечания == |
== Примечания == |
||
{{примечания}} |
{{примечания|2}} |
||
== Ссылки == |
== Ссылки == |
||
Версия от 19:18, 23 марта 2017

Альтернати́вный спла́йсинг — процесс в экспрессии генов, приводящий к образованию нескольких белков с одного гена. В ходе этого процесса отдельные экзоны могут быть включены в зрелую мРНК или же отброшены[1]. В результате белки, считанные с мРНК одного гена, подвергшихся альтернативному сплайсингу, будут иметь разные аминокислотные последовательности и нередко и разные функции. Благодаря альтернативному сплайсингу в человеческом геноме закодировано гораздо больше белков, чем можно было бы ожидать с его 20 тысяч белоккодирующих генов.
Альтернативный сплайсинг широко распространен у эукариот, значительно увеличивая разнообразие белков, закодированных в их геномах. У человека около 95 % многоэкзонных генов подвергаются альтернативному сплайсингу[2]. Существует много разновидностей альтернативного сплайсинга, из которых наиболее часто встречается пропуск экзонов. При пропуске экзонов отдельные экзоны в некоторых тканях или при определённых условиях могут быть включены в зрелую мРНК или пропущены[1].
Образование альтернативно сплайсированных мРНК находится под контролем системы транс-действующих белков, которые связываются с цис-сайтами самого первичного транскрипта. К этим белкам относятся активаторы сплайсинга, которые способствуют использованию отдельных сайтов сплайсинга, а также репрессоры сплайсинга, которые выключают использование некоторых сайтов сплайсинга. Механизмы альтернативного сплайсинга очень разнообразны, и постоянно открываются новые примеры, в частности, с помощью высокопроизводительных методов. Учёные работают над тем, чтобы полностью изучить все механизмы сплайсинга, чтобы с помощью «кода сплайсинга» можно было предсказывать продукты альтернативного сплайсинга для данного гена в определённых условиях[3][4].
Ненормальные случаи альтернативного сплайсинга нередко приводят к болезням; значительное количество генетических заболеваний человека возникает из-за альтернативного сплайсинга[3]. Нарушенный сплайсинг может приводить к развитию рака, и в различных видах рака гены факторов сплайсинга нередко мутируют[5][6][7][8][8].
История изучения
Впервые альтернативный сплайсинг был описан в 1977 году[9][10]. Аденовирус образует пять различных транскриптов в ранней стадии инфекционного цикла, до репликации вирусной ДНК, и ещё один после начала репликации ДНК. После начала репликации ДНК продолжают образовываться ранние первичные транскрипты. Дополнительный одиночный транскрипт, образуемый на поздних стадиях инфекционного цикла, очень крупный и считывается с 5/6 аденовирусного генома размером 32 килобазы. Он гораздо крупнее каждого из вирусных транскриптов, образованных в заражённой клетке на ранних стадиях инфекционного цикла. Учёные показали, что первичный транскрипт, образуемый аденовирусом типа 2 на поздних стадиях инфекции, подвергается сплайсингу разными способами, в результате чего образуются различные мРНК, кодирующие разные вирусные белки. Кроме того, первичный транскрипт содержит множество сайтов полиаденилирования, в результате чего у разных мРНК могут получаться разные 3'-концы[11][12][13].
В 1981 году альтернативный сплайсинг был описан у обычного клеточного гена. Было показано, что в клетках млекопитающих гормон кальцитонин образуется в результате альтернативного сплайсинга. Первичный транскрипт гена кальцитонина содержит 6 экзонов; в зрелую мРНК, кодирующую кальцитонин, входят экзоны 1—4, и сигнал полиаденилирования находится в экзоне 4. У другой мРНК, образуемой из того же первичного транскрипта, при сплайсинге экзон 4 пропускается, и зрелая мРНК содержит экзоны 1—3, 5 и 6. Она кодирует белок, известный как CGRP[англ.] (англ. calcitonin gene related peptide)[14][15]. В начале 1980-х также был открыт альтернативный сплайсинг в генах иммуноглобулинов млекопитающих[11][16].
С тех пор было показано, что альтернативный сплайсинг распространён среди всех эукариот[1]. Рекорд по количеству белков, образуемых с одного и того же гена, принадлежит гену плодовой мушки Drosophila melanogaster, известному как Dscam[англ.]. Он имеет 38016 разных вариантов сплайсинга[17].
Модели

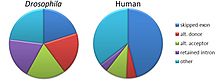
Существует пять моделей протекания альтернативного сплайсинга[1][2][3][18]:
- Пропуск экзона или кассетный экзон. В этом случае экзон может быть включен в состав зрелой мРНК или вырезан. Это наиболее распространённая модель протекания альтернативного сплайсинга у пре-мРНК млекопитающих[18].
- Взаимоисключающие экзоны. Из двух экзонов в зрелой мРНК находится только один, но не оба.
- Альтернативный донорный сайт. Используется альтернативный 5'-конец интрона (донорный сайт), так что меняется 3'-конец вышестоящего экзона.
- Альтернативный акцепторный сайт. Используется альтернативный 3'-конец интрона (акцепторный сайт), так что меняется 5'-конец нижестоящего экзона.
- Сохранение интрона. Последовательность может быть вырезана как интрон или оставлена в зрелой мРНК. Этот способ отличается от пропуска экзона, поскольку сохраняемая последовательность не фланкирована интронами. Если оставленный интрон попадает в кодирующую область?!, то он должен кодировать аминокислотную последовательность с такой же рамкой считывания, как и соседние экзоны. Если он будет содержать стоп-кодон или будет иметь другую рамку считывания, нежели окружающие экзоны, то произойдёт сдвиг рамки считывания и белок будет нефункциональным. Это самый редкий путь альтернативного сплайсинга у млекопитающих[18].
Кроме этих пяти основных моделей протекания альтернативного сплайсинга, существуют два способа получения нескольких белков с одного гена: использования множественных промоторов и множественных сайтов полиаденилирования. Использование множественных промоторов относится скорее к регуляции транскрипции, чем к альтернативному сплайсингу. Начиная транскрипцию с разных точек, можно получить транскрипты с различающимися 5'-концевыми экзонами. С другой стороны, использование множественных сайтов полиаденилирования приведёт к образованию разных 3'-концов у созревающих транскриптов. Оба этих механизма в сочетании с пятью моделями сплайсинга обеспечивают разнообразие мРНК, считываемых с одного и того же гена[1][3].

Эти модели хорошо описывают базовые механизмы сплайсинга, однако они могут не подходить для сложных случаев. Например, на рисунке справа представлены три формы мышиного гена гиалуронидазы 3, полученные в ходе аналогичного сплайсинга. Сравнение экзонов первой формы (зелёная) и второй (жёлтая) указывает на то, что имело место сохранение интрона, а сравнение второй формы с третьей (синяя) показывает, что произошёл пропуск экзона[18].
Механизм
Общая схема сплайсинга

Когда пре-мРНК транскрибируется с ДНК, она содержит несколько интронов и экзонов. Так, у нематод обычно имеется 4—5 интронов и экзонов, а у дрозофилы может быть более 100 интронов и экзонов в одной пре-мРНК. В ходе сплайсинга экзоны должны быть оставлены в транскрипте, а интроны удалены. Регуляция и выбор сайтов сплайсинга осуществляется транс-действующими белками-активаторами и репрессорами сплайсинга, а также цис-действующими элементами в самой пре-мРНК, такими как энхансеры и сайленсеры сплайсинга. Типичные эукариотические интроны имеют консенсусные последовательности?! в определённых местах; так, на 5'-конце каждого интрона располагается динуклеотид GU. Рядом с 3'-концом находится точка ветвления, и в точке ветвления всегда находится нуклеотид А, а вокруг него последовательности несколько варьируют. У человека консенсусная последовательность вокруг точки ветвления выглядит как yUnAy[19]. После точки ветвления располагается ряд пиримидинов (полипиримидиновый тракт[англ.]), 3'-конец которого выглядит как AG[3].
Сплайсинг пре-мРНК осуществляет РНК-белковый комплекс, известный как сплайсосома. В состав сплайсосомы входят малые ядерные рибонуклеопротеиды[англ.] (snRNP), обозначаемые U1[англ.], U2[англ.], U4[англ.], U5[англ.] и U6[англ.] (U3 не участвует в сплайсинге мРНК)[20]. U1 связывается с 5'-концевым динуклеотидом GU, а U2, при участии белковых факторов U2AF, связывается с точкой ветвления. На этой стадии комплекс известен как сплайсосомный А-комплекс. Во время формирования А-комплекса определяются 5'- и 3'-границы удаляемого интрона, а также концы экзонов, которые должны быть оставлены[3] .
Далее с А-комплексом связывается комплекс U4, U5, U6. После этого U6 замещает U1, а U1 и U4 покидает комплекс. Оставшийся комплекс осуществляет две реакции пересэтерификации. В ходе первой реакции 5'-конец интрона отрезается от вышележащего экзона и присоединяется к нуклеотиду А в точке ветвления с помощью 2',5'-фосфодиэфирной связи. В ходе второй реакции отрезается 3'-конец интрона, и два экзона соединяются фосфодиэфирной связью. Интрон, принимающий форму лассо, высвобождается и далее деградирует[1].
Регуляторные элементы и белки

Сплайсинг регулируется транс-действующими белками (активаторами и репрессорами) и соответствующими цис-регуляторными элементами (сайленсерами и энхансерами) на пре-мРНК. Впрочем, имеются данные, что во многих случаях действие фактора сплайсинга зависит от положения. Когда фактор сплайсинга связан с интронным энхансерным элементом, он действует как активатор сплайсинга, а когда связывается с регуляторным сайтом в экзоне, действует как репрессор, и наоборот[21]. В регуляции сплайсинга также принимает участие вторичная структура пре-мРНК, сближающая друг с другом два регуляторных элемента или маскируя последовательности, которые могли бы служить местами связывания факторов сплайсинга[22][23]. Вместе все эти элементы образуют «код сплайсинга», который определяет, как будет проходить сплайсинг в данных клеточных условиях[24][25].
Имеется два типа цис-активирующих элементов в пре-мРНК, и им соответствуют транс-активирующие РНК-связывающие белки. Сайленсеры сплайсинга — это элементы, с которыми связываются белки-репрессоры сайленсинга, при этом снижая вероятность того, что по соседству будет находиться сайт сплайсинга. Сайленсеры сплайсинга могут находиться в интронах (интронные сайленсеры сплайсинга, ISS) или в экзонах (экзонные сайленсеры сплайсинга, ESS). Их последовательности, как и связывающиеся белки, сильно разнятся. Большинство репрессоров сплайсинга являются гетерогенными ядерными рибонуклеопротеидами[англ.] (hnRNP), такие как hnRNPA1[англ.] и белок, связывающий полипиримидиновый тракт (PTB). С энхансерами сплайсинга связываются белки-активаторы сплайсинга, увеличивая вероятность того, что рядом будет находиться сайт сплайсинга. Они тоже могут находиться в интронах (интронные энхансеры сплайсинга, ISE) или в экзонах (экзонные энхансеры сплайсинга, ESE). Большая часть белков, связывающихся с ISE и ESE, относится к семейству белков SR. Эти белки содержат мотивы, распознающие РНК, а также домены, обогащённые аргинином и серином[3]Ошибка в сносках?: Неправильный вызов: неверные ключи, например было указано слишком много ключей или ключ был неправильным. Наличие определённых цис-регуляторных последовательностей РНК увеличивают вероятность того, что рядом будет находиться сайт сплайсинга, а в других уменьшают эту вероятность в зависимости от контекста. Например, некоторые такие элементы влияют на сплайсинг только тогда, когда рядом располагается ряд определённых элементов. Кроме того, цис-регуляторные элементы могут давать разный эффект при экспрессии в клетке определённых белков. Адаптивное значение энхансеров и сайленсеров сплайсинга подтверждается работами, показывающими, что в человеческих генах на мутации, приводящие к образованию новых сайленсеров или разрушению старых энхансеров, действует строгий отбор[26][27].
Примеры
Пропуск экзона: ген dsx дрозофилы
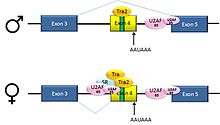
Пре-мРНК гена dsx[англ.] D. melanogaster содержит 6 экзонов. У самцов в зрелую мРНК входят экзоны 1, 2, 3, 5, 6, и они кодируют белок, который функционирует как регулятор транскрипции в развитии по мужскому типу. У самок в зрелую мРНК входят экзоны 1, 2, 3 и 4, и экзон 4 содержит сигнал полиаденилирования, по которому мРНК разрезается. Получившийся белок функционирует как регулятор транскрипции в развитии по женскому типу[28].
В описанном примере имеет место альтернативный сплайсинг по типу пропуска экзона. Интрон, лежащий выше экзона 4, содержит полипиримидиновый тракт, который не вполне удовлетворяет консенсусной последовательности сплайсинга, поэтому белки U2AF связываются с ним плохо в отсутствие активаторов сплайсинга. По этой причине у самцов этот 3'-акцепторный сайт сплайсинга не используется. У самок, однако, имеется активатор сплайсинга Transformer (Tra). SR-белок Tra2 образуется у обоих полов и связывается с ESE в экзоне 4; если имеется Tra, то он связывается с Tra2 и вместе с ещё одним SR-белком формирует комплекс, который содействует связыванию белков U2AF со слабым пиримидиновым трактом. U2 рекрутируется к соответствующей точке ветвления, и в результате экзон 4 включается в состав зрелой мРНК[28][29].
Альтернативные акцепторные сайты: Transformer дрозофилы

Пре-мРНК гена Transformer (Tra) D. melanogaster подвергаются альтернативному сплайсингу по модели альтенативных акцепторных сайтов. Ген Tra кодирует белок, экспрессирующийся только у самок. Первичный транскрипт этого гена содержит интрон с двумя возможными акцепторными сайтами. У самцов используется вышележащий акцепторный сайт. В итоге в мРНК включается удлинённый вариант экзона 2, содержащий преждевременный стоп-кодон. Поэтому у самцов образуется укороченный неактивный белок. У самок образуется белок, играющий ключевую роль в определении пола и известный как Sex lethal (Sxl). Белок Sxl является репрессором сплайсинга, который связывается с ISS в РНК-транскрипте Tra рядом с вышележащим акцепторным сайтом, предотвращая связывание белка U2AF с полипиримидиновым трактом. В результате сплайсосома связывается с нижележащим акцепторным сайтом. При использовании этого сайта преждевременный стоп-кодон удаляется в составе интрона. Получившаяся мРНК кодирует белок Tra, который сам выступает регулятором альтернативного сплайсинга других связанных с полом генов (см. выше пример гена dsx)[1].
Альтернативный сплайсинг рецептора Fas
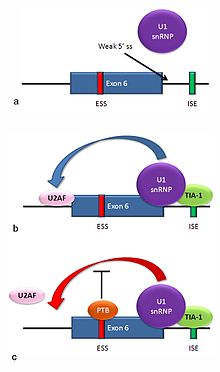
Множество изоформ рецептора Fas[англ.] образуются в результате альтернативного сплайсинга. Две нормальные изоформы у человека образуются по механизму пропуска экзона. мРНК, содержащая 6 экзонов, кодирует мембраносвязанную форму рецептора Fas, которая стимулирует апоптоз. Повышенное образование рецептора Fas в клетках, постоянно подвергающихся воздействию солнечного света, и отсутствие этого рецептора в клетках рака кожи свидетельствует о том, что этот механизм играет важную роль в элиминации клеток, которые встали на путь превращения в раковые[30]. Если экзон 6 пропускается, то образуется водорастворимая изоформа белка Fas, которая неспособна стимулировать апоптоз. Вставка или пропуск экзона зависят от действия двух белков-антагонистов TIA-1[англ.] и PTB.
Донорный сайт, расположенный на 5'-конце интрона, следующим после экзона 6 в пре-мРНК, плохо согласуется с консенсусной последовательностью сплайсинга, и не всегда связывается с snRNP U1[3]. Если U1 не связывается, экзон пропускается (картинка а на рисунке справа). Связывание белка TIA-1 с интронным энхансером сплайсинга стабилизирует связывание U1. Образующийся на 5'-конце интрона донорный сайт помогает связываться фактору сплайсинга U2AF с 3'-сайтом сплайсинга, расположенным выше экзона, хотя механизм этого ещё не понятен (картинка b на рисунке справа)[31]. Экзон 6 содержит обогащённый пиримидинами сайленсер сплайсинга (ure6), с которым может связываться PTB. Если связывание PTB происходит, то донорный сайт на 5'-конце интрона не способствует связыванию фактора U2AF, и экзон пропускается (картинка с на рисунке справа).
Описанный выше механизм является примером определения экзона при сплайсинге. Сплайсосома собирается в области интрона, и snRNP так укладывают РНК, что 5'- и 3'-концы интрона соединяются. Однако в описанном выше случае происходит также взаимодействие концов экзона. В этом случае взаимодействия, определяющие границы экзона, необходимы для связывания коровых факторов сплайсинга до сборки сплайсосомы на границах фланкирующих интронов[31].
Конкуренция репрессора и активатора: экзон 2 гена tat ВИЧ-1

ВИЧ — ретровирус, являющийся причиной развития СПИДа — образует единственную пре-мРНК, из которой далее посредством альтернативного сплайсинга образуется более 40 различных мРНК[32]. Равновесие между по-разному сплайсированными транскриптами обеспечивает образование мРНК, кодирующих все белки, необходимые для репликации вируса[33]. Один из по-разному сплайсированных транскриптов содержит транскрипт гена tat[англ.], у которого экзон 2 является кассетным, то есть может быть включён в итоговый транскрипт или не включён. Включение этого экзона регулируется репрессором сплайсинга hnRNP A1 и SR-белком[англ.] SC35. В экзоне 2 сайленсерная последовательность (ESS) и энхансерная последовательность (ESE) перекрываются. Если с ESS связывается репрессор А1, то он запускает кооперативное связывание молекул А1, закрывая 5'-концевой донорный сайт выше экзона 2 и препятствуя связыванию U2AF35 с полипиримидиновым трактом. Если SC35 связывается с ESE, то он препятствует связыванию А1, и 5'-донорный сайт остаётся доступным для сплайсосомы. Конкуренция между репрессором и активатором приводит к образованию РНК, содержащей или не содержащей экзон 2[32].
Адаптивное значение
Альтернативный сплайсинг — это одно из исключений из правила, что одному гену соответствует один белок (гипотеза «один ген — один фермент»)[34]. Корректнее было бы сказать, «один ген — много полипептидов». Внешняя информация нужна для того, чтобы решить, какой именно полипептид образовать с данной мРНК. Поскольку способы регуляции наследуются, то это даёт мутациям новый путь к изменению экспрессии генов[7].
Предполагается, что для эукариот альтернативный сплайсинг — очень важный шаг на пути к повышению эффективности, поскольку он даёт возможность хранить информацию более экономно. Один ген может давать начало нескольким белкам, а не один ген даёт начало только одному белку, поэтому одно и то же разнообразие протеома можно получить с генома существенно меньшего размера[1]. Это также обеспечивает эволюционную гибкость. Из-за единственной точечной мутации может привести к включению или исключению экзона из транскрипта, благодаря чему может быть получена новая изоформа белка без потери его основной формы[1]. Обнаружены действительно неупорядоченные регионы, которые содержат много неконститутивных экзонов, поэтому изоформы белка может выполнять новые функции, изменяя функциональные модули в этих местах[35][36][37]. Сравнительные оценки показывают, что возникновение альтернативного сплайсинга в ходе эволюции предшествовало появлению многоклеточности, и предполагается, что альтернативный сплайсинг был одним из средств, обеспечивающих возникновение многоклеточных организмов[38].
Исследования в рамках проекта «Геном человека», а также других проектов по секвенированию геномов показали, что геном человека всего лишь на 30% больше генома круглого червя Caenorhabditis elegans, и всего лишь в два раза больше, чем у плодовой мушки Drosophila melanogaster. Эти данные наводят на мысль, что сложность человека и позвоночных животных вообще может быть связана с более активным, по сравнению с беспозвоночными, использованию альтернативного сплайсинга[39][40]. Однако дальнейшее изучение последовательностей человека, мыши, крысы, коровы, D. melanogaster, C. elegans и растения Arabidopsis thaliana показало, что между человеком и другими животными не наблюдается значительной разнице в использовании альтернативного сплайсинга[41]. Впрочем, имеются сведения, что полученные данные были артефактом. При сравнении частоты использования альтернативного сплайсинга случайного набора генов каждого организма оказалось, что позвоночные всё-таки прибегают к альтернативному сплайсингу чаще беспозвоночных[42].
Клиническое значение
Изменения в машинерии процессинга РНК могут приводить к нарушениям сплайсинга многих транскриптов, а однонуклеотидные замены в сайтах сплайсинга или цис-регуляторных сайтах сплайсинга приводят к различиям в сплайсинге одного и того же гена, как и при спайсинге транскрипта мутировавшего гена. В работе 2005 года было показано, что свыше 60 % мутаций, приводящих к развитию болезней, влияют не на саму кодирующую последовательность, а на сплайсинг[43]. Показано также, что треть наследственных заболеваний связана со сплайсингом[21].
Ненормально сплайсированные клетки встречаются в заметной доле раковых клеток[5][6][8]. Анализ RNA-Seq и протеомов показал поразительно дифференциальную экспрессию сплайсинговых изоформ белков, участвующих в сигнальных путях, связанных с развитием рака[44]. Неизвестно, влияют ли нарушения сплайсинга на развития рака напрямую, или же они являются следствием поломки клеточных процессов в связи с переходом к раковому росту. Интересно, что при некоторых видах рака, такой как рак толстой кишки или рак простаты, количество ошибок в сплайсинге значительно варьировало у разных пациентов; этот феномен назвали транскриптомной нестабильностью[45][46]. Далее было показано, что транскриптомная нестабильность связана с пониженной экспрессией генов факторов сплайсинга. Действительно, в общем случае в раковых клетках альтернативный сплайсинг используется меньше, чем у нормальных клеток, причём модели сплайсинга тоже различаются. Так, в раковых клетках сохранение интрона происходит чаще, чем в нормальных клетках, а пропуск экзона — реже. Особенности сплайсинга в раковых клетках могут быть связаны с высокой частотой соматических мутаций в генах факторов сплайсинга, а некоторые особенности могут быть обусловлены изменениями в фосфорилировании транс-регуляторных факторов сплайсинга[47][7]. Некоторые особенности могут быть связаны с изменениями относительного количества факторов сплайсинга; например, в клетках рака груди наблюдаются повышенные уровни фактора сплайсинга SF2/ASF[англ.][48]. В одном исследовании было показано, что относительно небольшая доля (383 из 26000) вариантов альтернативного сплайсинга в раковых клетках встречалась сильно чаще, чем в нормальных, поэтому существует ограниченное количество генов, неправильный сплайсинг которых приводит к развитию опухоли[49]. Считается, однако, что губительное действие нарушенного сплайсинга сдерживается особым клеточным посттранскрипционным механизмом контроля — нонсенс-опосредованным распадом[50].
Одним из человеческих генов, специфический вариант сплайсинга которого связан с развитием рака, является один из генов DNMT. Три гена DNMT кодируют ферменты, которые добавляют метильные группы к ДНК, и эта модификация часто имеет регуляторные эффекты. Несколько неправильно сплайсированных мРНК гена DNMT3B[англ.] были найдены в опухолях и клетках раковых линий[англ.]. Экспрессия двух из этих мРНК вызывала изменения в метилировании ДНК в этих клетках. Клетки с одной ненормальной мРНК росли вдвое быстрее, чем контрольные клетки, поэтому эти мРНК связаны с развитием рака[7].
Другим примером может служить протоонкоген Ron (MST1R?!). Важным свойством раковых клеток является их способность переселяться в нормальные ткани и нарушать их работу. Образование ненормально сплайсированной мРНК Ron было связано с повышенными уровнями SF2/ASF в клетках рака груди. Ненормальная изоформа Ron, образованная с этой мРНК, увеличивала подвижность клеток[48].
Сверхэкспрессия укороченного варианта белка FOSB[англ.] — ΔFosB — в специфической популяции нейронов прилежащего ядра лежит в основе возникновения и поддержания привыкания к наркотикам и естественному вознаграждению (англ. natural reward)[51][52][53][54].
Недавние провокационные исследования показывают на роль структуры хроматина и модификаций гистонов в регуляции альтернативного сплайсинга. Поэтому эпигенетические факторы могут влиять не только на экспрессию генов, но и на их сплайсинг[55].
Полногеномный анализ
Полногеномный анализ альтернативного сплайсинга — сложная задача. Обычно альтернативно сплайсированные транскрипты обнаруживают при сравнении экспрессируемых последовательностей-меток[англ.] (англ. Expressed sequence tag, EST). Большинство библиотек EST[англ.] собраны из очень ограниченного числа тканей, поэтому тканеспецифичные транскрипты не были учтены. Однако появились высокопроизводительные методы для изучения сплайсинга, такие как ДНК-микрочипы и глубокое секвенирование[англ.] (англ. deep sequencing). Эти методы могут быть использованы для поиска полиморфизмов и мутаций, расположенных в или вокруг элементов сплайсинга, которые влияют на связывание белков. При сочетании с такими методами изучения сплайсинга, как in vitro анализ репортерных генов, могут быть исследованы функциональные эффекты полиморфизмов и мутаций на сплайсинг пре-мРНК[21][24][56].
При анализе с помощью микрочипов были использованы фрагменты ДНК, являющиеся отдельными экзонами (такие как микрочип Affymetrix[англ.]) или границами между экзонами. Затем в микрочип добавляется меченая кДНК из интересующей ткани. Пробы кДНК комплементарно связываются с ДНК, произошедшей от экзонов, которые присутствуют в мРНК из исследуемой ткани, или с ДНК от места соединения двух экзонов. Благодаря этому методу можно определить присутствие определённых альтернативно сплайсированных мРНК[57].
Метод CLIP (англ. Cross-linking and immunoprecipitation — образование поперечных сшивок и иммунопреципитация) использует УФ-излучение для образования сшивок между белками и РНК, подвергающихся сплайсингу. Затем транс-действующие регуляторные белки сплайсинга осаждаются при помощи специальных антител. Когда РНК, связанная с белком, изолируется и клонируется, то определяется последовательность РНК, связанная с регуляторным белком[4]. Использование репортерных генов позволяет определить белки сплайсинга, участвующие в специфичных случаях альтернативного спайсинга: в зависимости от того, каким образом прошёл сплайсинг, репортерный ген будет давать начало двум разным флуоресцентным белкам. Этот метод был использован для изоляции мутантов с нарушенным сплайсингом, а значит, и определения регуляторных белков сплайсинга, инактивированных у этих мутантов[4].
Примечания
- ↑ 1 2 3 4 5 6 7 8 9 Black D. L. Mechanisms of Alternative Pre-Messenger RNA Splicing // Annual Review of Biochemistry. — 2003. — Vol. 72. — P. 291—336. — doi:10.1146/annurev.biochem.72.121801.161720. — PMID 12626338.
- ↑ 1 2 Pan Qun, Shai O., Lee L. J., Frey B. J., Blencowe B. J. Deep Surveying of Alternative Splicing Complexity in the Human Transcriptome by High-Throughput Sequencing // Nature Genetics. — 2008. — Vol. 40, no. 12. — P. 1413—1415. — doi:10.1038/ng.259. — PMID 18978789.
- ↑ 1 2 3 4 5 6 7 8 9 Matlin A. J., Clark F., Smith C. W. Understanding Alternative Splicing: Towards a Cellular Code // Nature Reviews. Molecular Cell Biology. — 2005. — Vol. 6, no. . — P. 386—398. — doi:10.1038/nrm1645. — PMID 15956978.
- ↑ 1 2 3 David C. J., Manley J. L. The Search for Alternative Splicing Regulators: New Approaches Offer a Path to a Splicing Code // Genes & Development. — 2008. — Vol. 22, no. 3. — P. 279—285. — doi:10.1101/gad.1643108. — PMID 18245441.
- ↑ 1 2 Skotheim R. I., Nees M. Alternative Splicing in Cancer: Noise, Functional, or Systematic? // The International Journal of Biochemistry & Cell Biology. — 2007. — Vol. 39, no. 7-8. — P. 1432—1449. — doi:10.1016/j.biocel.2007.02.016. — PMID 17416541.
- ↑ 1 2 He Chunjiang, Zhou Fang, Zuo Zhixiang, Cheng Hanhua, Zhou Rongjia. A Global View of Cancer-Specific Transcript Variants by Subtractive Transcriptome-Wide Analysis // PLoS One. — 2009. — Vol. 4, no. 3. — P. e4732. — doi:10.1371/journal.pone.0004732. — PMID 19266097.
- ↑ 1 2 3 4 Fackenthal J. D., Godley L. A. Aberrant RNA Splicing and its Functional Consequences in Cancer Cells // Disease Models & Mechanisms. — 2008. — Vol. 1, no. 1. — P. 37—42. — doi:10.1242/dmm.000331. — PMID 19048051.
- ↑ 1 2 3 Sveen A., Kilpinen S., Ruusulehto A., Lothe R. A., Skotheim R. I. Aberrant RNA Splicing in Cancer; Expression Changes and Driver Mutations of Splicing Factor Genes // Oncogene. — 2016. — Vol. 35, no. 19. — P. 2413—2427. — doi:10.1038/onc.2015.318. — PMID 26300000.
- ↑ Chow L. T., Gelinas R. E., Broker T. R., Roberts R. J. An Amazing Sequence Arrangement at the 5' Ends of Adenovirus 2 messenger RNA // Cell. — 1977. — Vol. 12, no. 1. — P. 1—8. — doi:10.1016/0092-8674(77)90180-5. — PMID 902310.
- ↑ Berget S. M., Moore C., Sharp P. A. Spliced Segments at the 5' Terminus of Adenovirus 2 Late mRNA // Proc. Nat. Acad. Sci. USA. — 1977. — Vol. 74, no. 8. — P. 3171—3175. — PMID 269380.
- ↑ 1 2 Leff S. E., Rosenfeld M. G., Evans R. M. Complex Transcriptional Units: Diversity in Gene Expression by Alternative RNA Processing // Annual Review of Biochemistry. — 1986. — Vol. 55. — P. 1091—1117. — doi:10.1146/annurev.bi.55.070186.005303. — PMID 3017190.
- ↑ Chow L. T., Broker T. R. The Spliced Structures of Adenovirus 2 Fiber Message and the Other Late mRNAs // Cell. — 1978. — Vol. 15, no. 2. — P. 497—510. — doi:10.1016/0092-8674(78)90019-3. — PMID 719751.
- ↑ Nevins J. R., Darnell J. E. Steps in the Processing of Ad2 mRNA: Poly(A)+ Nuclear Sequences Are Conserved and Poly(A) Addition Precedes Splicing // Cell. — 1978. — Vol. 15, no. 4. — P. 1477—1493. — doi:10.1016/0092-8674(78)90071-5. — PMID 729004.
- ↑ Rosenfeld M. G., Amara S. G., Roos B. A., Ong E. S., Evans R. M. Altered Expression of the Calcitonin Gene Associated with RNA Polymorphism // Nature. — 1981. — Vol. 290, no. 5801. — P. 63—65. — PMID 7207587.
- ↑ Rosenfeld M. G., Lin C. R., Amara S. G., Stolarsky L., Roos B. A., Ong E. S., Evans R. M. Calcitonin mRNA Polymorphism: Peptide Switching Associated with Alternative RNA Splicing Events // Proc. Nat. Acad. Sci. USA. — 1982. — Vol. 79, no. 6. — P. 1717—1721. — PMID 6952224.
- ↑ Maki R., Roeder W., Traunecker A., Sidman C., Wabl M., Raschke W., Tonegawa S. The Role of DNA Rearrangement and Alternative RNA Processing in the Expression of Immunoglobulin Delta Genes // Cell. — 1981. — Vol. 24, no. 2. — P. 353—365. — doi:10.1016/0092-8674(81)90325-1. — PMID 6786756.
- ↑ Schmucker D., Clemens J. C., Shu Huidy, Worby C. A., Xiao Jian, Muda M., Dixon J. E., Zipursky S. L. Drosophila Dscam Is an Axon Guidance Receptor Exhibiting Extraordinary Molecular Diversity // Cell. — 2000. — Vol. 101, no. 6. — P. 671—684. — doi:10.1016/S0092-8674(00)80878-8. — PMID 10892653.
- ↑ 1 2 3 4 5 Sammeth M., Foissac S., Guigó R. A General Definition and Nomenclature for Alternative Splicing Events // PLOS Computational Biology. — 2008. — Vol. 4, no. 8. — P. e1000147. — doi:10.1371/journal.pcbi.1000147. — PMID 18688268.
- ↑ Gao Kaiping, Masuda A., Matsuura T,, Ohno K. Human Branch Point Consensus Sequence is yUnAy // Nucleic Acids Research. — 2008. — Vol. 36, no. 7. — P. 2257—2267. — doi:10.1093/nar/gkn073. — PMID 18285363.
- ↑ Clark, David. Molecular biology. — Amsterdam : Elsevier Academic Press, 2005. — ISBN 0-12-175551-7.
- ↑ 1 2 3 Lim Kian Huat, Ferraris L., Filloux M. E., Raphael B. J., Fairbrother W. G. Using Positional Distribution to Identify Splicing Elements and Predict pre-mRNA Processing Defects in Human Genes // Proc. Nat. Acad. Sci. USA. — 2011. — Vol. 108, no. 27. — P. 11093—11098. — doi:10.1073/pnas.1101135108. — PMID 21685335.
- ↑ Warf M. B., Berglund J. A. Role of RNA Structure in Regulating pre-mRNA Splicing // Trends in Biochemical Sciences. — 2010. — Vol. 35, no. 3. — P. 169—178. — doi:10.1016/j.tibs.2009.10.004. — PMID 19959365.
- ↑ PMID 198614269 (PMID 198614269)
Библиографическое описание появится автоматически через некоторое время. Вы можете подставить цитату вручную или используя бота. - ↑ 1 2 Wang Zefeng, Burge C. B. Splicing Regulation: from a Parts List of Regulatory Elements to an Integrated Splicing Code // RNA. — 2008. — Vol. 14, no. 5. — P. 802—813. — doi:10.1261/rna.876308. — PMID 18369186.
- ↑ Barash Y., Calarco J. A., Gao Weijun, Pan Qun, Wang Xinchen, Shai O., Blencowe B. J., Frey B. J. Deciphering the Splicing Code // Nature. — 2010. — Vol. 465, no. 7294. — P. 53—59. — doi:10.1038/nature09000. — PMID 20445623.
- ↑ Ke Shengdong, Zhang Xiang H.-F., Chasin L. A. Positive Selection Acting on Splicing Motifs Reflects Compensatory Evolution // Genome Research. — 2008. — Vol. 18, no. 4. — P. 533—543. — doi:10.1101/gr.070268.107. — PMID 18204002.
- ↑ Fairbrother W. G., Holste D., Burge C. B., Sharp P. A. Single Nucleotide Polymorphism-Based Validation of Exonic Splicing Enhancers // PLOS Biology. — 2004. — Vol. 2, no. 9. — P. e268. — doi:10.1371/journal.pbio.0020268. — PMID 15340491.
- ↑ 1 2 Lynch K. W., Maniatis T. Assembly of Specific SR Protein Complexes on Distinct Regulatory Elements of the Drosophila Doublesex Splicing Enhancer // Genes & Development. — 1996. — Vol. 10, no. 16. — P. 2089—2101. — doi:10.1101/gad.10.16.2089. — PMID 8769651.
- ↑ Graveley B. R., Hertel K. J., Maniatis T. The Role of U2AF35 and U2AF65 in Enhancer-Dependent Splicing // RNA. — 2001. — Vol. 7, no. 6. — P. 806—818. — PMID 11421359.
- ↑ Filipowicz E., Adegboyega P., Sanchez R. L., Gatalica Z. Expression of CD95 (Fas) in Sun-Exposed Human Skin and Cutaneous Carcinomas // Cancer. — 2002. — Vol. 94, no. 3. — P. 814—819. — doi:10.1002/cncr.10277. — PMID 11857317.
- ↑ 1 2 Izquierdo J. M., Majós N., Bonnal S., Martínez C., Castelo R., Guigó R., Bilbao D., Valcárcel J. Regulation of Fas Alternative Splicing by Antagonistic Effects of TIA-1 and PTB on Exon Definition // Molecular Cell. — 2005. — Vol. 19, no. 4. — P. 475—484. — doi:10.1016/j.molcel.2005.06.015. — PMID 16109372.
- ↑ 1 2 Zahler A. M., Damgaard C. K., Kjems J., Caputi M. SC35 and Heterogeneous Nuclear Ribonucleoprotein A/B Proteins Bind to a Juxtaposed Exonic Splicing Enhancer/Exonic Splicing Silencer Element to Regulate HIV-1 tat Exon 2 Splicing // The Journal of Biological chemistry. — 2004. — Vol. 279, no. 11. — P. 10077—10084. — doi:10.1074/jbc.M312743200. — PMID 14703516.
- ↑ Jacquenet S., Méreau A., Bilodeau P. S., Damier L., Stoltzfus C. M., Branlant C. A Second Exon Splicing Silencer within Human Immunodeficiency Virus Type 1 tat Exon 2 Represses Splicing of Tat mRNA and Binds Protein hnRNP H // The Journal of Biological Chemistry. — 2001. — Vol. 276, no. 44. — P. 40464—40475. — doi:10.1074/jbc.M104070200. — PMID 11526107.
- ↑ HHMI Bulletin September 2005: Alternative Splicing. www.hhmi.org. Дата обращения: 26 мая 2009. Архивировано 22 июня 2009 года.
- ↑ Romero P. R., Zaidi S., Fang Ya Yin, Uversky V. N., Radivojac P., Oldfield C. J., Cortese M. S., Sickmeier M., LeGall T., Obradovic Z., Dunker A. K. Alternative Splicing in Concert with Protein Intrinsic Disorder Enables Increased Functional Diversity in Multicellular Organisms // Proc. Nat. Acad. Sci. USA. — 2006. — Vol. 103, no. 22. — P. 8390—8395. — doi:10.1073/pnas.0507916103. — PMID 16717195.
- ↑ Li Hong-Dong, Menon R., Omenn G. S., Guan Yuanfang. The Emerging Era of Genomic Data Integration for Analyzing Splice Isoform Function // Trends in Genetics. — 2014. — Vol. 30, no. 8. — P. 340—347. — doi:10.1016/j.tig.2014.05.005. — PMID 24951248.
- ↑ Eksi R., Li Hong-Dong, Menon R., Wen Yuchen, Omenn G. S., Kretzler M., Guan Yuanfang. Systematically Differentiating Functions for Alternatively Spliced Isoforms through Integrating RNA-seq Data // PLOS Computational Biology. — 2013. — Vol. 9, no. 11. — P. e1003314. — doi:10.1371/journal.pcbi.1003314. — PMID 24244129.
- ↑ Irimia M., Rukov J. L., Penny D., Roy S. W. Functional and Evolutionary Analysis of Alternatively Spliced Genes Is Consistent with an Early Eukaryotic Origin of Alternative Splicing // BMC Evolutionary Biology. — 2007. — Vol. 7. — P. 188. — doi:10.1186/1471-2148-7-188. — PMID 17916237.
- ↑ Ewing B., Green P. Analysis of Expressed Sequence Tags Indicates 35,000 Human Genes // Nature Genetics. — 2000. — Vol. 25, no. 2. — P. 232—234. — doi:10.1038/76115. — PMID 10835644.
- ↑ Roest Crollius H., Jaillon O., Bernot A., Dasilva C., Bouneau L., Fischer C., Fizames C., Wincker P., Brottier P., Quétier F., Saurin W., Weissenbach J. Estimate of Human Gene Number Provided by Genome-Wide Analysis using Tetraodon nigroviridis DNA Sequence // Nature Genetics. — 2000. — Vol. 25, no. 2. — P. 235—238. — doi:10.1038/76118. — PMID 10835645.
- ↑ Brett D., Pospisil H., Valcárcel J., Reich J., Bork P. Alternative Splicing and Genome Complexity // Nature Genetics. — 2002. — Vol. 30, no. 1. — P. 29—30. — doi:10.1038/ng803. — PMID 11743582.
- ↑ Kim E., Magen A., Ast G. Different Levels of Alternative Splicing among Eukaryotes // Nucleic Acids Research. — 2007. — Vol. 35, no. 1. — P. 125—131. — doi:10.1093/nar/gkl924. — PMID 17158149.
- ↑ López-Bigas N., Audit B., Ouzounis C., Parra G., Guigó R. Are Splicing Mutations the Most Frequent Cause of Hereditary Disease? // FEBS Letters. — 2005. — Vol. 579, no. 9. — P. 1900—1903. — doi:10.1016/j.febslet.2005.02.047. — PMID 15792793.
- ↑ Omenn G. S., Guan Yuanfang, Menon R. A New Class of Protein Cancer Biomarker Candidates: Differentially Expressed Splice Variants of ERBB2 (HER2/neu) and ERBB1 (EGFR) in Breast Cancer Cell Lines // Journal of Proteomics. — 2014. — Vol. 107. — P. 103—112. — doi:10.1016/j.jprot.2014.04.012. — PMID 24802673.
- ↑ Sveen A., Johannessen B., Teixeira M. R., Lothe R. A., Skotheim R. I. Transcriptome Instability as a Molecular Pan-Cancer Characteristic of Carcinomas // BMC Genomics. — 2014. — Vol. 15. — P. 672. — doi:10.1186/1471-2164-15-672. — PMID 25109687.
- ↑ Sveen A., Agesen T. H., Nesbakken A., Rognum T. O., Lothe R. A., Skotheim R. I. Transcriptome Instability in Colorectal Cancer Identified by Exon Microarray Analyses: Associations with Splicing Factor Expression Levels and Patient Survival // Genome Medicine. — 2011. — Vol. 3, no. 5. — P. 32. — doi:10.1186/gm248. — PMID 21619627.
- ↑ Kim E., Goren A., Ast G. Insights into the Connection between Cancer and Alternative Splicing // Trends in Genetics. — 2008. — Vol. 24, no. 1. — P. 7—10. — doi:10.1016/j.tig.2007.10.001. — PMID 18054115.
- ↑ 1 2 Ghigna C., Giordano S., Shen Haihong, Benvenuto F., Castiglioni F., Comoglio P. M., Green M. R., Riva S., Biamonti G. Cell Motility is Controlled by SF2/ASF through Alternative Splicing of the Ron Protooncogene // Molecular Cell. — 2005. — Vol. 20, no. 6. — P. 881—890. — doi:10.1016/j.molcel.2005.10.026. — PMID 16364913.
- ↑ Hui Lijian, Zhang Xin, Wu Xin, Lin Zhixin, Wang Qingkang, Li Yixue, Hu Gengxi. Identification of Alternatively Spliced mRNA Variants Related to Cancers by Genome-Wide ESTs Alignment // Oncogene. — 2004. — Vol. 23, no. 17. — P. 3013—3023. — doi:10.1038/sj.onc.1207362. — PMID 15048092.
- ↑ Danckwardt S., Neu-Yilik G., Thermann R., Frede U., Hentze M. W., Kulozik A. E. Abnormally Spliced β-Globin mRNAs: a Single Point Mutation Generates Transcripts Sensitive and Insensitive to Nonsense-Mediated mRNA Decay // Blood. — 2002. — Vol. 99, no. 5. — P. 1811—1816. — PMID 11861299.
- ↑ Nestler E. J. Cellular Basis of Memory for Addiction // Dialogues in Clinical Neuroscience. — 2013. — Vol. 15, no. 4. — P. 431—443. — PMID 24459410.
- ↑ Ruffle J. K. Molecular Neurobiology of Addiction: What’s All the (Δ)FosB about? // The American Journal of Drug and Alcohol Abuse. — 2014. — Vol. 40, no. 6. — P. 428—437. — doi:10.3109/00952990.2014.933840. — PMID 25083822.
- ↑ Biliński P., Wojtyła A., Kapka-Skrzypczak L., Chwedorowicz R., Cyranka M., Studziński T. Epigenetic Regulation in Drug Addiction // Annals of Agricultural and Environmental Medicine. — 2012. — Vol. 19, no. 3. — P. 491—496. — PMID 23020045.
- ↑ Olsen C. M. Natural Rewards, Neuroplasticity, and Non-Drug Addictions // Neuropharmacology. — 2011. — Vol. 61, no. 7. — P. 1109—1122. — doi:10.1016/j.neuropharm.2011.03.010. — PMID 21459101.
- ↑ Luco R. F., Allo M., Schor I. E., Kornblihtt A. R., Misteli T. Epigenetics in Alternative pre-mRNA Splicing // Cell. — 2011. — Vol. 144, no. 1. — P. 16—26. — doi:10.1016/j.cell.2010.11.056. — PMID 21215366.
- ↑ Fairbrother W. G., Yeh R. F., Sharp P. A., Burge C. B. Predictive Identification of Exonic Splicing Enhancers in Human Genes // Science. — 2002. — Vol. 297, no. 5583. — P. 1007—1013. — doi:10.1126/science.1073774. — PMID 12114529.
- ↑ Pan Qun, Shai O., Misquitta C., Zhang Wen, Saltzman A. L., Mohammad N., Babak T., Siu H., Hughes T. R., Morris Q. D., Frey B. J., Blencowe B. J. Revealing Global Regulatory Features of Mammalian Alternative Splicing Using a Quantitative Microarray Platform // Molecular Cell. — 2004. — Vol. 16, no. 6. — P. 929—941. — doi:10.1016/j.molcel.2004.12.004. — PMID 15610736.
Ссылки
- Сплайсинг альтернативный: введение — «биология человека».