Многокопийная одноцепочечная ДНК
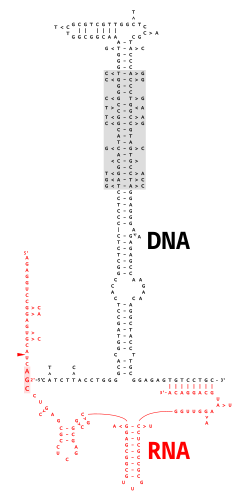
Многокопийная одноцепочечная ДНК (англ. Multicopy single-stranded DNA, msDNA) представляет собой тип внехромосомной сателлитной ДНК, состоящей из одноцепочечной молекулы ДНК, ковалентно связанной 2'-5' фосфодиэфирной связью с внутренним гуанозином молекулы РНК. Образовавшаяся в результате химера ДНК/РНК обладает двумя стеблями-петлями, соединёнными ветвью, аналогичной ветвям, обнаруживаемым в промежуточных продуктах сплайсинга РНК. Кодирующая область msDNA, называемая «ретрон», также кодирует тип обратной транскриптазы, которая необходима для синтеза msDNA[2].
Открытие[править | править код]
До открытия msDNA у миксобактерий[3][4], группы роящихся почвенных бактерий, считалось, что ферменты, известные как обратные транскриптазы (ОТ), существуют только у эукариот и вирусов. Открытие привело к увеличению исследований области. В результате было обнаружено, что msDNA широко распространена среди бактерий, включая различные штаммы Escherichia coli и патогенные бактерии[5]. Дальнейшие исследования обнаружили сходство между обратной транскриптазой, кодируемой ВИЧ, и открытой рамкой считывания (ORF), обнаруженной в кодирующей области msDNA. Тесты подтвердили наличие активности обратной транскриптазы в неочищенных лизатах ретронсодержащих штаммов[6]. Хотя домен РНКазы H был предварительно идентифицирован в ORF ретрона, позже было обнаружено, что активность РНКазы H, необходимая для синтеза мсДНК, фактически обеспечивается хозяином[7].
Ретроны[править | править код]
Открытие msDNA привело к более широким вопросам относительно происхождения обратной транскриптазы, поскольку гены, кодирующие обратную транскриптазу (не обязательно связанные с msDNA), были обнаружены у прокариот, эукариот, вирусов и даже архей. После того, как был обнаружен фрагмент ДНК, кодирующий продукцию мДНК в E. coli[8], было высказано предположение, что бактериофаги могли быть ответственны за введение гена RT в E. coli[9]. Эти открытия предполагают, что обратная транскриптаза сыграла роль в эволюции вирусов из бактерий, при этом одна из гипотез гласит, что с помощью обратной транскриптазы вирусы могли возникнуть как отколовшийся ген msDNA, приобретший белковую оболочку. Поскольку почти все гены RT участвуют в репликации ретровирусов и/или перемещении мобильных элементов, разумно предположить, что ретроны могут быть мобильными генетическими элементами, но для такой гипотезы имеется мало доказательств, за исключением наблюдаемого факта, что мсДНК широко, но спорадически распространяется среди видов бактерий, что свидетельствует как о горизонтальном, так и о вертикальном переносе[10][11][12]. Поскольку неизвестно, представляют ли последовательности ретронов сами по себе мобильные элементы, ретроны функционально определяются их способностью продуцировать msDNA, при этом преднамеренно избегая предположений о других возможных активностях.
Функция[править | править код]
Функция msDNA остается неизвестной, даже несмотря на то, что в клетках присутствует множество копий. Нокаутные мутации, которые не экспрессируют msDNA, жизнеспособны, поэтому производство msDNA не является необходимым для жизни в лабораторных условиях. Сверхэкспрессия msDNA является мутагенной, по-видимому, в результате титрования белков репарации несовпадающими парами оснований, типичными для их структуры[13]. Было высказано предположение, что msDNA может играть определённую роль в патогенности или адаптации к стрессовым условиям[14]. Сравнение последовательностей msDNA Myxococcus xanthus, Stigmatella aurantiaca[15], и многих других бактерий[10][14] выявило консервативные и гипервариабельные домены, напоминающие консервативные и гипервариабельные последовательности, обнаруженные в молекулах аллораспознавания[16]. Основные msDNA M. xanthus и S. aurantiaca, например, имеют 94 % гомологии последовательностей, за исключением домена из 19 пар оснований, который имеет гомологию последовательностей только 42 %[1]. Наличие таких доменов важно, поскольку миксобактерии демонстрируют сложное совместное социальное поведение, включая роение и образование плодовых тел, в то время как E. coli и другие патогенные бактерии образуют биоплёнки, проявляющие повышенную устойчивость к антибиотикам и детергентам. Устойчивость социальных собраний, требующих значительных индивидуальных затрат энергии, обычно зависит от эволюции механизмов всеопознания, которые позволяют группам различать себя от других[17].
Биосинтез[править | править код]

Предполагается, что биосинтез msDNA следует уникальному пути, не встречающемуся больше нигде в биохимии ДНК/РНК. Из-за сходства 2'-5' соединения ответвления с ответвлениями, обнаруженными в интермедиатах сплайсинга РНК, можно было сначала ожидать, что образование ответвления будет происходить посредством лигирования, опосредованного сплайсосомами или рибозимами. Неожиданно, однако, эксперименты в бесклеточных системах с использованием очищенной обратной транскриптазы Retron показали, что синтез кДНК напрямую инициируется от 2'-ОН группы специфического внутреннего остатка G праймерной РНК[18]. RT распознает специфические структуры «стебель-петля» в РНК-предшественнике, что делает синтез мсДНК с помощью RT высокоспецифичным для его собственного ретрона[19]. Подготовка к синтезу мДНК бросает вызов текущему пониманию синтеза ДНК. ДНК-полимеразы (которые включают RT) имеют общие структурные особенности, а это означает, что их активные каталитические центры мало различаются от вида к виду или даже между ДНК-полимеразами, использующими ДНК в качестве матрицы, и ДНК-полимеразами, использующими РНК в качестве матрицы. Каталитическая область эукариотической обратной транскриптазы состоит из трех доменов, называемых «пальцы», «ладонь» и «большой палец», которые удерживают двухцепочечный праймер-шаблон в правом захвате с 3'-ОН праймера, погруженным в активный центр полимеразы[20], кластер высококонсервативных кислотных и полярных остатков, расположенный на ладони между указательным и средним пальцами. В эукариотических RT домен РНКазы H расположен на запястье ниже основания большого пальца, но в ретронных RT активность РНКазы H отсутствует. Расщелина, связывающая нуклеиновую кислоту, простирающаяся от активного сайта полимеразы до активного сайта РНКазы H, имеет длину около 60 Å в эукариотических RT, что соответствует почти двум спиральным виткам[21]. Когда эукариотическая RT удлиняет обычный праймер, растущая двойная спираль ДНК/РНК закручивается по спирали вдоль щели, и когда двойная спираль проходит через домен РНКазы H, матричная РНК расщепляется с высвобождением зарождающейся цепи кДНК. Однако в случае удлинения праймера мсДНК длинная цепь РНК остаётся присоединённой к 3'-ОН праймера G. Хотя можно смоделировать матричный комплекс ОТ-праймер, который сделал бы 2'-ОН доступным для реакция прайминга[19], дальнейшее удлинение цепи ДНК представляет собой проблему: по мере прогрессирования синтеза ДНК объёмная цепь РНК, отходящая от 3'-ОН, должна каким-то образом спирально спускаться по связывающей щели, не блокируясь стерическими препятствиями. Чтобы решить эту проблему, обратной транскриптазе мсДНК явно потребуются специальные функции, не присущие другим RT[13].
Использованная литература[править | править код]
- ↑ 1 2 "Structure of msDNA from Myxococcus xanthus: evidence for a long, self-annealing RNA precursor for the covalently linked, branched RNA". Cell. 51 (6): 1105—12. December 1987. doi:10.1016/0092-8674(87)90596-4. PMID 2446773.
- ↑ "Two independent retrons with highly diverse reverse transcriptases in Myxococcus xanthus". Proceedings of the National Academy of Sciences of the United States of America. 87 (3): 942—5. February 1990. Bibcode:1990PNAS...87..942I. doi:10.1073/pnas.87.3.942. PMID 1689062.
- ↑ "Multicopy single-stranded DNA isolated from a gram-negative bacterium, Myxococcus xanthus". Cell. 38 (1): 203—9. August 1984. doi:10.1016/0092-8674(84)90541-5. PMID 6088065.
- ↑ "Biosynthesis and structure of stable branched RNA covalently linked to the 5' end of multicopy single-stranded DNA of Stigmatella aurantiaca". Cell. 48 (1): 55—62. January 1987. doi:10.1016/0092-8674(87)90355-2. PMID 2431795.
- ↑ "Comparative Study of different msDNA (multicopy single-stranded DNA) structures and phylogenetic comparison of reverse transcriptases (RTs): evidence for vertical inheritance" (PDF). Bioinformation. 7 (4): 176—9. 2011. doi:10.6026/97320630007176. PMID 22102774. Архивировано (PDF) 27 сентября 2022. Дата обращения: 27 сентября 2022.
- ↑ "Reverse transcriptase in a clinical strain of Escherichia coli: production of branched RNA-linked msDNA" (PDF). Science. 243 (4894 Pt 1): 1033—8. February 1989. Bibcode:1989Sci...243.1033L. doi:10.1126/science.2466332. PMID 2466332. Архивировано (PDF) 22 декабря 2014. Дата обращения: 8 февраля 2012.
- ↑ "Isolation and characterization of host mutants defective in msDNA synthesis: role of ribonuclease H in msDNA synthesis". Plasmid. 33 (3): 235—8. May 1995. doi:10.1006/plas.1995.1026. PMID 7568472.
- ↑ "Retron for the 67-base multicopy single-stranded DNA from Escherichia coli: a potential transposable element encoding both reverse transcriptase and Dam methylase functions". Proceedings of the National Academy of Sciences of the United States of America. 87 (23): 9454—8. December 1990. Bibcode:1990PNAS...87.9454H. doi:10.1073/pnas.87.23.9454. PMID 1701261.
- ↑ Inouye S. Reverse transcriptase. — Cold Spring Harbor Laboratory Press, 1993. — ISBN 978-0-87969-382-4.
- ↑ 1 2 "Comparative Study of different msDNA (multicopy single-stranded DNA) structures and phylogenetic comparison of reverse transcriptases (RTs): evidence for vertical inheritance" (PDF). Bioinformation. 7 (4): 176—9. 2011. doi:10.6026/97320630007176. PMID 22102774. Архивировано (PDF) 27 сентября 2022. Дата обращения: 27 сентября 2022.
- ↑ "Retrons, msDNA, and the bacterial genome" (PDF). Cytogenetic and Genome Research. 110 (1—4): 491—9. 2005. doi:10.1159/000084982. PMID 16093702. Архивировано (PDF) 5 марта 2016. Дата обращения: 8 февраля 2012.
- ↑ "A diversity of uncharacterized reverse transcriptases in bacteria". Nucleic Acids Research. 36 (22): 7219—29. December 2008. doi:10.1093/nar/gkn867. PMID 19004871.
- ↑ 1 2 3 "Retrons, msDNA, and the bacterial genome" (PDF). Cytogenetic and Genome Research. 110 (1—4): 491—9. 2005. doi:10.1159/000084982. PMID 16093702. Архивировано (PDF) 5 марта 2016. Дата обращения: 8 февраля 2012.
- ↑ 1 2 Das R., Shimamoto T., Arifuzzaman M. A Novel msDNA (Multicopy Single-Stranded DNA) Strain Present in Yersinia frederiksenii ATCC 33641 Contig01029 Enteropathogenic Bacteria with the Genomic Analysis of It's Retron. (англ.) // Journal Of Pathogens. — 2011. — Vol. 2011. — P. 693769—693769. — doi:10.4061/2011/693769. — PMID 22567337. — PMC 3335539.
- ↑ Dhundale A., Lampson B., Furuichi T., Inouye M., Inouye S. Structure of msDNA from Myxococcus xanthus: evidence for a long, self-annealing RNA precursor for the covalently linked, branched RNA. (англ.) // Cell. — 1987. — 24 December (vol. 51, no. 6). — P. 1105—1112. — doi:10.1016/0092-8674(87)90596-4. — PMID 2446773.
- ↑ Sherman L. A., Chattopadhyay S. The molecular basis of allorecognition. (англ.) // Annual Review Of Immunology. — 1993. — Vol. 11. — P. 385—402. — doi:10.1146/annurev.iy.11.040193.002125. — PMID 8476567.
- ↑ Buss, Leo. The Evolution of Individuality. — Princeton University Press, 2006. — ISBN 978-0-691-08469-5.
- ↑ "In vitro synthesis of multicopy single-stranded DNA, using separate primer and template RNAs, by Escherichia coli reverse transcriptase". Journal of Bacteriology. 180 (11): 2999—3002. June 1998. doi:10.1128/JB.180.11.2999-3002.1998. PMID 9603895.
- ↑ 1 2 "Highly specific recognition of primer RNA structures for 2'-OH priming reaction by bacterial reverse transcriptases". The Journal of Biological Chemistry. 274 (44): 31236—44. October 1999. doi:10.1074/jbc.274.44.31236. PMID 10531319.
{{cite journal}}: Википедия:Обслуживание CS1 (не помеченный открытым DOI) (ссылка) - ↑ "Crystal structure of human immunodeficiency virus type 1 reverse transcriptase complexed with double-stranded DNA at 3.0 A resolution shows bent DNA". Proceedings of the National Academy of Sciences of the United States of America. 90 (13): 6320—4. July 1993. Bibcode:1993PNAS...90.6320J. doi:10.1073/pnas.90.13.6320. PMID 7687065.
- ↑ "Crystal structure of HIV-1 reverse transcriptase in complex with a polypurine tract RNA:DNA". The EMBO Journal. 20 (6): 1449—61. March 2001. doi:10.1093/emboj/20.6.1449. PMID 11250910.
Дальнейшее чтение[править | править код]
- The msDNAs of bacteria. — 2001. — Vol. 67. — P. 65–91. — ISBN 9780125400671. — doi:10.1016/S0079-6603(01)67025-9.
- The Dynamic Bacterial Genome. — 2005. — Vol. 8. — P. 121–148. — ISBN 978-0-511-54154-4. — doi:10.1017/CBO9780511541544.004.