Z-ДНК
Z-ДНК — одна из многих возможных структур двойной спирали ДНК, представляет собой левозакрученную двойную спираль (в отличие от правозакрученной, как наиболее распространённая форма В-ДНК). Z-ДНК является одной из трёх биологически активных двойных спиральных структур ДНК, наряду с А-ДНК и В-ДНК, хотя точные её функции к настоящему моменту не определены[1].
История изучения[править | править код]
Левозакрученная ДНК впервые была открыта Робертом Уэллсом и коллегами при изучении полимера, образованного повторениями инозин-цитозина[2]. Они наблюдали «обратный» круговой дихроизм в таких ДНК, из чего сделали верный вывод, что её цепи обвивают друг друга в направлении налево. Впоследствии была опубликована кристаллическая структура Z-ДНК, где в ходе рентгеноструктурного анализа выяснилось, что она является первым однокристаллическим фрагментом ДНК (самокомплементарный гексамер ДНК d(CG)3). Было установлено, что Z-ДНК представляет собой левозакрученную двойную спираль ДНК из двух антипараллельных цепей, соединённых связями между парами азотистых оснований. Эти работы были проведены Эндрю Уонг (англ. Andrew Wang), Александром Ричем и их сотрудниками в Массачусетском технологическом институте[3].
В 1970 году было показано, что наиболее распространённая B-форма ДНК может переходить в Z-форму. В этом эксперименте было продемонстрировано, что круговой дихроизм полимера (dG-dC) в ультрафиолетовых лучах при в растворе 4М NaCl менялся на строго противоположный[4]. То, что при этом переходе В-форма перешла в Z-форму, было подтверждено результатами рамановской спектроскопии[5]. Кристаллизация соединения В- и Z-ДНК, проведённая в 2005 году[6], дала лучшее понимание потенциальной роли, которую Z-ДНК играет в клетке. Везде, где есть сегменты форм Z-ДНК, должны быть также В-Z-соединения на их концах, связывающие Z-форму с B-формой, встречающейся во всём остальном геноме.
В 2007 году была описана РНК-версия Z-ДНК как трансформированная форма двойной правозакрученной спирали A-РНК в левозакрученную спираль[7]. Переход от А-РНК в Z-РНК, тем не менее, был описан уже в 1984 году[8].
Структура[править | править код]
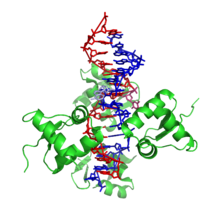
Z-ДНК значительно отличается от правозакрученных форм. Z-ДНК — левозакрученная и имеет первичную структуру, повторяющуюся через каждые 2 пары оснований. На один поворот спирали приходится 12 пар оснований. В отличие от А- и В-ДНК, в Z-ДНК большая бороздка слабо различима, малая бороздка узкая и глубокая[9]. Вообще, структура Z-ДНК энергетически невыгодна, хотя некоторые условия могут активизировать её формирования, как то: чередующиеся пуриново-пиримидиновые последовательности (особенно поли(dGC)2), негативная сверхспирализация ДНК, высокое содержание солей и некоторые катионы (все при физиологической температуре — 37 °C и pH 7,3—7,4). Z-ДНК может соединяться с B-ДНК в структуру, приводящую к вытеснению пар оснований (см. рис.)[10].

Ещё одной особенностью Z-ДНК является чередование конформаций нуклеотидных остатков. Дезоксицитидин находится в стандартной конформации: сахар в С2'-эндоконформации (см. рис.), а основание — в анти-конформации (то есть основание повёрнуто в сторону, противоположную гидроксильной группе при пятом атоме углерода; в таком положении находятся основания в полинуклеотидной цепи[11]). У дезоксигуанозина сахар находится в С3'-эндоконформации, а основание имеет крайне нетипичную син-конформацию[12].
Стэкинг оснований в Z-ДНК обладает новыми, присущими лишь этой форме свойствами. Так, стэкинговые взаимодействия имеются только между остатками цитозина противоположных цепей, а остатки гуанина вообще не взаимодействуют друг с другом[1].
Фосфаты в Z-ДНК не эквивалентны друг другу и удалены на различные расстояния от оси спирали; для гуаниновых нуклеотидов это расстояние равно 0,62 нм, а для цитозиновых — 0,76 нм. При этом соседние сахара «смотрят» в противоположные стороны, и из-за этого линия, последовательно соединяющая атомы фосфора в цепи, становится зигзагообразной (отсюда название — Z-ДНК)[1].
Структура Z-ДНК сложна для изучения, потому что она практически не существует в стабильной форме двойной спирали. Напротив, левозакрученная спираль Z-ДНК является временной структурой, появляющейся в результате биологической активности и быстро исчезающей[13].

Переход из В-ДНК в Z-ДНК[править | править код]
Как уже говорилось, В- и Z-формы способны переходить друг в друга. Это происходит при изменении ионной силы раствора или концентрации катионов, нейтрализующих отрицательный заряд фосфодиэфирного каркаса. При этом для перехода нет необходимости для расхождения цепей, он инициируется разрывом водородных связей у нескольких пар оснований, после чего гуанин фиксируется в син-конформации, водородные связи восстанавливаются, и основания вновь образуют уотсон-криковские пары. Область перехода движется по спирали в виде петли[1].
Предсказание структуры Z-ДНК[править | править код]
В настоящий момент возможно предсказать правдоподобную последовательность ДНК, находящейся в форме Z-ДНК. Алгоритм для предсказания склонности ДНК перестраиваться из В-формы в Z-форму, ZHunt, был написан в 1984 году д-ром P. Shing Ho из Массачусеткого технологического института[14]. Позже этот алгоритм был развит Трейси Кэмп и коллегами для определения мест образования Z-ДНК во всём геноме[15].
Алгоритм ZHunt доступен по ссылке Z-Hunt online.
Биологическое значение[править | править код]
Z-ДНК обнаружены у представителей всех трёх доменов жизни: архей (в частности, у галоархей[16]), бактерий и эукариот[9]. Пока чётких биологических функций Z-ДНК не определено, однако предположительно она участвует в регуляции экспрессии генов на уровне транскрипции. Действительно, достоверно известно, что с регуляцией экспрессии генов у эукариот связана последовательность dm5-dG, которая в физиологических условиях находится в форме Z-ДНК. Эта регуляция может быть опосредована сверхспирализацией, связыванием с белками, специфическим к Z-ДНК, определёнными катионами типа спермидина и метилированием дезоксицитидина[17].
Предположение о том, что Z-ДНК обеспечивает сверхспирализацию ДНК во время транскрипции[6][18], подтверждается тем, что потенциал к образованию Z-форм обнаруживается на участках, задействованных в активной транскрипции. Была показана связь мест образования Z-ДНК в генах 22-й хромосомы человека и известных для них сайтов начала транскрипции[15].
Z-ДНК образуется после начала транскрипции. Первый домен, связывающийся с Z-ДНК и имеющий к ней большое сродство, был обнаружен у фермента ADAR1 (РНК-специфической аденозиндеаминазы)[19][20] (этот домен получил название Z-альфа домена). Кристаллографические исследования и исследования, проведённые методом ядерного магнитного резонанса, подтвердили, что этот домен связывает Z-ДНК вне зависимости от её последовательности нуклеотидов[21][22][23]. Схожие участки были обнаружены в некоторых других белках, гомологичных ADAR1[20]. Идентификация Z-альфа домена легла в основу характеризации Z-РНК и соединения B- с Z-ДНК. Исследования показали, что домен ADAR1, связывающий Z-ДНК, позволяет этому ферменту локализоваться в местах активной транскрипции, где он и выполняет свою функцию — изменяет последовательность новообразованной РНК[24][25].
В 2003 году биофизик Александр Рич из Массачусетского технологического института заметил, что фактор вирулентности поксвируса, называемый E3L, имеет Z-альфа-родственный участок, схожий с белком млекопитающих, связывающим Z-ДНК[26][27]. В 2005 году Рич и коллеги выяснили, какое значение E3L имеет для поксвируса. При экспрессии генов E3L вызывает повышение транскрипции нескольких генов хозяйской клетки от 5 до 10 раз, причём эти гены блокируют способность клеток к саморазрушению (апоптозу) как к защитной реакции против инфекции.
Рич предположил, что Z-ДНК необходима для транскрипции и E3L стабилизирует Z-ДНК, таким образом увеличивая экспрессию антиапоптических генов. Он также выдвинул идею, что малые молекулы могут связываться с E3L, препятствуя соединению этого белка с Z-ДНК, и в итоге мешают экспрессии антиапоптозных генов. Потенциально это может быть использовано в основе метода защиты от оспы, вызываемой поксвирусами.
С помощью антител к Z-ДНК эта форма ДНК была обнаружена в междисковых областях политенных хромосом. Дело в том, что нуклеосомы имеются только у В-ДНК, а переход в Z-форму разрушает структуру нуклеосомы и, следовательно, состоящего из нуклеосом хроматина. В связи с этим предполагается, что Z-форма может выполнять какую-то регуляторную роль, тем более, переход В → Z обратим[1].
Установлено, что токсический эффект бромистого этидия на трипаносомы связан с переходом их кинетопластной ДНК в Z-форму. Этот эффект обусловлен интеркаляцией EtBr в ДНК, из-за чего ДНК теряет свою нативную структуру, расплетается, переходит в Z-форму и из-за этого становится неспособной к репликации[28].
Сравнение геометрических параметров некоторых форм ДНК[править | править код]
| Геометрический параметр | A-форма | B-форма | Z-форма |
|---|---|---|---|
| Направление | правозакрученная | правозакрученная | левозакрученная |
| Единица повтора | 1 пара оснований (п. о.) | 1 п. о. | 2 п. о. |
| Оборот (в градусах) | 32,7° | 35,9° | 60°/2 |
| Изгиб | 11 п. о. | 10,5 п. о. | 12 п. о. |
| Расположение п.о. относительно оси |
+19° | −1.2° | −9° |
| Подъём вдоль оси | 2,3 Å (0,23 нм) | 3,32 Å (0,332 нм) | 3,8 Å (0,38 нм) |
| Наклон | 28,2 Å (2,82 нм) | 33,2 Å (3,32 нм) | 45,6 Å (4,56 нм) |
| Скрученность | +18° | +16° | 0° |
| Конформация основания | анти- | анти- | C: анти-, G: син- |
| Конформация сахара | C3'-эндо | C2'-эндо | C: C2'-эндо, G: C3'-эндо |
| Диаметр | 23 Å (2,3 нм) | 20 Å (2,0 нм) | 18 Å (1,8 нм) |
| Источники:[29][30][31] | |||

Примечания[править | править код]
- ↑ 1 2 3 4 5 Коничев, Севастьянова, 2012, с. 93.
- ↑ Mitsui et al. Physical and enzymatic studies on poly d(I-C)-poly d(I-C), an unusual double-helical DNA (англ.) // Nature (London) : journal. — 1970. — Vol. 228, no. 5277. — P. 1166—1169. — PMID 4321098.
- ↑ Wang AHJ, Quigley G. J., Kolpak F. J., Crawford J. L., van Boom J. H., Van der Marel G., Rich A. Molecular structure of a left-handed double helical DNA fragment at atomic resolution (англ.) // Nature (London) : journal. — 1979. — Vol. 282, no. 5740. — P. 680—686. — doi:10.1038/282680a0. — . — PMID 514347.
- ↑ Pohl F. M., Jovin T. M. Salt-induced co-operative conformational change of a synthetic DNA: equilibrium and kinetic studies with poly(dG-dC) (англ.) // J. Mol. Biol. : journal. — 1972. — Vol. 67. — P. 375—396. — doi:10.1016/0022-2836(72)90457-3. — PMID 5045303.
- ↑ Thamann T. J., Lord R. C., Wang AHJ, Rich A. High salt form of poly(dG-dC)•poly(dG-dC) is left handed Z-DNA: raman spectra of crystals and solutions (англ.) // Nucl. Acids Res. : journal. — 1981. — Vol. 9. — P. 5443—5457. — doi:10.1093/nar/9.20.5443. — PMID 7301594.
- ↑ 1 2 Ha S. C., Lowenhaupt K., Rich A., Kim Y. G., Kim K. K. Crystal structure of a junction between B-DNA and Z-DNA reveals two extruded bases (англ.) // Nature : journal. — 2005. — Vol. 437, no. 7062. — P. 1183—1186. — doi:10.1038/nature04088. — . — PMID 16237447.
- ↑ Placido D., Brown BA 2nd, Lowenhaupt K., Rich A., Athanasiadis A. A left-handed RNA double helix bound by the Zalpha domain of the RNA-editing enzyme ADAR1 (англ.) // Structure : journal. — 2007. — Vol. 15, no. 4. — P. 395—404. — doi:10.1016/j.str.2007.03.001. — PMID 17437712. — PMC 2082211.
- ↑ Hall K., Cruz P., Tinoco I Jr, Jovin T. M., van de Sande J. H. 'Z-RNA'--a left-handed RNA double helix (англ.) // Nature. — 1984. — October (vol. 311, no. 5986). — P. 584—586. — doi:10.1038/311584a0. — . — PMID 6482970.
- ↑ 1 2 Nelson, Cox, 2008, p. 281.
- ↑ de Rosa M., de Sanctis D., Rosario A. L., Archer M., Rich A., Athanasiadis A., Carrondo M. A. Crystal structure of a junction between two Z-DNA helices (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2010. — 18 May (vol. 107, no. 20). — P. 9088—9092. — doi:10.1073/pnas.1003182107. — . — PMID 20439751. — PMC 2889044.
- ↑ Коничев, Севастьянова, 2012, с. 82.
- ↑ Коничев, Севастьянова, 2012, с. 92.
- ↑ Zhang H., Yu H., Ren J., Qu X. Reversible B/Z-DNA transition under the low salt condition and non-B-form polydApolydT selectivity by a cubane-like europium-L-aspartic acid complex (англ.) // Biophysical Journal : journal. — 2006. — Vol. 90, no. 9. — P. 3203—3207. — doi:10.1529/biophysj.105.078402. — . — PMID 16473901. — PMC 1432110. Архивировано 12 октября 2008 года.
- ↑ Ho P. S., Ellison M. J., Quigley G. J., Rich A. A computer aided thermodynamic approach for predicting the formation of Z-DNA in naturally occurring sequences (англ.) // EMBO Journal : journal. — 1986. — Vol. 5, no. 10. — P. 2737—2744. — PMID 3780676. — PMC 1167176.
- ↑ 1 2 Champ P. C., Maurice S., Vargason J. M., Camp T., Ho P. S. Distributions of Z-DNA and nuclear factor I in human chromosome 22: a model for coupled transcriptional regulation (англ.) // Nucleic Acids Res. : journal. — 2004. — Vol. 32, no. 22. — P. 6501—6510. — doi:10.1093/nar/gkh988. — PMID 15598822. — PMC 545456. Архивировано 25 сентября 2019 года.
- ↑ Paul Blum. Archaea: Ancient Microbes, Extreme Environments, and the Origin of Life. — Academic Press, 2001. — Vol. 50. — P. 206. — (Advances in Applied Microbiology).
- ↑ Коничев, Севастьянова, 2012, с. 93—94.
- ↑ Rich A., Zhang S. Timeline: Z-DNA: the long road to biological function (англ.) // Nature Review Genetics : journal. — 2003. — Vol. 4, no. 7. — P. 566—572. — doi:10.1038/nrg1115. — PMID 12838348.
- ↑ Herbert A., Rich A. A method to identify and characterize Z-DNA binding proteins using a linear oligodeoxynucleotide (англ.) // Nucleic Acids Res : journal. — 1993. — Vol. 21, no. 11. — P. 2669—2672. — doi:10.1093/nar/21.11.2669. — PMID 8332463. — PMC 309597.
- ↑ 1 2 Herbert A., Alfken J., Kim Y. G., Mian I. S., Nishikura K., Rich A. A Z-DNA binding domain present in the human editing enzyme, double-stranded RNA adenosine deaminase (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1997. — Vol. 94, no. 16. — P. 8421—8426. — doi:10.1073/pnas.94.16.8421. — . — PMID 9237992. — PMC 22942.
- ↑ Herbert A., Schade M., Lowenhaupt K., Alfken J., Schwartz T., Shlyakhtenko L. S., Lyubchenko Y. L., Rich A. The Zalpha domain from human ADAR1 binds to the Z-DNA conformer of many different sequences (англ.) // Nucleic Acids Res : journal. — 1998. — Vol. 26, no. 15. — P. 2669—2672. — doi:10.1093/nar/26.15.3486. — PMID 9671809. — PMC 147729.
- ↑ Schwartz T., Rould M. A., Lowenhaupt K., Herbert A., Rich A. Crystal structure of the Zalpha domain of the human editing enzyme ADAR1 bound to left-handed Z-DNA (англ.) // Science : journal. — 1999. — Vol. 284, no. 5421. — P. 1841—1845. — doi:10.1126/science.284.5421.1841. — PMID 10364558.
- ↑ Schade M., Turner C. J., Kühne R., Schmieder P., Lowenhaupt K., Herbert A., Rich A., Oschkinat H. The solution structure of the Zalpha domain of the human RNA editing enzyme ADAR1 reveals a prepositioned binding surface for Z-DNA (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1999. — Vol. 96, no. 22. — P. 2465—2470. — doi:10.1073/pnas.96.22.12465. — . — PMID 10535945. — PMC 22950.
- ↑ Herbert A., Rich A. The role of binding domains for dsRNA and Z-DNA in the in vivo editing of minimal substrates by ADAR1 (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2001. — Vol. 98, no. 21. — P. 12132—12137. — doi:10.1073/pnas.211419898. — . — PMID 11593027.
- ↑ Halber D. Scientists observe biological activities of 'left-handed' DNA. MIT News Office (11 сентября 1999). Дата обращения: 29 сентября 2008. Архивировано 16 февраля 2013 года.
- ↑ Kim Y. G., Muralinath M., Brandt T., Pearcy M., Hauns K., Lowenhaupt K., Jacobs B. L., Rich A. A role for Z-DNA binding in vaccinia virus pathogenesis (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2003. — Vol. 100, no. 12. — P. 6974—6979. — doi:10.1073/pnas.0431131100. — . — PMID 12777633. — PMC 165815.
- ↑ Kim Y. G., Lowenhaupt K., Oh D. B., Kim K. K., Rich A. Evidence that vaccinia virulence factor E3L binds to Z-DNA in vivo: Implications for development of a therapy for poxvirus infection (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2004. — Vol. 101, no. 6. — P. 1514—1518. — doi:10.1073/pnas.0308260100. — . — PMID 14757814. — PMC 341766.
- ↑ Roy Chowdhury, Arnab; Bakshi, Rahul; Wang, Jianyang; Yildirir, Gokben; Liu, Beiyu; Pappas-Brown, Valeria; Tolun, Gökhan; Griffith, Jack D.; Shapiro, Theresa A.; Jensen, Robert E.; Englund, Paul T.; Ullu, Elisabetta. The Killing of African Trypanosomes by Ethidium Bromide (англ.) // PLoS Pathogens : journal. — 2010. — 16 December (vol. 6, no. 12). — P. e1001226. — doi:10.1371/journal.ppat.1001226.
- ↑ Sinden, Richard R. DNA structure and function (неопр.). — 1st. — Academic Press, 1994. — С. 398. — ISBN 0-126-45750-6.
- ↑ Rich A., Norheim A., Wang AHJ. The chemistry and biology of left-handed Z-DNA (англ.) // Annual Review of Biochemistry : journal. — 1984. — Vol. 53, no. 1. — P. 791—846. — doi:10.1146/annurev.bi.53.070184.004043. — PMID 6383204.
- ↑ Ho P. S. The non-B-DNA structure of d(CA/TG)n does not differ from that of Z-DNA (англ.) // Proceedings of the National Academy of Sciences of the United States of America : journal. — 1994. — 27 September (vol. 91, no. 20). — P. 9549—9553. — doi:10.1073/pnas.91.20.9549. — . — PMID 7937803. — PMC 44850.
Литература[править | править код]
- Коничев А. С., Севастьянова Г. А. Молекулярная биология. — Издательский центр «Академия», 2012. — 400 с. — ISBN 978-5-7695-9147-1.
- David L. Nelson, Michael M. Cox. Lehninger Principles of biochemistry. — Fifth edition. — New York: W. H. Freeman and company, 2008. — 1158 p. — ISBN 978-0-7167-7108-1.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |