Генетический код: различия между версиями
| [отпатрулированная версия] | [отпатрулированная версия] |
Спасено источников — 1, отмечено мёртвыми — 0. Сообщить об ошибке. См. FAQ. Внимание: согласно обсуждению бот больше не будет оставлять сообщений на СО. #IABot (v2.0beta9) |
Minina (обсуждение | вклад) |
||
| Строка 28: | Строка 28: | ||
Трансляция начинается с так называемого старт-кодона — как правило, AUG, причём у [[Эукариоты|эукариот]] он читается как [[метионин]], а у бактерий — как [[N-Формилметионин|формилметионин]]. Одного старт-кодона недостаточно для запуска трансляции; для него необходимы [[факторы инициации трансляции]], а также особые элементы в соседних последовательностях, например, [[последовательность Шайна — Дальгарно]] у бактерий. У некоторых организмов в роли старт-кодонов используются кодоны GUG, который в норме кодирует [[валин]], и UUG, который в стандартном коде соответствует [[лейцин]]у<ref name="pmid12867081">{{cite pmid|12867081}}</ref>. |
Трансляция начинается с так называемого старт-кодона — как правило, AUG, причём у [[Эукариоты|эукариот]] он читается как [[метионин]], а у бактерий — как [[N-Формилметионин|формилметионин]]. Одного старт-кодона недостаточно для запуска трансляции; для него необходимы [[факторы инициации трансляции]], а также особые элементы в соседних последовательностях, например, [[последовательность Шайна — Дальгарно]] у бактерий. У некоторых организмов в роли старт-кодонов используются кодоны GUG, который в норме кодирует [[валин]], и UUG, который в стандартном коде соответствует [[лейцин]]у<ref name="pmid12867081">{{cite pmid|12867081}}</ref>. |
||
Существует три стоп-кодона, каждый из которых имеет своё название: UAG (янтарь), UGA (опал) и UAA (охра). Стоп-кодоны также называют терминаторными. В клетках нет тРНК, соответствующих стоп-кодонам, поэтому, когда рибосома доходит до стоп-кодона, вместо тРНК с ним |
Существует три стоп-кодона, каждый из которых имеет своё название: UAG (янтарь), UGA (опал) и UAA (охра). Стоп-кодоны также называют терминаторными. В клетках нет тРНК, соответствующих стоп-кодонам, поэтому, когда рибосома доходит до стоп-кодона, вместо тРНК с ним взаимодействуют факторы терминации трансляции, которые заставляют рибосому [[Диссоциация (химия)|диссоциировать]]<ref name="urlHow nonsense mutations got their names">{{cite web | url = http://www.sci.sdsu.edu/~smaloy/MicrobialGenetics/topics/rev-sup/amber-name.html | title = How nonsense mutations got their names | author = Maloy S | date = 2003-11-29 | work = Microbial Genetics Course | publisher = San Diego State University | accessdate = 2010-03-10 }}</ref>. |
||
Распределение стоп-кодонов в геноме организма неслучайно и может быть связано с [[GC-состав]]ом генома<ref>{{Cite pmid|22974057}}</ref><ref>{{cite pmid|25217634}}</ref>. Например, у штамма ''E. coli'' K-12 в геноме имеется 2705 кодонов TAA (63 %), 1257 TGA (29 %) и 326 TAG (8 %) при GC-составе 50,8 %<ref>{{cite web |title=Escherichia coli str. K-12 substr. MG1655, complete genome [Genbank Accession Number: U00096] |publisher=NCBI |work=GenBank |url=https://www.ncbi.nlm.nih.gov/nuccore/U00096 |accessdate=2013-01-27}}</ref>. Масштабное исследование геномов разных видов бактерий показало, что доля кодона TAA положительно коррелирует с GC-составом, а доля TGA — положительно. Частота самого редко используемого стоп-кодона, TAG, не связана с GC-составом<ref>{{cite pmid|18708500}}</ref>. |
|||
Иногда стоп-кодоны выступают в роли смысловых. Например, кодон UGA кодирует нестандартную аминокислоту [[селеноцистеин]], если рядом с ним в транскрипте находится так называемый [[SECIS-элемент]]<ref>{{cite pmid|17508906}}</ref>. Схожим образом стоп-кодон UAG может кодировать другую нестандартную аминокислоту — [[пирролизин]]. Иногда стоп-кодон распознаётся как смысловой при мутациях, затрагивающих тРНК. Наиболее часто это явление наблюдается у [[Вирусы|вирусов]], но оно также описано у бактерий, [[Дрожжи|дрожжей]], дрозофилы и человека, у которых играет регуляторную роль<ref name="pmid14759362">{{cite pmid|14759362}}</ref><ref name="pmid25247702">{{cite pmid|25247702}}</ref>. |
|||
=== Генетический код и мутации === |
=== Генетический код и мутации === |
||
Версия от 17:19, 23 сентября 2018
Схема генетического кода
Генети́ческий код (англ. Genetic code) — совокупность правил, согласно которым в живых клетках информация переводится с языка нуклеотидов (ген и мРНК) в язык аминокислот (белок). Собственно перевод (трансляцию) осуществляет рибосома, которая соединяет аминокислоты в цепочку согласно инструкции, записанной в кодонах мРНК. Соответствующие аминокислоты доставляются в рибосому молекулами тРНК. У всех живых организмов генетический код практически не отличается. Простейшим представлением генетического кода может служить таблица из 64 ячеек, в которой каждая ячейка соответствует одному из 64 возможных кодонов[1].
Правила генетического кода определяют, какой аминокислоте соответствует триплет (три подряд идущих нуклеотида) в мРНК. За редкими исключениями, каждому кодону соответствует только одна аминокислота[2]. Вариант генетического кода, который используется подавляющим большинством живых организмов, называют стандартным, или каноническим, генетическим кодом. Однако известно несколько десятков исключений из стандартного генетического кода, например, при трансляции в митохондриях человека используются несколько изменённые правила генетического кода.
История изучения

Попытки понять, каким образом последовательность ДНК кодирует аминокислотную последовательность белков, начали предприниматься почти сразу же после того, как в 1953 году была установлена структура ДНК (двойная спираль?!). Георгий Гамов предположил, что кодоны должны состоять из трёх нуклеотидов, чтобы кодонов хватило для всех 20 аминокислот (всего же возможно 64 различных кодона из трёх нуклеотидов: на каждую из трёх позиций можно поставить один из четырёх нуклеотидов)[3]. В 1961 году триплетность генетического кода удалось подтвердить экспериментально. В том же году Маршалл Ниренберг и его коллега Heinrich Matthaei использовали бесклеточную систему для трансляции in vitro. В качестве матрицы был взят олигонуклеотид, состоящий из остатков урацила (UUUU...). Пептид, синтезированный с него, содержал только аминокислоту фенилаланин[4]. Так впервые было установлено значение кодона: кодон UUU кодирует фенилаланин. Дальнейшие правила соответствия между кодонами и аминокислотами были установлены в лаборатории Северо Очоа. Было показано, что полиадениновая РНК (ААА...) транслируется в полилизиновый пептид[5], а на матрице полицитозиновой РНК (ССС...) синтезируется пептид, состоящий только из остатков пролина[6]. Значение остальных кодонов было установлено при помощи разнообразных сополимеров в ходе экспериментов, проведённых в лаборатории Хара Гобинда Кораны. Вскоре после этого Роберт Холли установил структуру молекулы тРНК, которая служит посредником при трансляции. В 1968 году Ниренберг, Корана и Холли были удостоены Нобелевской премии по физиологии и медицине[7].
После установления правил генетического кода многие учёные занялись его искусственными преобразованиями[англ.]. Так, начиная с 2001 года в генетический код были внедрены 40 аминокислот, которые в природе не входят в состав белков. Для каждой аминокислоты создавался свой кодон и соответствующая аминоацил-тРНК-синтетаза. Искусственное расширение генетического кода и создание белков с новыми аминокислотами могут помочь глубже изучить структуру белковых молекул, а также получить искусственные белки с заданными свойствами[8][9]. Х. Мураками и М. Сисидо смогли превратить некоторые кодоны из трёхнуклеотидных в четырёх- и пятинуклеотидные. Стивен Бреннер получил 65-й кодон, который был функционален in vivo[10]. В 2015 году у бактерии Escherichia coli удалось изменить значение всех кодонов UGG с триптофана на тиенопиррол-аланин, не встречающийся в природе[11]. В 2016 году был получен первый полусинтетический организм — бактерия, геном которой содержал два искусственных азотистых основания (X и Y), сохраняющихся при делении[12][13]. В 2017 году исследователи из Южной Кореи заявили о создании мыши с расширенным генетическим кодом, способной синтезировать белки с аминокислотами, не встречающимися в природе[14].
Свойства
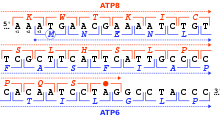
Рамка считывания
Рамка считывания определяется самым первым триплетом, с которого начинается трансляция. Последовательность неперекрывающихся кодонов, начинающуюся со старт-кодона и заканчивающуюся стоп-кодоном, называют открытой рамкой считывания. Например, последовательность 5'-AAATGAACG-3' (см. рис.) при чтении с первого нуклеотида разбивается на кодоны AAA, TGA и ACG. Если чтение начинается со второго кодона, то ей соответствуют кодоны AAT и GAA. Наконец, при чтении с третьего нуклеотида используются кодоны ATG и AAC. Таким образом, любую последовательность можно прочесть в направлении 5' → 3' тремя разными способами (с тремя разными рамками считывания), причём в каждом случае последовательность белкового продукта будет отличаться из-за распознавания рибосомой разных кодонов. Если учесть, что ДНК имеет двуцепочечную структуру, то возможны 6 рамок считывания: три на одной цепи и три на другой[15].
Старт- и стоп-кодоны
Трансляция начинается с так называемого старт-кодона — как правило, AUG, причём у эукариот он читается как метионин, а у бактерий — как формилметионин. Одного старт-кодона недостаточно для запуска трансляции; для него необходимы факторы инициации трансляции, а также особые элементы в соседних последовательностях, например, последовательность Шайна — Дальгарно у бактерий. У некоторых организмов в роли старт-кодонов используются кодоны GUG, который в норме кодирует валин, и UUG, который в стандартном коде соответствует лейцину[16].
Существует три стоп-кодона, каждый из которых имеет своё название: UAG (янтарь), UGA (опал) и UAA (охра). Стоп-кодоны также называют терминаторными. В клетках нет тРНК, соответствующих стоп-кодонам, поэтому, когда рибосома доходит до стоп-кодона, вместо тРНК с ним взаимодействуют факторы терминации трансляции, которые заставляют рибосому диссоциировать[17].
Распределение стоп-кодонов в геноме организма неслучайно и может быть связано с GC-составом генома[18][19]. Например, у штамма E. coli K-12 в геноме имеется 2705 кодонов TAA (63 %), 1257 TGA (29 %) и 326 TAG (8 %) при GC-составе 50,8 %[20]. Масштабное исследование геномов разных видов бактерий показало, что доля кодона TAA положительно коррелирует с GC-составом, а доля TGA — положительно. Частота самого редко используемого стоп-кодона, TAG, не связана с GC-составом[21].
Иногда стоп-кодоны выступают в роли смысловых. Например, кодон UGA кодирует нестандартную аминокислоту селеноцистеин, если рядом с ним в транскрипте находится так называемый SECIS-элемент[22]. Схожим образом стоп-кодон UAG может кодировать другую нестандартную аминокислоту — пирролизин. Иногда стоп-кодон распознаётся как смысловой при мутациях, затрагивающих тРНК. Наиболее часто это явление наблюдается у вирусов, но оно также описано у бактерий, дрожжей, дрозофилы и человека, у которых играет регуляторную роль[23][24].
Генетический код и мутации
В ходе репликации ДНК изредка возникают ошибки при синтезе дочерней цепи. Эти ошибки, называемые мутациями, могут повлиять на фенотип организма, особенно если они затрагивают кодирующую область?! гена. Ошибки происходят с частотой 1 на каждые 10—100 миллионов пар оснований (п. о.), так как ДНК-полимеразы могут эффективно исправлять свои ошибки[25][26].
Примерами точечных мутаций, т. е. единичных замен азотистых оснований, являются миссенс- и нонсенс-мутации. Они могут вызывать такие заболевания, как серповидноклеточная анемия и талассемия соответственно[27][28]. Клинически значимые миссенс-мутации приводят к замене аминокислотного остатка на остаток с совершенно другими физико-химическими свойствами, а нонсенс-мутации заключаются в появлении преждевременного стоп-кодона[15].
Мутации, при которых нарушается правильная рамка считывания из-за вставок и делеций (в совокупности они называются инделами[англ.]), содержащих не кратное 3 число нуклеотидов, называются мутациями сдвига рамки считывания. При этих мутациях белковый продукт получается совершенно иной, чем в диком типе. Как правило, при сдвигах рамки считывания появляются преждевременные стоп-кодоны, которые вызывают образование усечённых белков[29]. Поскольку эти мутации значительно нарушают функцию белка, они довольно редко закрепляются отбором: нередко отсутствие белка приводит к гибели организма ещё до рождения[30]. Мутации сдвига рамки считывания связаны с такими заболеваниями, как болезнь Тея — Сакса[31].
Хотя подавляющее число мутаций вредны или нейтральны[англ.], некоторые оказываются полезными[32]. Они могут давать организму лучшую приспособленность по сравнению с диким типом к определённым условиям окружающей среды или дают ему возможность размножаться быстрее особей дикого типа. В этом случае мутация будет постепенно распространяться в популяции в ходе нейтрального отбора[33]. Вирусы, геномы которых представлены РНК, мутируют очень быстро[34], что нередко приносит им пользу, потому что иммунная система, эффективно распознающая одни варианты вирусных антигены, оказывается бессильна против слегка изменённых[35]. В больших популяциях организмов, размножающихся бесполым путём, например, E. coli, одновременно может происходить несколько полезных мутаций. Этот феномен получил название клональной интерференции[англ.] и вызывает конкуренцию между мутациями[36].
Вырожденность
Впервые генетический код назвали вырожденным[англ.] Ниренберг и Бернфилд. Однако, несмотря на вырожденность, в генетическом коде полностью отсутствует двусмысленность. Например, кодоны GAA и GAG оба кодируют глутамат, но ни один из них не кодирует одновременно ещё какую-то аминокислоту. Кодоны, соответствующие одной аминокислоте, могут различаться по любым позициям, однако чаще всего две первые позиции у таких кодонов совпадают, а различается только последняя. Благодаря этому мутация, затронувшая третью позицию кодона, скорее всего, не скажется на белковом продукте. Кодоны близких по физико-химическим свойствам аминокислот также нередко похожи, благодаря чему мутации не приводят к значительным нарушениям белковой структуры. Так, кодоны NUN (N — любой нуклеотид) обычно кодируют гидрофобные аминокислоты. NCN кодируют маленькие аминокислоты с умеренной гидрофобностью, а NAN кодируют гидрофильные аминокислоты среднего размера. Генетический код устроен настолько оптимально с точки зрения гидрофобности, что математический анализ при помощи сингулярного разложения 12 переменных (4 нуклеотида на 3 позиции) даёт значимую корреляцию (0,95) для предсказания гидрофобности аминокислоты по её кодону[37]. На восемь аминокислот мутации по третьим позициям не влияют вообще, а мутации по второй позиции, как правило, приводят к замене на аминокислоту с совершенно другими физико-химическими свойствами. Однако наибольшее влияние на белковый продукт имеют мутации по первым позициям. Так, мутации, приводящие к замене заряженной аминокислоты на аминокислоту с противоположным зарядом, могут затрагивать только первую позицию, а вторую — никогда. Такая замена заряда, вероятнее всего, окажет сильный эффект на структуру белка[38].
Стандартный генетический код
В таблице ниже представлен генетический код, общий для большинства про- и эукариот. В таблице приведены все 64 кодона и указаны соответствующие аминокислоты. Порядок оснований — от 5'- к 3'-концу мРНК. Приведены трёхбуквенные и однобуквенные обозначения аминокислот.
| неполярный | полярный | основный | кислотный | (стоп-кодон) |
| 1-е основание |
2-е основание | 3-е основание | |||||||
|---|---|---|---|---|---|---|---|---|---|
| U | C | A | G | ||||||
| U | UUU | (Phe/F) Фенилаланин | UCU | (Ser/S) Серин | UAU | (Tyr/Y) Тирозин | UGU | (Cys/C) Цистеин | U |
| UUC | UCC | UAC | UGC | C | |||||
| UUA | (Leu/L) Лейцин | UCA | UAA | Стоп (охра)[B] | UGA | Стоп (опал)[B] | A | ||
| UUG[A] | UCG | UAG | Стоп (янтарь)[B] | UGG | (Trp/W) Триптофан | G | |||
| C | CUU | CCU | (Pro/P) Пролин | CAU | (His/H) Гистидин | CGU | (Arg/R) Аргинин | U | |
| CUC | CCC | CAC | CGC | C | |||||
| CUA | CCA | CAA | (Gln/Q) Глутамин | CGA | A | ||||
| CUG[A] | CCG | CAG | CGG | G | |||||
| A | AUU | (Ile/I) Изолейцин | ACU | (Thr/T) Треонин | AAU | (Asn/N) Аспарагин | AGU | (Ser/S) Серин | U |
| AUC | ACC | AAC | AGC | C | |||||
| AUA | ACA | AAA | (Lys/K) Лизин | AGA | (Arg/R) Аргинин | A | |||
| AUG[A] | (Met/M) Метионин | ACG | AAG | AGG | G | ||||
| G | GUU | (Val/V) Валин | GCU | (Ala/A) Аланин | GAU | (Asp/D) Аспарагиновая кислота | GGU | (Gly/G) Глицин | U |
| GUC | GCC | GAC | GGC | C | |||||
| GUA | GCA | GAA | (Glu/E) Глутаминовая кислота | GGA | A | ||||
| GUG | GCG | GAG | GGG | G | |||||
- A Кодон AUG кодирует метионин и одновременно является сайтом инициации трансляции: первый кодон AUG в кодирующей области мРНК служит началом синтеза белка[39]. Другие старт-кодоны (CUG, UUG и др.) редко используются в эукариотических ядерных геномах, но довольно часто — в прокариотах, митохондриях и пластидах[40].
- B ^ ^ ^ Историческая подоплёка для обозначения трёх типов стоп-кодонов как янтарь (UAG), охра (UAA) и опал/умбра (UGA) описана в статье Стоп-кодон.
| Ala/A | GCU, GCC, GCA, GCG | Leu/L | UUA, UUG, CUU, CUC, CUA, CUG |
|---|---|---|---|
| Arg/R | CGU, CGC, CGA, CGG, AGA, AGG | Lys/K | AAA, AAG |
| Asn/N | AAU, AAC | Met/M | AUG |
| Asp/D | GAU, GAC | Phe/F | UUU, UUC |
| Cys/C | UGU, UGC | Pro/P | CCU, CCC, CCA, CCG |
| Gln/Q | CAA, CAG | Ser/S | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu/E | GAA, GAG | Thr/T | ACU, ACC, ACA, ACG |
| Gly/G | GGU, GGC, GGA, GGG | Trp/W | UGG |
| His/H | CAU, CAC | Tyr/Y | UAU, UAC |
| Ile/I | AUU, AUC, AUA | Val/V | GUU, GUC, GUA, GUG |
| START | AUG | STOP | UAG, UGA, UAA |
Альтернативные генетические коды
Нестандартные аминокислоты
В некоторых белках нестандартные аминокислоты кодируются стоп-кодонами в зависимости от наличия особой сигнальной последовательности в мРНК. Например, стоп-кодон UGA может кодировать селеноцистеин, а UAG — пирролизин. Селеноцистеин и пирролизин рассматривают как 21-ю и 22-ю протеиногенную аминокислоту соответственно. В отличие от селеноцистеина, у пирролизина есть собственная аминоацил-тРНК-синтетаза[41]. Хотя обычно генетический код, используемый клетками одного организма, фиксирован, архея Acetohalobium arabaticum[англ.] может переключаться с 20-аминокислотного кода на 21-аминокислотный (включая пирролизин) при разных условиях роста?![42].
Вариации

Существование отклонений от стандартного генетического кода предсказывалось ещё в 1970-х[43]. Первое отклонение было описано в 1979 году в митохондриях человека[44]. Впоследствии было описано ещё несколько альтернативных генетических кодов, слегка отличающихся от стандартного, в том числе альтернативные митохондриальные коды[45]. Например, у бактерий рода Mycoplasma стоп-кодон UGA кодирует триптофан, а у дрожжей из так называемой «CTG-клады» (в том числе патогенного вида Candida albicans) кодон CUG кодирует серин, а не лейцин, как в стандартном генетическом коде[46][47][48]. Поскольку вирусы используют тот же генетический код, что и клетки-хозяева, отклонения от стандартного генетического кода могут нарушить размножение вирусов[49]. Впрочем, некоторые вирусы, например, вирусы рода Totivirus[англ.], используют тот же альтернативный генетический код, что и организм-хозяин[50]. У бактерий и архей GUG и UUG нередко выступают старт-кодонами[51]. Некоторые отклонения от стандартного генетического кода есть и в ядерном геноме человека: так, в 4 % мРНК фермента малатдегидрогеназы один из стоп-кодонов кодирует триптофан или аргинин[52]. Значение стоп-кодона зависит от его окружения[24]. Отклонения в генетическом коде организма можно обнаружить, если найти в его геноме очень консервативные гены и сравнить их кодоны с соответствующими аминокислотами гомологичных белков близкородственных организмов. По такому принципу работает программа FACIL, которая рассчитывает, с какой частотой каждый кодон соответствует той или иной аминокислоте, а также определяет поддержку стоп-кодона, и представляет результат в виде логотипа (LOGO)[53]. Впрочем, несмотря на все перечисленные отличия, генетические коды, используемые всеми организмами, в общих чертах схожи[54].
В таблице ниже перечислены известные на данный момент нестандартные генетические коды[55][56].
| Биохимические свойства аминокислот | неполярная | полярная | основная | кислая | Терминация: стоп-кодон |
| Код | Таблица трансляции |
ДНК-кодон | РНК-кодон | Трансляция с данным кодом |
Стандартная трансляция | Примечания | ||
|---|---|---|---|---|---|---|---|---|
| Стандартный[англ.] | 1 | Включает таблицу трансляции 8 (хлоропласты растений) | ||||||
| Митохондриальный код позвоночных | 2 | AGA | AGA | Ter (*) | Arg (R) | |||
| AGG | AGG | Ter (*) | Arg (R) | |||||
| ATA | AUA | Met (M) | Ile (I) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Митохондриальный генетический код дрожжей[англ.] | 3 | ATA | AUA | Met (M) | Ile (I) | |||
| CTT | CUU | Thr (T) | Leu (L) | |||||
| CTC | CUC | Thr (T) | Leu (L) | |||||
| CTA | CUA | Thr (T) | Leu (L) | |||||
| CTG | CUG | Thr (T) | Leu (L) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| CGA | CGA | absent | Arg (R) | |||||
| CGC | CGC | absent | Arg (R) | |||||
| Митохондриальный генетический код слизевиков, простейших, стрекающих и генетический код Mycoplasma и Spiroplasma[англ.] | 4 | TGA | UGA | Trp (W) | Ter (*) | Включает таблицу трансляции 7 (кинетопласт) | ||
| Митохондриальный код беспозвоночных[англ.] | 5 | AGA | AGA | Ser (S) | Arg (R) | |||
| AGG | AGG | Ser (S) | Arg (R) | |||||
| ATA | AUA | Met (M) | Ile (I) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Генетический код инфузорий, Dasycladacea и Hexamita[англ.] | 6 | TAA | UAA | Gln (Q) | Ter (*) | |||
| TAG | UAG | Gln (Q) | Ter (*) | |||||
| Митохондриальный генетический код иглокожих и плоских червей[англ.] | 9 | AAA | AAA | Asn (N) | Lys (K) | |||
| AGA | AGA | Ser (S) | Arg (R) | |||||
| AGG | AGG | Ser (S) | Arg (R) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Генетический код Euplotidae[англ.] | 10 | TGA | UGA | Cys (C) | Ter (*) | |||
| Генетический код бактерий, архей и пластид растений[англ.] | 11 | См. таблицу трансляции 1 | ||||||
| Альтернативный генетический код дрожжей[англ.] | 12 | CTG | CUG | Ser (S) | Leu (L) | |||
| Митохондриальный генетический код асцидий[англ.] | 13 | AGA | AGA | Gly (G) | Arg (R) | |||
| AGG | AGG | Gly (G) | Arg (R) | |||||
| ATA | AUA | Met (M) | Ile (I) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Альтернативный митохондриальный генетический код плоских червей[англ.] | 14 | AAA | AAA | Asn (N) | Lys (K) | |||
| AGA | AGA | Ser (S) | Arg (R) | |||||
| AGG | AGG | Ser (S) | Arg (R) | |||||
| TAA | UAA | Tyr (Y) | Ter (*) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Генетический код Blepharisma[англ.] | 15 | TAG | UAG | Gln (Q) | Ter (*) | |||
| Митохондриальный генетический код хлорофициевых[англ.] | 16 | TAG | UAG | Leu (L) | Ter (*) | |||
| Митохондриальный генетический код трематод[англ.] | 21 | TGA | UGA | Trp (W) | Ter (*) | |||
| ATA | AUA | Met (M) | Ile (I) | |||||
| AGA | AGA | Ser (S) | Arg (R) | |||||
| AGG | AGG | Ser (S) | Arg (R) | |||||
| AAA | AAA | Asn (N) | Lys (K) | |||||
| Митохондриальный генетический код Scenedesmus obliquus[англ.] | 22 | TCA | UCA | Ter (*) | Ser (S) | |||
| TAG | UAG | Leu (L) | Ter (*) | |||||
| Митохондриальный генетический код Thraustochytrium[англ.] | 23 | TTA | UUA | Ter (*) | Leu (L) | Сходна с таблицей трансляции 11. | ||
| Митохондриальный генетический код крыложаберных[англ.] | 24 | AGA | AGA | Ser (S) | Arg (R) | |||
| AGG | AGG | Lys (K) | Arg (R) | |||||
| TGA | UGA | Trp (W) | Ter (*) | |||||
| Генетический код возможных групп SR1 и Gracilibacteria[англ.] | 25 | TGA | UGA | Gly (G) | Ter (*) | |||
| Генетический код Pachysolen tannophilus[англ.] | 26 | CTG | CUG | Ala (A) | Leu (L) | |||
| Генетический код Karyorelictea[англ.] | 27 | TAA | UAA | Gln (Q) | Ter (*) | |||
| TAG | UAG | Gln (Q) | Ter (*) | |||||
| TGA | UGA | Ter (*) | или | Trp (W) | Ter (*) | |||
| Генетический код Condylostoma[англ.] | 28 | TAA | UAA | Ter (*) | или | Gln (Q) | Ter (*) | |
| TAG | UAG | Ter (*) | или | Gln (Q) | Ter (*) | |||
| TGA | UGA | Ter (*) | или | Trp (W) | Ter (*) | |||
| Генетический код Mesodinium[англ.] | 29 | TAA | UAA | Tyr (Y) | Ter (*) | |||
| TAG | UAG | Tyr (Y) | Ter (*) | |||||
| Генетический код Peritrichia[англ.] | 30 | TAA | UAA | Glu (E) | Ter (*) | |||
| TAG | UAG | Glu (E) | Ter (*) | |||||
| Генетический код Blastocrithidia[англ.] | 31 | TAA | UAA | Ter (*) | или | Gln (Q) | Ter (*) | |
| TAG | UAG | Ter (*) | или | Gln (Q) | Ter (*) | |||
| TGA | UGA | Trp (W) | Ter (*) | |||||
Происхождение
В настоящее время наиболее общепринятой гипотезой о происхождении жизни на Земле является гипотеза мира РНК. Любая модель возникновения генетического кода использует гипотезу о передаче основных функций от РНК-ферментов (рибозимов) к белковым ферментам. Как и предполагает гипотеза мира РНК, тРНК появились раньше аминоацил-тРНК-синтетаз, поэтому эти ферменты не могли оказать влияние на свойства тРНК[57].
Если бы соответствие между кодонами и аминокислотами было случайным, в природе существовало бы 1,5 × 1084 генетических кодов[58]. Это число получилось в результате расчёта количества способов, которыми можно 21 предмет (20 кодонов, кодирующих аминокислоты, и один стоп-кодон) разложить в 64 корзины, так чтобы каждый предмет был использован по крайней мере единожды[59]. Однако соответствия кодонов и аминокислот неслучайны[60]. Аминокислоты, которые имеют общий путь биосинтеза, как правило, имеют общую первую позицию кодонов. Этот факт может быть пережитком раннего, более простого генетического кода, который содержал меньше аминокислот, чем современный, и постепенно включил в свой состав все 20 аминокислот[61]. Кодоны аминокислот со схожими физико-химическими свойствами так же, как правило, похожи, что смягчает последствия точечных мутаций и нарушений трансляции[62][63].
Поскольку генетический код неслучаен, правдоподобная гипотеза о его возникновении должна объяснять такие свойства стандартного генетического кода, как отсутствие кодонов для D-аминокислот, включение всего лишь 20 аминокислот из возможных 64, ограничение синонимичных замен третьей позицией кодонов, функционирование в качестве стоп-кодонов именно кодонов UAG, UGA и UAA[64]. Существуют три основные гипотезы происхождения генетического кода. Каждая из них представлена множеством моделей, многие модели гибридны[65].
- Замороженная случайность: генетический код возник случайно и в таком виде закрепился. Возможно, древние рибозимы, подобные современным тРНК, имели разное сродство к аминокислотам, причём кодоны из разных частей одного и того же рибозима могли обладать наибольшим сродством к разным аминокислотам. После того, как появились первые функциональные пептиды, любое изменение генетического кода было бы летальным, поэтому он оказался «заморожен»[66].
- Стереохимическое сродство: генетический код определяется высоким сродством каждой аминокислоты с соответствующими ей кодонами и антикодонами. Сродство аминокислоты и антикодона означает, что предковым тРНК соответствовали те аминокислоты, с которыми они связывались с наибольшим сродством. В ходе эволюции соответствие антикодонов и аминокислот заменилось соответствием аминоацил-тРНК-синтетаз и аминокислот[67].
- Оптимальность: генетический код продолжал некоторое время изменяться после своего появления, поэтому современный код обеспечивает максимальную приспособленность и минимизирует последствия мутаций, то есть является лучшим из возможных генетических кодов[65].
Примечания
- ↑ Shu J. J. A new integrated symmetrical table for genetic codes. (англ.) // Bio Systems. — 2017. — January (vol. 151). — P. 21—26. — doi:10.1016/j.biosystems.2016.11.004. — PMID 27887904.
- ↑ Turanov A. A., Lobanov A. V., Fomenko D. E., Morrison H. G., Sogin M. L., Klobutcher L. A., Hatfield D. L., Gladyshev V. N. Genetic code supports targeted insertion of two amino acids by one codon. (англ.) // Science (New York, N.Y.). — 2009. — 9 January (vol. 323, no. 5911). — P. 259—261. — doi:10.1126/science.1164748. — PMID 19131629.
- ↑ Crick, Francis. Chapter 8: The genetic code // What Mad Pursuit: A Personal View of Scientific Discovery. — Basic Books, 10 July 1990. — P. 89–101. — ISBN 978-0-465-09138-6.
- ↑ NIRENBERG MW, MATTHAEI JH. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1961. — 15 October (vol. 47). — P. 1588—1602. — PMID 14479932.
- ↑ GARDNER RS, WAHBA AJ, BASILIO C, MILLER RS, LENGYEL P, SPEYER JF. Synthetic polynucleotides and the amino acid code. VII. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1962. — 15 December (vol. 48). — P. 2087—2094. — PMID 13946552.
- ↑ WAHBA AJ, GARDNER RS, BASILIO C, MILLER RS, SPEYER JF, LENGYEL P. Synthetic polynucleotides and the amino acid code. VIII. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1963. — 15 January (vol. 49). — P. 116—122. — PMID 13998282.
- ↑ "The Nobel Prize in Physiology or Medicine 1959" (Press release). The Royal Swedish Academy of Science. 1959. Дата обращения: 27 февраля 2010.
The Nobel Prize in Physiology or Medicine 1959 was awarded jointly to Severo Ochoa and Arthur Kornberg 'for their discovery of the mechanisms in the biological synthesis of ribonucleic acid and deoxyribonucleic acid'.
- ↑ Xie J., Schultz P. G. Adding amino acids to the genetic repertoire. (англ.) // Current Opinion In Chemical Biology. — 2005. — December (vol. 9, no. 6). — P. 548—554. — doi:10.1016/j.cbpa.2005.10.011. — PMID 16260173.
- ↑ Wang Q., Parrish A. R., Wang L. Expanding the genetic code for biological studies. (англ.) // Chemistry & Biology. — 2009. — 27 March (vol. 16, no. 3). — P. 323—336. — doi:10.1016/j.chembiol.2009.03.001. — PMID 19318213.
- ↑ Simon, Matthew. Emergent Computation: Emphasizing Bioinformatics. — Springer Science & Business Media, 7 January 2005. — P. 105–106. — ISBN 978-0-387-22046-8.
- ↑ Hoesl M. G., Oehm S., Durkin P., Darmon E., Peil L., Aerni H. R., Rappsilber J., Rinehart J., Leach D., Söll D., Budisa N. Chemical Evolution of a Bacterial Proteome. (англ.) // Angewandte Chemie (International Ed. In English). — 2015. — 17 August (vol. 54, no. 34). — P. 10030—10034. — doi:10.1002/anie.201502868. — PMID 26136259.
- ↑ First stable semisynthetic organism created | KurzweilAI. www.kurzweilai.net (3 февраля 2017). Дата обращения: 9 февраля 2017.
- ↑ Zhang Y., Lamb B. M., Feldman A. W., Zhou A. X., Lavergne T., Li L., Romesberg F. E. A semisynthetic organism engineered for the stable expansion of the genetic alphabet. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2017. — 7 February (vol. 114, no. 6). — P. 1317—1322. — doi:10.1073/pnas.1616443114. — PMID 28115716.
- ↑ Han S., Yang A., Lee S., Lee H. W., Park C. B., Park H. S. Expanding the genetic code of Mus musculus. (англ.) // Nature Communications. — 2017. — 21 February (vol. 8). — P. 14568—14568. — doi:10.1038/ncomms14568. — PMID 28220771.
- ↑ 1 2 King, Robert C. A Dictionary of Genetics / Robert C. King, Pamela Mulligan, William Stansfield. — OUP USA, 10 January 2013. — P. 608. — ISBN 978-0-19-976644-4.
- ↑ Touriol C., Bornes S., Bonnal S., Audigier S., Prats H., Prats A. C., Vagner S. Generation of protein isoform diversity by alternative initiation of translation at non-AUG codons. (англ.) // Biology Of The Cell. — 2003. — May (vol. 95, no. 3-4). — P. 169—178. — PMID 12867081.
- ↑ Maloy S. How nonsense mutations got their names. Microbial Genetics Course. San Diego State University (29 ноября 2003). Дата обращения: 10 марта 2010.
- ↑ Povolotskaya I. S., Kondrashov F. A., Ledda A., Vlasov P. K. Stop codons in bacteria are not selectively equivalent. (англ.) // Biology Direct. — 2012. — 13 September (vol. 7). — P. 30. — doi:10.1186/1745-6150-7-30. — PMID 22974057.
- ↑ Korkmaz G., Holm M., Wiens T., Sanyal S. Comprehensive analysis of stop codon usage in bacteria and its correlation with release factor abundance. (англ.) // The Journal Of Biological Chemistry. — 2014. — 31 October (vol. 289, no. 44). — P. 30334—30342. — doi:10.1074/jbc.M114.606632. — PMID 25217634.
- ↑ Escherichia coli str. K-12 substr. MG1655, complete genome [Genbank Accession Number: U00096]. GenBank. NCBI. Дата обращения: 27 января 2013.
- ↑ Wong T. Y., Fernandes S., Sankhon N., Leong P. P., Kuo J., Liu J. K. Role of premature stop codons in bacterial evolution. (англ.) // Journal Of Bacteriology. — 2008. — October (vol. 190, no. 20). — P. 6718—6725. — doi:10.1128/JB.00682-08. — PMID 18708500.
- ↑ Papp L. V., Lu J., Holmgren A., Khanna K. K. From selenium to selenoproteins: synthesis, identity, and their role in human health. (англ.) // Antioxidants & Redox Signaling. — 2007. — July (vol. 9, no. 7). — P. 775—806. — doi:10.1089/ars.2007.1528. — PMID 17508906.
- ↑ Namy O., Rousset J. P., Napthine S., Brierley I. Reprogrammed genetic decoding in cellular gene expression. (англ.) // Molecular Cell. — 2004. — 30 January (vol. 13, no. 2). — P. 157—168. — PMID 14759362.
- ↑ 1 2 Schueren F., Lingner T., George R., Hofhuis J., Dickel C., Gärtner J., Thoms S. Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals. (англ.) // ELife. — 2014. — 23 September (vol. 3). — P. e03640—03640. — doi:10.7554/eLife.03640. — PMID 25247702.
- ↑ Spontaneous mutations // An Introduction to Genetic Analysis. — 7th. — New York : W. H. Freeman, 2000. — ISBN 978-0-7167-3520-5.
- ↑ Freisinger E., Grollman A. P., Miller H., Kisker C. Lesion (in)tolerance reveals insights into DNA replication fidelity. (англ.) // The EMBO Journal. — 2004. — 7 April (vol. 23, no. 7). — P. 1494—1505. — doi:10.1038/sj.emboj.7600158. — PMID 15057282.
- ↑ Chang J. C., Kan Y. W. beta 0 thalassemia, a nonsense mutation in man. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1979. — June (vol. 76, no. 6). — P. 2886—2889. — PMID 88735.
- ↑ Boillée S., Vande Velde C., Cleveland D. W. ALS: a disease of motor neurons and their nonneuronal neighbors. (англ.) // Neuron. — 2006. — 5 October (vol. 52, no. 1). — P. 39—59. — doi:10.1016/j.neuron.2006.09.018. — PMID 17015226.
- ↑ Isbrandt D., Hopwood J. J., von Figura K., Peters C. Two novel frameshift mutations causing premature stop codons in a patient with the severe form of Maroteaux-Lamy syndrome. (англ.) // Human Mutation. — 1996. — Vol. 7, no. 4. — P. 361—363. — doi:10.1002/(SICI)1098-1004(1996)7:4<361::AID-HUMU12>3.0.CO;2-0. — PMID 8723688.
- ↑ Crow J. F. How much do we know about spontaneous human mutation rates? (англ.) // Environmental And Molecular Mutagenesis. — 1993. — Vol. 21, no. 2. — P. 122—129. — PMID 8444142.
- ↑ Lewis, Ricki. Human Genetics: Concepts and Applications. — 6th. — Boston, Mass : McGraw Hill, 2005. — P. 227–228. — ISBN 978-0-07-111156-0.
- ↑ Sawyer S. A., Parsch J., Zhang Z., Hartl D. L. Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2007. — 17 April (vol. 104, no. 16). — P. 6504—6510. — doi:10.1073/pnas.0701572104. — PMID 17409186.
- ↑ Bridges KR (2002). "Malaria and the Red Cell". Harvard. Архивировано 27 ноября 2011. Дата обращения: 2 сентября 2018.
{{cite journal}}: Неизвестный параметр|deadurl=игнорируется (|url-status=предлагается) (справка) - ↑ Drake J. W., Holland J. J. Mutation rates among RNA viruses. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1999. — 23 November (vol. 96, no. 24). — P. 13910—13913. — PMID 10570172.
- ↑ Holland J., Spindler K., Horodyski F., Grabau E., Nichol S., VandePol S. Rapid evolution of RNA genomes. (англ.) // Science (New York, N.Y.). — 1982. — 26 March (vol. 215, no. 4540). — P. 1577—1585. — PMID 7041255.
- ↑ de Visser J. A., Rozen D. E. Clonal interference and the periodic selection of new beneficial mutations in Escherichia coli. (англ.) // Genetics. — 2006. — April (vol. 172, no. 4). — P. 2093—2100. — doi:10.1534/genetics.105.052373. — PMID 16489229.
- ↑ Michel-Beyerle, Maria Elisabeth. Reaction centers of photosynthetic bacteria: Feldafing-II-Meeting. — Springer-Verlag, 1990. — ISBN 978-3-540-53420-4.
- ↑ Fricke M., Gerst R., Ibrahim B., Niepmann M., Marz M. Global importance of RNA secondary structures in protein coding sequences. (англ.) // Bioinformatics. — 2018. — 7 August. — doi:10.1093/bioinformatics/bty678. — PMID 30101307.
- ↑ Nakamoto T. Evolution and the universality of the mechanism of initiation of protein synthesis. (англ.) // Gene. — 2009. — 1 March (vol. 432, no. 1-2). — P. 1—6. — doi:10.1016/j.gene.2008.11.001. — PMID 19056476.
- ↑ Elzanowski A., Ostell J. The Genetic Codes. NCBI. Дата обращения: 3 июня 2023.
- ↑ Krzycki J. A. The direct genetic encoding of pyrrolysine. (англ.) // Current Opinion In Microbiology. — 2005. — December (vol. 8, no. 6). — P. 706—712. — doi:10.1016/j.mib.2005.10.009. — PMID 16256420.
- ↑ Prat L., Heinemann I. U., Aerni H. R., Rinehart J., O'Donoghue P., Söll D. Carbon source-dependent expansion of the genetic code in bacteria. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 2012. — 18 December (vol. 109, no. 51). — P. 21070—21075. — doi:10.1073/pnas.1218613110. — PMID 23185002.
- ↑ Crick F.H.C., Orgel L.E. Directed panspermia (англ.) // Icarus. — 1973. — July (vol. 19, no. 3). — P. 341—346. — ISSN 0019-1035. — doi:10.1016/0019-1035(73)90110-3.
- ↑ Barrell B. G., Bankier A. T., Drouin J. A different genetic code in human mitochondria. (англ.) // Nature. — 1979. — 8 November (vol. 282, no. 5735). — P. 189—194. — PMID 226894.
- ↑ Jukes T. H., Osawa S. The genetic code in mitochondria and chloroplasts. (англ.) // Experientia. — 1990. — 1 December (vol. 46, no. 11-12). — P. 1117—1126. — PMID 2253709.
- ↑ Fitzpatrick D. A., Logue M. E., Stajich J. E., Butler G. A fungal phylogeny based on 42 complete genomes derived from supertree and combined gene analysis. (англ.) // BMC Evolutionary Biology. — 2006. — 22 November (vol. 6). — P. 99—99. — doi:10.1186/1471-2148-6-99. — PMID 17121679.
- ↑ Santos M. A., Tuite M. F. The CUG codon is decoded in vivo as serine and not leucine in Candida albicans. (англ.) // Nucleic Acids Research. — 1995. — 11 May (vol. 23, no. 9). — P. 1481—1486. — PMID 7784200.
- ↑ Butler G., Rasmussen M. D., Lin M. F., Santos M. A., Sakthikumar S., Munro C. A., Rheinbay E., Grabherr M., Forche A., Reedy J. L., Agrafioti I., Arnaud M. B., Bates S., Brown A. J., Brunke S., Costanzo M. C., Fitzpatrick D. A., de Groot P. W., Harris D., Hoyer L. L., Hube B., Klis F. M., Kodira C., Lennard N., Logue M. E., Martin R., Neiman A. M., Nikolaou E., Quail M. A., Quinn J., Santos M. C., Schmitzberger F. F., Sherlock G., Shah P., Silverstein K. A., Skrzypek M. S., Soll D., Staggs R., Stansfield I., Stumpf M. P., Sudbery P. E., Srikantha T., Zeng Q., Berman J., Berriman M., Heitman J., Gow N. A., Lorenz M. C., Birren B. W., Kellis M., Cuomo C. A. Evolution of pathogenicity and sexual reproduction in eight Candida genomes. (англ.) // Nature. — 2009. — 4 June (vol. 459, no. 7247). — P. 657—662. — doi:10.1038/nature08064. — PMID 19465905.
- ↑ Witzany G. Crucial steps to life: From chemical reactions to code using agents. (англ.) // Bio Systems. — 2016. — February (vol. 140). — P. 49—57. — doi:10.1016/j.biosystems.2015.12.007. — PMID 26723230.
- ↑ Taylor D. J., Ballinger M. J., Bowman S. M., Bruenn J. A. Virus-host co-evolution under a modified nuclear genetic code. (англ.) // PeerJ. — 2013. — Vol. 1. — P. e50—50. — doi:10.7717/peerj.50. — PMID 23638388.
- ↑ The Genetic Codes. National Center for Biotechnology Information (NCBI) (7 апреля 2008). Дата обращения: 10 марта 2010.
- ↑ Hofhuis J., Schueren F., Nötzel C., Lingner T., Gärtner J., Jahn O., Thoms S. The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code. (англ.) // Open Biology. — 2016. — November (vol. 6, no. 11). — doi:10.1098/rsob.160246. — PMID 27881739.
- ↑ Dutilh B. E., Jurgelenaite R., Szklarczyk R., van Hijum S. A., Harhangi H. R., Schmid M., de Wild B., Françoijs K. J., Stunnenberg H. G., Strous M., Jetten M. S., Op den Camp H. J., Huynen M. A. FACIL: Fast and Accurate Genetic Code Inference and Logo. (англ.) // Bioinformatics. — 2011. — 15 July (vol. 27, no. 14). — P. 1929—1933. — doi:10.1093/bioinformatics/btr316. — PMID 21653513.
- ↑ Kubyshkin V., Acevedo-Rocha C. G., Budisa N. On universal coding events in protein biogenesis. (англ.) // Bio Systems. — 2018. — February (vol. 164). — P. 16—25. — doi:10.1016/j.biosystems.2017.10.004. — PMID 29030023.
- ↑ Elzanowski, Andrzej The Genetic Codes. National Center for Biotechnology Information (7 июля 2010). Дата обращения: 6 мая 2013.
- ↑ Watanabe Kimitsuna, Suzuki Tsutomu. Genetic Code and its Variants (англ.) // Encyclopedia of Life Sciences. — 2001. — 19 April. — ISBN 047001590X. — doi:10.1038/npg.els.0000810.
- ↑ Ribas de Pouplana L., Turner R. J., Steer B. A., Schimmel P. Genetic code origins: tRNAs older than their synthetases? (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1998. — 15 September (vol. 95, no. 19). — P. 11295—11300. — PMID 9736730.
- ↑ Yarus, Michael. Life from an RNA World: The Ancestor Within. — Harvard University Press, 2010. — ISBN 978-0-674-05075-4.
- ↑ Mathematica function for # possible arrangements of items in bins? - Online Technical Discussion Groups—Wolfram Community (амер. англ.). community.wolfram.com. Дата обращения: 3 февраля 2017.
- ↑ Freeland S. J., Hurst L. D. The genetic code is one in a million. (англ.) // Journal Of Molecular Evolution. — 1998. — September (vol. 47, no. 3). — P. 238—248. — PMID 9732450.
- ↑ Taylor F. J., Coates D. The code within the codons. (англ.) // Bio Systems. — 1989. — Vol. 22, no. 3. — P. 177—187. — PMID 2650752.
- ↑ Di Giulio M. The extension reached by the minimization of the polarity distances during the evolution of the genetic code. (англ.) // Journal Of Molecular Evolution. — 1989. — October (vol. 29, no. 4). — P. 288—293. — PMID 2514270.
- ↑ Wong J. T. Role of minimization of chemical distances between amino acids in the evolution of the genetic code. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1980. — February (vol. 77, no. 2). — P. 1083—1086. — PMID 6928661.
- ↑ Erives A. A model of proto-anti-codon RNA enzymes requiring L-amino acid homochirality. (англ.) // Journal Of Molecular Evolution. — 2011. — August (vol. 73, no. 1-2). — P. 10—22. — doi:10.1007/s00239-011-9453-4. — PMID 21779963.
- ↑ 1 2 Freeland S. J., Knight R. D., Landweber L. F., Hurst L. D. Early fixation of an optimal genetic code. (англ.) // Molecular Biology And Evolution. — 2000. — April (vol. 17, no. 4). — P. 511—518. — doi:10.1093/oxfordjournals.molbev.a026331. — PMID 10742043.
- ↑ Crick F. H. The origin of the genetic code. (англ.) // Journal Of Molecular Biology. — 1968. — December (vol. 38, no. 3). — P. 367—379. — PMID 4887876.
- ↑ Hopfield J. J. Origin of the genetic code: a testable hypothesis based on tRNA structure, sequence, and kinetic proofreading. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1978. — September (vol. 75, no. 9). — P. 4334—4338. — PMID 279919.
Статья является кандидатом в избранные статьи со 2 сентября 2018.
|

