Дезоксирибонуклеиновая кислота


Дезоксирибонуклеи́новая кислота́ (ДНК) — макромолекула, обеспечивающая хранение, передачу из поколения в поколение и реализацию генетической программы развития и функционирования живых организмов. Основная роль ДНК в клетках — долговременное хранение информации о структуре РНК и белков.
В клетках эукариотов (например, животных или растений) ДНК находится в ядре клетки в составе хромосом, а также в некоторых клеточных органоидах (митохондриях и пластидах). В клетках прокариотических организмов (бактерий и архей) кольцевая или линейная молекула ДНК, так называемый нуклеоид, прикреплена изнутри к клеточной мембране. У них и у низших эукариот (например, дрожжей) встречаются также небольшие автономные, преимущественно кольцевые молекулы ДНК, называемые плазмидами. Кроме того, одно- или двухцепочечные молекулы ДНК могут образовывать геном ДНК-содержащих вирусов.
С химической точки зрения ДНК — это длинная полимерная молекула, состоящая из повторяющихся блоков — нуклеотидов. Каждый нуклеотид состоит из азотистого основания, сахара (дезоксирибозы) и фосфатной группы. Связи между нуклеотидами в цепи образуются за счёт дезоксирибозы и фосфатной группы. В подавляющем большинстве случаев (кроме некоторых вирусов, содержащих одноцепочечную ДНК) макромолекула ДНК состоит из двух цепей, ориентированных азотистыми основаниями друг к другу. Эта двухцепочечная молекула спирализована. В целом структура молекулы ДНК получила название «двойной спирали».
В ДНК встречается четыре вида азотистых оснований (аденин, гуанин, тимин и цитозин). Азотистые основания одной из цепей соединены с азотистыми основаниями другой цепи водородными связями согласно принципу комплементарности: аденин соединяется только с тимином, гуанин — только с цитозином. Последовательность нуклеотидов позволяет «кодировать» информацию о различных типах РНК, наиболее важными из которых являются информационные, или матричные (мРНК), рибосомальные (рРНК) и транспортные (тРНК). Все эти типы РНК синтезируются на матрице ДНК за счёт копирования последовательности ДНК в последовательность РНК, синтезируемой в процессе транскрипции, и принимают участие в биосинтезе белков (процессе трансляции). Помимо кодирующих последовательностей, ДНК клеток содержит последовательности, выполняющие регуляторные и структурные функции. Кроме того, в геноме эукариот часто встречаются участки, принадлежащие «генетическим паразитам», например, транспозонам.
Расшифровка структуры ДНК (1953 г.) стала одним из поворотных моментов в истории биологии. За выдающийся вклад в это открытие Фрэнсису Крику, Джеймсу Уотсону, Морису Уилкинсу была присуждена Нобелевская премия по физиологии и медицине 1962 г.
История изучения


ДНК была открыта Иоганном Фридрихом Мишером в 1869 году. Из остатков клеток, содержащихся в гное, он выделил вещество, в состав которого входят азот и фосфор. Вначале новое вещество получило название нуклеин, а позже, когда Мишер определил, что это вещество обладает кислотными свойствами, вещество получило название нуклеиновая кислота[1]. Биологическая функция новооткрытого вещества была неясна, и долгое время ДНК считалась запасником фосфора в организме. Более того, даже в начале XX века многие биологи считали, что ДНК не имеет никакого отношения к передаче информации, поскольку строение молекулы, по их мнению, было слишком однообразным и не могло содержать закодированную информацию.
Постепенно было доказано, что именно ДНК, а не белки, как считалось раньше, является носителем генетической информации. Одно из первых решающих доказательств принесли эксперименты О. Эвери, Колина Мак-Леода и Маклин Мак-Карти (1944 г.) по трансформации бактерий. Им удалось показать, что за так называемую трансформацию (приобретение болезнетворных свойств безвредной культурой в результате добавления в неё мёртвых болезнетворных бактерий) отвечают выделенные из пневмококков ДНК. Эксперимент американских учёных Алфреда Херши и Марты Чейз (эксперимент Херши—Чейз, 1952 г.) с помеченными радиоактивными изотопами белками и ДНК бактериофагов показали, что в заражённую клетку передаётся только нуклеиновая кислота фага, а новое поколение фага содержит такие же белки и нуклеиновую кислоту, как исходный фаг[2].
Вплоть до 50-х годов XX века точное строение ДНК, как и способ передачи наследственной информации, оставалось неизвестным. Хотя и было доподлинно известно, что ДНК состоит из нескольких цепочек, состоящих из нуклеотидов, никто не знал точно, сколько этих цепочек и как они соединены.
Структура двойной спирали ДНК была предложена Френсисом Криком и Джеймсом Уотсоном в 1953 году на основании рентгеноструктурных данных, полученных Морисом Уилкинсом и Розалинд Франклин, и «правил Чаргаффа», согласно которым в каждой молекуле ДНК соблюдаются строгие соотношения, связывающие между собой количество азотистых оснований разных типов[3]. Позже предложенная Уотсоном и Криком модель строения ДНК была доказана, а их работа отмечена Нобелевской премией по физиологии и медицине 1962 г. Среди лауреатов не было скончавшейся к тому времени Розалинды Франклин, так как премия не присуждается посмертно[4].
Структура молекулы
Нуклеотиды
Дезоксирибонуклеиновая кислота (ДНК) представляет собой биополимер (полианион), мономером которого является нуклеотид[5][6].
Каждый нуклеотид состоит из остатка фосфорной кислоты, присоединённого по 5'-положению к сахару дезоксирибозе, к которому также через гликозидную связь (C—N) по 1'-положению присоединено одно из четырёх азотистых оснований. Именно наличие характерного сахара и составляет одно из главных различий между ДНК и РНК, зафиксированное в названиях этих нуклеиновых кислот (в состав РНК входит сахар рибоза)[7]. Пример нуклеотида — аденозинмонофосфат — где основание, присоединённое к фосфату и рибозе, это аденин, показан на рисунке.
Исходя из структуры молекул, основания, входящие в состав нуклеотидов, разделяют на две группы: пурины (аденин [A] и гуанин [G]) образованы соединёнными пяти- и шестичленным гетероциклами; пиримидины (цитозин [C] и тимин [T]) — шестичленным гетероциклом[8].
В виде исключения, например, у бактериофага PBS1, в ДНК встречается пятый тип оснований — урацил ([U]), пиримидиновое основание, отличающееся от тимина отсутствием метильной группы на кольце, обычно заменяющее тимин в РНК[9].
Следует отметить, что тимин и урацил не так строго приурочены к ДНК и РНК соответственно, как это считалось ранее. Так, после синтеза некоторых молекул РНК значительное число урацилов в этих молекулах метилируется с помощью специальных ферментов, превращаясь в тимин. Это происходит в транспортных и рибосомальных РНК[10].
Двойная спираль

Полимер ДНК обладает довольно сложной структурой. Нуклеотиды соединены между собой ковалентно в длинные полинуклеотидные цепи. Эти цепи в подавляющем большинстве случаев (кроме некоторых вирусов, обладающих одноцепочечными ДНК-геномами) попарно объединяются при помощи водородных связей в структуру, получившую название двойной спирали[3][7]. Остов каждой из цепей состоит из чередующихся фосфатов и сахаров[11]. Фосфатные группы формируют фосфодиэфирные связи между третьим и пятым атомами углерода соседних молекул дезоксирибозы в результате взаимодействия между 3'-гидроксильной (3'—ОН) группой одной молекулы дезоксирибозы и 5'-фосфатной группой (5'—РО3) другой. Асимметричные концы цепи ДНК называются 3' (три прим) и 5' (пять прим). Полярность цепи играет важную роль при синтезе ДНК (удлинение цепи возможно только путём присоединения новых нуклеотидов к свободному 3'-концу).
Как уже было сказано выше, у подавляющего большинства живых организмов ДНК состоит не из одной, а из двух полинуклеотидных цепей. Эти две длинные цепи закручены одна вокруг другой в виде двойной спирали, стабилизированной водородными связями, образующимися между обращёнными друг к другу азотистыми основаниями входящих в неё цепей. В природе эта спираль, чаще всего, правозакрученная. Направления от 3'-конца к 5'-концу в двух цепях, из которых состоит молекула ДНК, противоположны (цепи «антипараллельны» друг другу).
Ширина двойной спирали составляет от 22 до 24 Å, или 2,2 — 2,4 нм, длина каждого нуклеотида 3,3 Å (0,33 нм)[12]. Подобно тому, как в винтовой лестнице сбоку можно увидеть ступеньки, на двойной спирали ДНК в промежутках между фосфатным остовом молекулы можно видеть рёбра оснований, кольца которых расположены в плоскости, перпендикулярной по отношению к продольной оси макромолекулы.
В двойной спирали различают малую (12 Å) и большую (22 Å) бороздки[13]. Белки, например, факторы транскрипции, которые присоединяются к определённым последовательностям в двухцепочечной ДНК, обычно взаимодействуют с краями оснований в большой бороздке, где те более доступны[14].
Образование связей между основаниями
Каждое основание на одной из цепей связывается с одним определённым основанием на второй цепи. Такое специфическое связывание называется комплементарным. Пурины комплементарны пиримидинам (то есть, способны к образованию водородных связей с ними): аденин образует связи только с тимином, а цитозин — с гуанином. В двойной спирали цепочки также связаны с помощью гидрофобных взаимодействий и стэкинга, которые не зависят от последовательности оснований ДНК[15].
Комплементарность двойной спирали означает, что информация, содержащаяся в одной цепи, содержится и в другой цепи. Обратимость и специфичность взаимодействий между комплементарными парами оснований важна для репликации ДНК и всех остальных функций ДНК в живых организмах.
Так как водородные связи нековалентны, они легко разрываются и восстанавливаются. Цепочки двойной спирали могут расходиться как замок-молния под действием ферментов (хеликазы) или при высокой температуре[16]. Разные пары оснований образуют разное количество водородных связей. АТ связаны двумя, ГЦ — тремя водородными связями, поэтому на разрыв ГЦ требуется больше энергии. Процент ГЦ пар и длина молекулы ДНК определяют количество энергии, необходимой для диссоциации цепей: длинные молекулы ДНК с большим содержанием ГЦ более тугоплавки[17].
Части молекул ДНК, которые из-за их функций должны быть легко разделяемы, например ТАТА последовательность в бактериальных промоторах, обычно содержат большое количество А и Т.
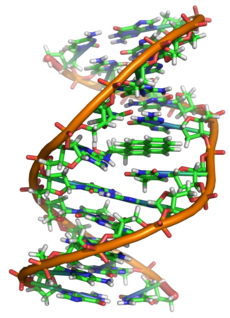
Химические модификации оснований
Структура хроматина влияет на транскрипцию генов: участки гетерохроматина (отсутствие или низкий уровень транскрипции генов) коррелируют с метилированием цитозина. Например, метилирование цитозина с образованием 5-метилцитозина важно для инактивации Х-хромосомы[19]. Средний уровень метилирования отличается у разных организмов, так, у нематоды Caenorhabditis elegans метилирование цитозина не наблюдается, а у позвоночных обнаружен высокий уровень метилирования — до 1 %[20].
Несмотря на биологическую роль, 5-метилцитозин может спонтанно утрачивать аминную группу (деаминироваться), превращаясь в тимин, поэтому метилированные цитозины являются источником повышенного числа мутаций[21]. Другие модификации оснований включают метилирование аденина у бактерий и гликозилирование урацила с образованием «J-основания» в кинетопластах[22].
Повреждение ДНК
ДНК может повреждаться разнообразными мутагенами, к которым относятся окисляющие и алкилирующие вещества, а также высокоэнергетическая электромагнитная радиация — ультрафиолетовое и рентгеновское излучение. Тип повреждения ДНК зависит от типа мутагена. Например, ультрафиолет повреждает ДНК путём образования в ней димеров тимина, которые возникают при образовании ковалентных связей между соседними основаниями[23].
Оксиданты, такие как свободные радикалы или перекись водорода, приводят к нескольким типам повреждения ДНК, включая модификации оснований, в особенности гуанозина, а также двухцепочечные разрывы в ДНК[24]. По некоторым оценкам, в каждой клетке человека окисляющими соединениями ежедневно повреждается порядка 500 оснований[25][26]. Среди разных типов повреждений наиболее опасные — это двухцепочечные разрывы, потому что они трудно репарируются и могут привести к потерям участков хромосом (делециям) и транслокациям.
Многие молекулы мутагенов вставляются (интеркалируют) между двумя соседними парами оснований. Большинство этих соединений, например, этидий, даунорубицин, доксорубицин и талидомид имеют ароматическую структуру. Для того чтобы интеркалирующее соединение могло поместиться между основаниями, они должны разойтись, расплетая и нарушая структуру двойной спирали. Эти изменения в структуре ДНК мешают транскрипции и репликации, вызывая мутации. Поэтому интеркалирующие соединения часто являются канцерогенами, наиболее известные из которых — бензопирен, акридины, афлатоксин и бромистый этидий[27][28][29]. Несмотря на эти негативные свойства, в силу их способности подавлять транскрипцию и репликацию ДНК, интеркалирующие соединения используются в химиотерапии для подавления быстро растущих клеток рака[30].
Суперскрученность
Если взяться за концы верёвки и начать скручивать их в разные стороны, она становится короче и на верёвке образуются «супервитки». Так же может быть суперскручена и ДНК. В обычном состоянии цепочка ДНК делает один оборот на каждые 10,4 основания, но в суперскрученном состоянии спираль может быть свёрнута туже или расплетена[31]. Выделяют два типа суперскручивания: положительное — в направлении нормальных витков, при котором основания расположены ближе друг к другу; и отрицательное — в противоположном направлении. В природе молекулы ДНК обычно находятся в отрицательном суперскручивании, которое вносится ферментами — топоизомеразами[32]. Эти ферменты удаляют дополнительное скручивание, возникающее в ДНК в результате транскрипции и репликации[33].

Структуры на концах хромосом
На концах линейных хромосом находятся специализированные структуры ДНК, называемые теломерами. Основная функция этих участков — поддержание целостности концов хромосом[35]. Теломеры также защищают концы ДНК от деградации экзонуклеазами и предотвращают активацию системы репарации[36]. Поскольку обычные ДНК-полимеразы не могут реплицировать 3' концы хромосом, это делает специальный фермент — теломераза.
В клетках человека теломеры обычно представлены одноцепочечной ДНК и состоят из несколько тысяч повторяющихся единиц последовательности ТТАГГГ[37]. Эти последовательности с высоким содержанием гуанина стабилизируют концы хромосом, формируя очень необычные структуры, называемые G-квадруплексами и состоящие из четырёх, а не двух взаимодействующих оснований. Четыре гуаниновых основания, все атомы которых находятся в одной плоскости, образуют пластинку, стабилизированную водородными связями между основаниями и хелатированием в центре неё иона металла (чаще всего калия). Эти пластинки располагаются стопкой друг над другом[38].
На концах хромосом могут образовываться и другие структуры: основания могут быть расположены в одной цепочке или в разных параллельных цепочках. Кроме этих «стопочных» структур теломеры формируют большие петлеобразные структуры, называемые Т-петли или теломерные петли. В них одноцепочечная ДНК располагается в виде широкого кольца, стабилизированного теломерными белками[39]. В конце Т-петли одноцепочечная теломерная ДНК присоединяется к двухцепочечной ДНК, нарушая спаривание цепочек в этой молекуле и образуя связи с одной из цепей. Это трёхцепочечное образование называется Д-петля (от англ. displacement loop)[38].
Биологические функции
ДНК является носителем генетической информации, записанной в виде последовательности нуклеотидов с помощью генетического кода. С молекулами ДНК связаны два основополагающих свойства живых организмов — наследственность и изменчивость. В ходе процесса, называемого репликацией ДНК, образуются две копии исходной цепочки, наследуемые дочерними клетками при делении, таким образом образовавшиеся клетки оказываются генетически идентичны исходной.
Генетическая информация реализуется при экспрессии генов в процессах транскрипции (синтеза молекул РНК на матрице ДНК) и трансляции (синтеза белков на матрице РНК).
Последовательность нуклеотидов «кодирует» информацию о различных типах РНК: информационных, или матричных (мРНК), рибосомальных (рРНК) и транспортных (тРНК). Все эти типы РНК синтезируются на основе ДНК в процессе транскрипции. Роль их в биосинтезе белков (процессе трансляции) различна. Информационная РНК содержит информацию о последовательности аминокислот в белке, рибосомальные РНК служат основой для рибосом (сложных нуклеопротеиновых комплексов, основная функция которых — сборка белка из отдельных аминокислот на основе иРНК), транспортные РНК доставляют аминокислоты к месту сборки белков — в активный центр рибосомы, «ползущей» по иРНК.
Структура генома

Большинство природных ДНК имеет двухцепочечную структуру, линейную (эукариоты, некоторые вирусы и отдельные роды бактерий) или кольцевую (прокариоты, хлоропласты и митохондрии). Линейную одноцепочечную ДНК содержат некоторые вирусы и бактериофаги. Молекулы ДНК находятся in vivo в плотно упакованном, конденсированном состоянии.[40] В клетках эукариот ДНК располагается главным образом в ядре в виде набора хромосом. Бактериальная (прокариоты) ДНК обычно представлена одной кольцевой молекулой ДНК, расположенной в неправильной формы образовании в цитоплазме, называемым нуклеоидом[41]. Генетическая информация генома состоит из генов. Ген — единица передачи наследственной информации и участок ДНК, который влияет на определённую характеристику организма. Ген содержит открытую рамку считывания, которая транскрибируется, а также регуляторные последовательности, например, промотор и энхансер, которые контролируют экспрессию открытых рамок считывания.
У многих видов только малая часть общей последовательности генома кодирует белки. Так, только около 1,5 % генома человека состоит из кодирующих белок экзонов, а больше 50 % ДНК человека состоит из некодирующих повторяющихся последовательностей ДНК[42]. Причины наличия такого большого количества некодирующей ДНК в эукариотических геномах и огромная разница в размерах геномов (С-значение) — одна из неразрешённых научных загадок[43]; исследования в этой области также указывают на большое количество фрагментов реликтовых вирусов в этой части ДНК.
Последовательности генома, не кодирующие белок
В настоящее время накапливается всё больше данных, противоречащих идее о некодирующих последовательностях как «мусорной ДНК» (англ. junk DNA). Теломеры и центромеры содержат малое число генов, но они важны для функционирования и стабильности хромосом[36][44]. Часто встречающаяся форма некодирующих последовательностей человека — псевдогены, копии генов, инактивированные в результате мутаций[45]. Эти последовательности нечто вроде молекулярных ископаемых, хотя иногда они могут служить исходным материалом для дупликации и последующей дивергенции генов[46]. Другой источник разнообразия белков в организме — это использование интронов в качестве «линий разреза и склеивания» в альтернативном сплайсинге[47]. Наконец, некодирующие белок последовательности могут кодировать вспомогательные клеточные РНК, например, мяРНК[48]. Недавнее исследование транскрипции генома человека показало, что 10 % генома даёт начало полиаденилированным РНК[49], а исследование и генома мыши показало, что 62 % его транскрибируется[50].
Транскрипция и трансляция
Генетическая информация, закодированная в ДНК, должна быть прочитана и в конечном итоге выражена в синтезе различных биополимеров, из которых состоят клетки. Последовательность оснований в цепочке ДНК напрямую определяет последовательность оснований в РНК, на которую она «переписывается» в процессе, называемом транскрипцией. В случае мРНК эта последовательность определяет аминокислоты белка. Соотношение между нуклеотидной последовательностью мРНК и аминокислотной последовательностью определяется правилами трансляции, которые называются генетическим кодом. Генетический код состоит из трёхбуквенных «слов», называемых кодонами, состоящих из трёх нуклеотидов (то есть ACT CAG TTT и т. п.). Во время транскрипции нуклеотиды гена копируются на синтезируемую РНК РНК-полимеразой. Эта копия в случае мРНК декодируется рибосомой, которая «читает» последовательность мРНК, осуществляя спаривание матричной РНК с транспортными РНК, которые присоединены к аминокислотам. Поскольку в трёхбуквенных комбинациях используются 4 основания, всего возможны 64 кодона (4³ комбинации). Кодоны кодируют 20 стандартных аминокислот, каждой из которых соответствует в большинстве случаев более одного кодона. Один из трёх кодонов, которые располагаются в конце мРНК, не означает аминокислоту и определяет конец белка, это «стоп» или «нонсенс» кодоны — TAA, TGA, TAG.
Репликация
Деление клеток необходимо для размножения одноклеточного и роста многоклеточного организма, но до деления клетка должна удвоить геном, чтобы дочерние клетки содержали ту же генетическую информацию, что и исходная клетка. Из нескольких теоретически возможных механизмов удвоения (репликации) ДНК реализуется полуконсервативный. Две цепочки разделяются, а затем каждая недостающая комплементарная последовательность ДНК воспроизводится ферментом ДНК-полимеразой. Этот фермент строит полинуклеотидную цепь, находя правильное основание через комплементарное спаривание оснований и присоединяя его к растущей цепочке. ДНК-полимераза не может начинать новую цепь, а только лишь наращивать уже существующую, поэтому она нуждается в короткой цепочке нуклеотидов (праймере), синтезируемой праймазой. Так как ДНК-полимеразы могут строить цепочку только в направлении 5' --> 3', для копирования антипараллельных цепей используются разные механизмы[51].
Взаимодействие с белками

Все функции ДНК зависят от её взаимодействия с белками. Взаимодействия могут быть неспецифическими, когда белок присоединяется к любой молекуле ДНК, или зависеть от наличия особой последовательности. Ферменты также могут взаимодействовать с ДНК, из них наиболее важные — это РНК-полимеразы, которые копируют последовательность оснований ДНК на РНК в транскрипции или при синтезе новой цепи ДНК — репликации.
Структурные и регуляторные белки
Хорошо изученными примерами взаимодействия белков и ДНК, не зависящего от нуклеотидной последовательности ДНК, является взаимодействие со структурными белками. В клетке ДНК связана с этими белками, образуя компактную структуру, которая называется хроматин. У прокариот хроматин образован при присоединении к ДНК небольших щелочных белков — гистонов, менее упорядоченный хроматин прокариот содержит гистон-подобные белки[52][53]. Гистоны формируют дискообразную белковую структуру — нуклеосому, вокруг каждой из которых вмещается два оборота спирали ДНК. Неспецифические связи между гистонами и ДНК образуются за счёт ионных связей щелочных аминокислот гистонов и кислотных остатков сахарофосфатного остова ДНК[54]. Химические модификации этих аминокислот включают метилирование, фосфорилирование и ацетилирование[55]. Эти химические модификации изменяют силу взаимодействия между ДНК и гистонами, влияя на доступность специфических последовательностей для факторов транскрипции и изменяя скорость транскрипции[56]. Другие белки в составе хроматина, которые присоединяются к неспецифическим последовательностям — белки с высокой подвижностью в гелях, которые ассоциируют большей частью с согнутой ДНК[57]. Эти белки важны для образования в хроматине структур более высокого порядка[58]. Особая группа белков, присоединяющихся к ДНК, — это белки, которые ассоциируют с одноцепочечной ДНК. Наиболее хорошо охарактеризованный белок этой группы у человека — репликационный белок А, без которого невозможно протекание большинства процессов, где расплетается двойная спираль, включая репликацию, рекомбинацию и репарацию. Белки этой группы стабилизируют одноцепочечную ДНК и предотвращают формирование стеблей-петель или деградации нуклеазами[59].
В то же время другие белки узнают и присоединяются к специфическим последовательностям. Наиболее изученная группа таких белков — различные классы факторов транскрипции, то есть белки, регулирующие транскрипцию. Каждый из этих белков узнаёт свою последовательность, часто в промоторе, и активирует или подавляет транскрипцию гена. Это происходит при ассоциации факторов транскрипции с РНК-полимеразой либо напрямую, либо через белки-посредники. Полимераза ассоциирует сначала с белками, а потом начинает транскрипцию[60]. В других случаях факторы транскрипции могут присоединяться к ферментам, которые модифицируют находящиеся на промоторах гистоны, что изменяет доступность ДНК для полимераз[61].
Так как специфические последовательности встречаются во многих местах генома, изменения в активности одного типа фактора транскрипции могут изменить активность тысяч генов[62]. Соответственно, эти белки часто регулируются в процессах ответа на изменения в окружающей среде, развития организма и дифференцировки клеток. Специфичность взаимодействия факторов транскрипции с ДНК обеспечивается многочисленными контактами между аминокислотами и основаниями ДНК, что позволяет им «читать» последовательность ДНК. Большинство контактов с основаниями происходит в главной бороздке, где основания более доступны[14].
Ферменты, модифицирующие ДНК
Топоизомеразы и хеликазы
В клетке ДНК находится в компактном, т. н. суперскрученном состоянии, иначе она не смогла бы в ней уместиться. Для протекания жизненно важных процессов ДНК должна быть раскручена, что производится двумя группами белков — топоизомеразами и хеликазами.
Топоизомеразы — ферменты, которые имеют и нуклеазную и лигазную активности. Эти белки изменяют степень суперскрученности в ДНК. Некоторые из этих ферментов разрезают спираль ДНК и позволяют вращаться одной из цепей, тем самым уменьшая уровень суперскрученности, после чего фермент заделывает разрыв[32]. Другие ферменты могут разрезать одну из цепей и проводить вторую цепь через разрыв, а потом лигировать разрыв в первой цепи[63]. Топоизомеразы необходимы во многих процессах, связанных с ДНК, таких как репликация и транскрипция[33].
Хеликазы — белки, которые являются одним из молекулярных моторов. Они используют химическую энергию нуклеотидтрифосфатов, чаще всего АТФ, для разрыва водородных связей между основаниями, раскручивая двойную спираль на отдельные цепочки[64]. Эти ферменты важны для большинства процессов, где белкам необходим доступ к основаниям ДНК.
Нуклеазы и лигазы
В различных процессах, происходящих в клетке, например, рекомбинации и репарации, участвуют ферменты, способные разрезать и восстанавливать целостность нитей ДНК. Ферменты, разрезающие ДНК, носят название нуклеаз. Нуклеазы, которые гидролизуют нуклеотиды на концах молекулы ДНК, называются экзонуклеазами, а эндонуклеазы разрезают ДНК внутри цепи. Наиболее часто используемые в молекулярной биологии и генетической инженерии нуклеазы — это рестриктазы, которые разрезают ДНК около специфических последовательностей. Например, фермент EcoRV (рестрикционный фермент № 5 из E. coli) узнаёт шестинуклеотидную последовательность 5'-GAT|ATC-3' и разрезает ДНК в месте, указанном вертикальной линией. В природе эти ферменты защищают бактерии от заражения бактериофагами, разрезая ДНК фага, когда она вводится в бактериальную клетку. В этом случае нуклеазы — часть системы модификации-рестрикции[65]. ДНК-лигазы сшивают сахарофосфатные основания в молекуле ДНК, используя энергию АТФ. Рестрикционные нуклеазы и лигазы используются в клонировании и фингерпринтинге.
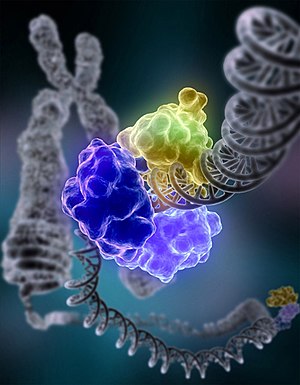
Полимеразы
Существует также важная для метаболизма ДНК группа ферментов, которые синтезируют цепи полинуклеотидов из нуклеозидтрифосфатов — ДНК-полимеразы. Они добавляют нуклеотиды к 3'-гидроксильной группе предыдущего нуклеотида в цепи ДНК, поэтому все полимеразы работают в направлении 5'--> 3'[66]. В активном центре этих ферментов субстрат — нуклеозидтрифосфат — спаривается с комплементарным основанием в составе одноцепочечной полинуклеотидной цепочки — матрицы.
В процессе репликации ДНК ДНК-зависимая ДНК-полимераза синтезирует копию исходной последовательности ДНК. Точность очень важна в этом процессе, так как ошибки в полимеризации приведут к мутациям, поэтому многие полимеразы обладают способностью к «редактированию» — исправлению ошибок. Полимераза узнаёт ошибки в синтезе по отсутствию спаривания между неправильными нуклеотидами. После определения отсутствия спаривания активируется 3'--> 5' экзонуклеазная активность полимеразы, и неправильное основание удаляется[67]. В большинстве организмов ДНК-полимеразы работают в виде большого комплекса, называемого реплисомой, которая содержит многочисленные дополнительные субъединицы, например, хеликазы[68].
РНК-зависимые ДНК-полимеразы — специализированный тип полимераз, которые копируют последовательность РНК на ДНК. К этому типу относится вирусный фермент обратная транскриптаза, который используется ретровирусами при инфекции клеток, а также теломераза, необходимая для репликации теломер[69]. Теломераза — необычный фермент, потому что она содержит собственную матричную РНК[36].
Транскрипция осуществляется ДНК-зависимой РНК-полимеразой, которая копирует последовательность ДНК одной цепочки на мРНК. В начале транскрипции гена РНК-полимераза присоединяется к последовательности в начале гена, называемой промотором, и расплетает спираль ДНК. Потом она копирует последовательность гена на матричную РНК до тех пор, пока не дойдёт до участка ДНК в конце гена — терминатора, где она останавливается и отсоединяется от ДНК. Также как ДНК-зависимая ДНК-полимераза человека, РНК-полимераза II, которая транскрибирует большую часть генов в геноме человека, работает в составе большого белкового комплекса, содержащего регуляторные и дополнительные единицы[70].
Генетическая рекомбинация
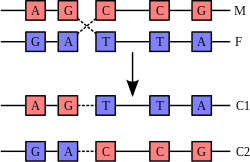
Двойная спираль ДНК обычно не взаимодействует с другими сегментами ДНК, и в человеческих клетках разные хромосомы пространственно разделены в ядре[71]. Это расстояние между разными хромосомами важно для способности ДНК действовать в качестве стабильного носителя информации. В процессе рекомбинации с помощью ферментов две спирали ДНК разрываются, обмениваются участками, после чего непрерывность спиралей восстанавливается, поэтому обмен участками негомологичных хромосом может привести к повреждению целостности генетического материала.
Рекомбинация позволяет хромосомам обмениваться генетической информацией, в результате этого образуются новые комбинации генов, что увеличивает эффективность естественного отбора и важно для быстрой эволюции новых белков[72]. Генетическая рекомбинация также играет роль в репарации, особенно в ответе клетки на разрыв обеих цепей ДНК.[73]
Самая распространённая форма кроссинговера — это гомологичная рекомбинация, когда принимающие участие в рекомбинации хромосомы имеют очень похожие последовательности. Иногда в качестве участков гомологии выступают транспозоны. Негомологичная рекомбинация может привести к повреждению клетки, поскольку в результате такой рекомбинации возникают транслокации. Реакция рекомбинации катализируется ферментами, которые называются рекомбиназы, например, Cre. На первом этапе реакции рекомбиназа делает разрыв в одной из цепей ДНК, позволяя этой цепи отделиться от комплементарной цепи и присоединиться к одной из цепей второй хроматиды. Второй разрыв в цепи второй хроматиды позволяет ей также отделиться и присоединиться к оставшейся без пары цепи из первой хроматиды, формируя структуру Холлидея. Структура Холлидея может передвигаться вдоль соединённой пары хромосом, меняя цепи местами. Реакция рекомбинации завершается, когда фермент разрезает соединение, а две цепи лигируются.[74]
Эволюция метаболизма, основанного на ДНК
ДНК содержит генетическую информацию, которая делает возможной жизнедеятельность, рост, развитие и размножение всех современных организмов. Однако как долго в течение четырёх миллиардов лет истории жизни на Земле ДНК была главным носителем генетической информации, неизвестно. Существуют гипотезы, что РНК играла центральную роль в обмене веществ, поскольку она может и переносить генетическую информацию, и осуществлять катализ с помощью рибозимов[75][76][77]. Кроме того, РНК — один из основных компонентов «фабрик белка» — рибосом. Древний РНК-мир, где нуклеиновая кислота была использована и для катализа, и для переноса информации, мог послужить источником современного генетического кода, состоящего из четырёх оснований. Это могло произойти в результате того, что число оснований в организме было компромиссом между небольшим числом оснований, увеличивавшим точность репликации, и большим числом оснований, увеличивающим каталитическую активность рибозимов[78].
К сожалению, древние генетические системы не дошли до наших дней. ДНК в окружающей среде в среднем сохраняется в течение 1 миллиона лет, а потом деградирует до коротких фрагментов. Извлечение ДНК и определение последовательности их 16S рРНК генов из бактериальных спор, заключённых в кристаллах соли 250 млн лет назад,[79] служит темой оживлённой дискуссии в научной среде.[80][81]
См. также
- Использование ДНК в технологии
- Вектор (биология)
- Генетическая генеалогия
- Действие излучений на структуру и функции ДНК
- Метилирование ДНК
- Мобильные элементы генома
- Мутация
- Нуклеопротеиды
- РНК
- Футпринтинг ДНК
- Центральная догма молекулярной биологии
- Спиртовая преципитация
- Цис-элемент
Примечания
- ↑ Dahm R (2005). "Friedrich Miescher and the discovery of DNA". Dev Biol. 278 (2): 274—88. PMID 15680349.
- ↑ Hershey A, Chase M (1952). "Independent functions of viral protein and nucleic acid in growth of bacteriophage" (PDF). J Gen Physiol. 36 (1): 39—56. PMID 12981234.
- ↑ 1 2 Watson J, Crick F (1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737—8. PMID 13054692.
- ↑ The Nobel Prize in Physiology or Medicine 1962 Nobelprize .org Accessed 22 Dec 06
- ↑ Alberts, Bruce. Molecular Biology of the Cell; Fourth Edition. — New York and London : Garland Science, 2002. — ISBN ISBN 0-8153-3218-1.
- ↑ Butler, John M. (2001) Forensic DNA Typing «Elsevier». pp. 14 — 15. ISBN 978-0-12-147951-0
- ↑ 1 2 Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
- ↑ Takahashi I, Marmur J. (1963). "Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis". Nature. 197: 794—5. PMID 13980287.
- ↑ Agris P (2004). "Decoding the genome: a modified view". Nucleic Acids Res. 32 (1): 223—38. PMID 14715921.
- ↑ Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z". Acta Crystallogr D Biol Crystallogr. 59 (Pt 4): 620—6. PMID 12657780.
- ↑ Mandelkern M, Elias J, Eden D, Crothers D (1981). "The dimensions of DNA in solution". J Mol Biol. 152 (1): 153—61. PMID 7338906.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). "Crystal structure analysis of a complete turn of B-DNA". Nature. 287 (5784): 755—8. PMID 7432492.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Pabo C, Sauer R. "Protein-DNA recognition". Annu Rev Biochem. 53: 293—321. PMID 6236744.
- ↑ Ponnuswamy P, Gromiha M (1994). "On the conformational stability of oligonucleotide duplexes and tRNA molecules". J Theor Biol. 169 (4): 419—32. PMID 7526075.
- ↑ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). "Mechanical stability of single DNA molecules". Biophys J. 78 (4): 1997—2007. PMID 10733978.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Chalikian T, Völker J, Plum G, Breslauer K (1999). "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques". Proc Natl Acad Sci U S A. 96 (14): 7853—8. PMID 10393911.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Created from PDB 1JDG
- ↑ Klose R, Bird A (2006). "Genomic DNA methylation: the mark and its mediators". Trends Biochem Sci. 31 (2): 89—97. PMID 16403636.
- ↑ Bird A (2002). "DNA methylation patterns and epigenetic memory". Genes Dev. 16 (1): 6—21. PMID 11782440.
- ↑ Walsh C, Xu G. "Cytosine methylation and DNA repair". Curr Top Microbiol Immunol. 301: 283—315. PMID 16570853.
- ↑ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei". Cell. 75 (6): 1129—36. PMID 8261512.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation". Biochemistry. 42 (30): 9221—6. PMID 12885257.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). "Hydroxyl radicals and DNA base damage". Mutat Res. 424 (1—2): 9—21. PMID 10064846.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Shigenaga M, Gimeno C, Ames B (1989). "Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage". Proc Natl Acad Sci U S A. 86 (24): 9697—701. PMID 2602371.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Cathcart R, Schwiers E, Saul R, Ames B (1984). "Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage" (PDF). Proc Natl Acad Sci U S A. 81 (18): 5633—7. PMID 6592579.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Ferguson L, Denny W (1991). "The genetic toxicology of acridines". Mutat Res. 258 (2): 123—60. PMID 1881402.
- ↑ Jeffrey A (1985). "DNA modification by chemical carcinogens". Pharmacol Ther. 28 (2): 237—72. PMID 3936066.
- ↑ Stephens T, Bunde C, Fillmore B (2000). "Mechanism of action in thalidomide teratogenesis". Biochem Pharmacol. 59 (12): 1489—99. PMID 10799645.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). "Intercalators as anticancer drugs". Curr Pharm Des. 7 (17): 1745—80. PMID 11562309.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Benham C, Mielke S (2005). "DNA mechanics". Annu Rev Biomed Eng. 7: 21—53. PMID 16004565.
- ↑ 1 2 Champoux J (2001). "DNA topoisomerases: structure, function, and mechanism". Annu Rev Biochem. 70: 369—413. PMID 11395412.
- ↑ 1 2 Wang J (2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nat Rev Mol Cell Biol. 3 (6): 430—40. PMID 12042765.
- ↑ Created from NDB UD0017
- ↑ Greider C, Blackburn E (1985). "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts". Cell. 43 (2 Pt 1): 405—13. PMID 3907856.
- ↑ 1 2 3 Nugent C, Lundblad V (1998). "The telomerase reverse transcriptase: components and regulation". Genes Dev. 12 (8): 1073—85. PMID 9553037.
- ↑ Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997). "Normal human chromosomes have long G-rich telomeric overhangs at one end". Genes Dev. 11 (21): 2801—9. PMID 9353250.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ 1 2 Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Res. 34 (19): 5402—15. PMID 17012276.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). "Mammalian telomeres end in a large duplex loop". Cell. 97 (4): 503—14. PMID 10338214.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Teif V.B. and Bohinc K. (2010). "Condensed DNA: condensing the concepts". Progress in Biophysics and Molecular Biology. doi:10.1016/j.pbiomolbio.2010.07.002.
- ↑ Thanbichler M, Wang S, Shapiro L (2005). "The bacterial nucleoid: a highly organized and dynamic structure". J Cell Biochem. 96 (3): 506—21. PMID 15988757.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Wolfsberg T, McEntyre J, Schuler G (2001). "Guide to the draft human genome". Nature. 409 (6822): 824—6. PMID 11236998.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Gregory T (2005). "The C-value enigma in plants and animals: a review of parallels and an appeal for partnership". Ann Bot (Lond). 95 (1): 133—46. PMID 15596463.
- ↑ Pidoux A, Allshire R (2005). "The role of heterochromatin in centromere function" (PDF). Philos Trans R Soc Lond B Biol Sci. 360 (1455): 569—79. PMID 15905142.
- ↑ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Res. 12 (2): 272—80. PMID 11827946.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Harrison P, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". J Mol Biol. 318 (5): 1155—74. PMID 12083509.
- ↑ Soller M (2006). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Cell Mol Life Sci. 63 (7–9): 796—819. PMID 16465448.
- ↑ Michalak P. (2006). "RNA world - the dark matter of evolutionary genomics". 19 (6): 1768—74. PMID 17040373.
{{cite journal}}: Cite journal требует|journal=(справка) - ↑ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S; et al. (2005). "RNA world - the dark matter of evolutionary genomics". 308: 1149—54. PMID 15790807.
{{cite journal}}: Cite journal требует|journal=(справка); Явное указание et al. в:|author=(справка)Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Mattick JS (2004). "RNA regulation: a new genetics?". Nat Rev Genet. 5: 316—323. PMID 15131654.
- ↑ Albà M (2001). "Replicative DNA polymerases". Genome Biol. 2 (1): REVIEWS3002. PMID 11178285.
- ↑ Sandman K, Pereira S, Reeve J (1998). "Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome". Cell Mol Life Sci. 54 (12): 1350—64. PMID 9893710.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Dame RT (2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Mol. Microbiol. 56 (4): 858—70. PMID 15853876.
- ↑ Luger K, Mäder A, Richmond R, Sargent D, Richmond T (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251—60. PMID 9305837.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Jenuwein T, Allis C (2001). "Translating the histone code". Science. 293 (5532): 1074—80. PMID 11498575.
- ↑ Ito T. "Nucleosome assembly and remodelling". Curr Top Microbiol Immunol. 274: 1—22. PMID 12596902.
- ↑ Thomas J (2001). "HMG1 and 2: architectural DNA-binding proteins". Biochem Soc Trans. 29 (Pt 4): 395—401. PMID 11497996.
- ↑ Grosschedl R, Giese K, Pagel J (1994). "HMG domain proteins: architectural elements in the assembly of nucleoprotein structures". Trends Genet. 10 (3): 94—100. PMID 8178371.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Iftode C, Daniely Y, Borowiec J (1999). "Replication protein A (RPA): the eukaryotic SSB". Crit Rev Biochem Mol Biol. 34 (3): 141—80. PMID 10473346.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Myers L, Kornberg R. "Mediator of transcriptional regulation". Annu Rev Biochem. 69: 729—49. PMID 10966474.
- ↑ Spiegelman B, Heinrich R (2004). "Biological control through regulated transcriptional coactivators". Cell. 119 (2): 157—67. PMID 15479634.
- ↑ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003). "A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells". Proc Natl Acad Sci U S A. 100 (14): 8164—9. PMID 12808131.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Schoeffler A, Berger J (2005). "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism". Biochem Soc Trans. 33 (Pt 6): 1465—70. PMID 16246147.
- ↑ Tuteja N, Tuteja R (2004). "Unraveling DNA helicases. Motif, structure, mechanism and function". Eur J Biochem. 271 (10): 1849—63. PMID 15128295.
- ↑ Bickle T, Krüger D (1993). "Biology of DNA restriction". Microbiol Rev. 57 (2): 434—50. PMID 8336674.
- ↑ Joyce C, Steitz T (1995). "Polymerase structures and function: variations on a theme?". J Bacteriol. 177 (22): 6321—9. PMID 7592405.
- ↑ Hubscher U, Maga G, Spadari S. "Eukaryotic DNA polymerases". Annu Rev Biochem. 71: 133—63. PMID 12045093.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Johnson A, O'Donnell M. "Cellular DNA replicases: components and dynamics at the replication fork". Annu Rev Biochem. 74: 283—315. PMID 15952889.
- ↑ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994). "The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention". FASEB J. 8 (8): 497—503. PMID 7514143.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Martinez E (2002). "Multi-protein complexes in eukaryotic gene transcription". Plant Mol Biol. 50 (6): 925—47. PMID 12516863.
- ↑ Cremer T, Cremer C (2001). "Chromosome territories, nuclear architecture and gene regulation in mammalian cells". Nat Rev Genet. 2 (4): 292—301. PMID 11283701.
- ↑ Pál C, Papp B, Lercher M (2006). "An integrated view of protein evolution". Nat Rev Genet. 7 (5): 337—48. PMID 16619049.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ O'Driscoll M, Jeggo P (2006). "The role of double-strand break repair - insights from human genetics". Nat Rev Genet. 7 (1): 45—54. PMID 16369571.
- ↑ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). "The RuvABC resolvasome". Eur J Biochem. 269 (22): 5492—501. PMID 12423347.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Joyce G (2002). "The antiquity of RNA-based evolution". Nature. 418 (6894): 214—21. PMID 12110897.
- ↑ Orgel L. "Prebiotic chemistry and the origin of the RNA world" (PDF). Crit Rev Biochem Mol Biol. 39 (2): 99—123. PMID 15217990.
- ↑ Davenport R (2001). "Ribozymes. Making copies in the RNA world". Science. 292 (5520): 1278. PMID 11360970.
- ↑ Szathmáry E (1992). "What is the optimum size for the genetic alphabet?" (PDF). Proc Natl Acad Sci U S A. 89 (7): 2614—8. PMID 1372984.
- ↑ Vreeland R, Rosenzweig W, Powers D (2000). "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal". Nature. 407 (6806): 897—900. PMID 11057666.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Hebsgaard M, Phillips M, Willerslev E (2005). "Geologically ancient DNA: fact or artefact?". Trends Microbiol. 13 (5): 212—20. PMID 15866038.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка) - ↑ Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002). "Curiously modern DNA for a "250 million-year-old" bacterium". J Mol Evol. 54 (1): 134—7. PMID 11734907.
{{cite journal}}: Википедия:Обслуживание CS1 (множественные имена: authors list) (ссылка)
Литература
- Альбертс Б., Брей Д., Льюис Дж. и др. Молекулярная биология клетки в 3-х томах. — М.: Мир, 1994. — 1558 с. — ISBN 5-03-001986-3.
- Докинз Р. Эгоистичный ген. — М.: Мир.
- История биологии с начала XX века до наших дней. — М.: Наука, 1975. — 660 с.
- Льюин Б. Гены. — М.: Мир, 1987. — 1064 с.
- Пташне М. Переключение генов. Регуляция генной активности и фаг лямбда. — М.: Мир, 1989. — 160 с. Все форумы > Книга «переключение генов» М. Пташне
- Уотсон Дж. Д. Двойная спираль: воспоминания об открытии структуры ДНК. — М.: Мир, 1969. — 152 с.
Ссылки
- Методы выделения и исследования ДНК
- Веб-адреса молекулярно-биологических журналов
- Международная база данных — последовательности ДНК из разных организмов (англ.).
- Веб-сайт Сэнгеровского Института одного из мировых лидеров в области определения последовательностей ДНК и их анализа (англ.)
- Software for forensic DNA Typing — eQMS::DNA
Эта статья входит в число избранных статей русскоязычного раздела Википедии. |
Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link GA Шаблон:Link GA